Chapter 9 SQL과 R
Chapter leads: Martijn Schuemie & Peter Rijnbeek
공통 데이터 모델 Common Data Model(CDM)은 모든 데이터가 필드가 있는 테이블의 레코드로 표시되는 관계형 데이터베이스 모델이다. 이는 일반적으로 PostgreSQL, Oracle, Microsoft SQL Server와 같은 소프트웨어 플랫폼을 사용하여 데이터가 관계형 데이터베이스에 저장된다는 것을 의미한다. 사용자는 ATLAS와 Methods Library 같은 다양한 OHDSI 도구를 통해 데이터베이스에 간접적으로 쿼리를 작성하여 분석을 수행하지만, 적절한 접근 권한이 있으면 이 도구를 사용하지 않고 직접 데이터베이스에 쿼리를 이용해 분석할 수도 있다. 직접 쿼리를 작성하는 주된 이유는 기존 도구가 지원하지 않는 분석을 수행하기 위한 것이다. 그러나 OHDSI 도구는 사용자가 데이터를 적절하게 분석을 할 수 있도록 (전문가가 오랜 시간 고려하여 만든 지침을 따르도록) 설계되어 있고, 직접 쿼리를 작성하다보면 실수를 범할 위험이 더 커진다. 직접 쿼리를 작성할 때는 질의하는 것은 그런 지침을 제공하지 않는다. 관계형 데이터베이스에 쿼리를 작성하는 표준 언어는 Structured Query Language(SQL)이며, 이는 데이터에 대한 변경뿐만 아니라 데이터 추출용 쿼리를 위해 사용할 수 있다. SQL의 기본 명령어는 실제로 표준이고 소프트웨어 플랫폼 전반에 걸쳐 같은 의미가 있지만, 각각의 SQL 플랫폼마다 미묘한 변경되는 고유한 문법을 가지고 있다. 예를 들면, SQL Server에서 PERSON 테이블에서 상위 10개의 행을 검색하려면 다음과 같은 쿼리를 입력한다:
SELECT TOP 10 * FROM person;PostgreSQL의 동일한 쿼리는 다음과 같다:
SELECT * FROM person LIMIT 10;OHDSI에서는 플랫폼이 사용하는 고유한 문법에 구애받지 않고 모든 OHDSI 데이터베이스에서 동일한 SQL 언어를 사용하고자 한다. 이러한 이유로 OHDSI는 이 장의 뒤에서 논의하게 될, 하나의 표준 문법을 다른 여러 개의 문법으로 번역해줄 수 있는 패키지인 SqlRender를 개발하였다. 이 표준 언어 - OHDSI SQL - 는 주로 SQL Server SQL 언어의 하위 집합이다. 이 장에서 제공되는 SQL 문에는 모두 OHDSI SQL을 사용한다.
각 데이터베이스 플랫폼에는 SQL을 사용하여 데이터베이스를 쿼리하기 위한 자체 소프트웨어 도구가 제공된다. OHDSI는 여러 데이터베이스 플랫폼에 연결할 수 있는 하나의 R 패키지인 DatabaseConnector를 개발하였다. DatabaseConnector도 이 장의 뒤에서 논의할 것이다.
따라서 OHDSI 도구를 사용하지 않고도 CDM에 맞게 쿼리를 작성할 수 있지만 DatabaseConnector 및 SqlRender 패키지를 사용하는 것을 권장한다. 이를 통해 한 기관에서 개발한 쿼리를 수정하지 않고도 다른 기관에서 사용할 수 있다. 즉, SQL server를 사용하는 기관이 작성한 쿼리를 PostgreSQL을 사용하는 기관에서도 적용할 수 있다. R 자체는 통계 분석 및 대화식 그래프 생성과 같이 데이터베이스에서 추출된 데이터를 추가로 분석하는 기능도 직접 제공한다.
이 장에서는 독자가 SQL에 대한 기본 지식을 가지고 있다고 가정한다. 먼저 SqlRender 및 DatabaseConnector 사용 방법을 안내한다. 독자가 이 패키지를 사용할 생각이 없다면 이 절은 건너뛰어도 된다. 9.3절에서는 SQL(OHDSI SQL)을 사용하여 CDM에 쿼리하는 방법에 관해 설명한다. 그 다음 절에서는 CDM 쿼리 작성 시 OHDSI 표준 용어를 사용하는 방법을 강조한다. 공개적으로 이용 가능한 CDM에 대해 일반적으로 사용되는 쿼리 모음인 QueryLibrary를 특히 자세히 살펴본다. 발생률을 추정하는 예제 연구로 이 장을 마무리하고 SqlRender 및 DatabaseConnector를 사용하여 이 연구를 구현한다.
9.1 SqlRender
SqlRender 패키지는 Comprehensive R Archive Network(CRAN)에서 이용할 수 있으므로 다음을 사용하여 설치할 수 있다:
install.packages("SqlRender")SqlRender는 전통적인 데이터베이스 시스템 (PostgreSQL, Microsoft SQL Server, SQLite, and Oracle), 병렬 데이터 웨어하우스 (Microsoft APS, IBM Netezza, and Amazon RedShift), 빅데이터 플랫폼 (Hadoop through Impala, and Google BigQuery)을 포함한 다수의 기술 플랫폼을 지원한다.
9.1.1 SQL의 매개 변수화
패키지의 기능 중 하나는 SQL문에 매개 변수를 지원하는 것이다. 때때로 일부 매개 변수에 기반하여 SQL문을 조금씩 변형할 필요가 있다. 일반적인 SQL 문법에서 매개변수 이용은 매우 복잡하며, 매개변수화 지원은 SqlRender 의 매우 강력한 기능이다. SqlRender는 매개 변수를 허용하기 위해 SQL 코드 내에서 간단한 마크업 구문을 제공한다. 매개 변수를 기반으로 SQL을 렌더링하는 것은 render() 함수를 사용하여 수행한다.
매개 변수값 대체하기
@ 문자는 렌더링 시 실제 매개 변수값과 교환해야 하는 매개 변수 이름을 나타내는 데 사용할 수 있다. 다음 예에서 a라고 불리는 변수가 SQL에서 언급되어 있다. 렌더 함수를 호출할 때 이 매개 변수의 값이 정의된다:
sql <- "SELECT * FROM concept WHERE concept_id = @a;"
render(sql, a = 123)## [1] "SELECT * FROM concept WHERE concept_id = 123;"대부분의 SQL 문법과 달리 테이블 또는 필드 이름을 값으로 매개 변수화하기 매우 쉽다!
sql <- "SELECT * FROM @x WHERE person_id = @a;"
render(sql, x = "observation", a = 123)## [1] "SELECT * FROM observation WHERE person_id = 123;"매개 변수값은 숫자, 문자열, 부울 및 쉼표를 기준으로 항목을 나눈 리스트로 변환된 벡터일 수도 있다.
sql <- "SELECT * FROM concept WHERE concept_id IN (@a);"
render(sql, a = c(123, 234, 345))## [1] "SELECT * FROM concept WHERE concept_id IN (123,234,345);"If-Then-Else
때로는 하나 이상의 매개 변수값에 따라 코드 블록을 켜거나 끌 필요가 있다. 이 작업은 {Condition} ? {if true} : {if false} 구문을 사용한다. 조건이 참 또는 1로 평가되면, if true 블록이 사용되고 그렇지 않으면, if false 블록이 표시된다 (있는 경우). (역자 주: 매개변수화 기능은 전반적으로 정말 놀라운 기능이다!)
sql <- "SELECT * FROM cohort {@x} ? {WHERE subject_id = 1}"
render(sql, x = FALSE)## [1] "SELECT * FROM cohort "render(sql, x = TRUE)## [1] "SELECT * FROM cohort WHERE subject_id = 1"간단한 비교도 지원된다:
sql <- "SELECT * FROM cohort {@x == 1} ? {WHERE subject_id = 1};"
render(sql, x = 1)## [1] "SELECT * FROM cohort WHERE subject_id = 1;"render(sql, x = 2)## [1] "SELECT * FROM cohort ;"IN 연산자도 지원된다:
sql <- "SELECT * FROM cohort {@x IN (1,2,3)} ? {WHERE subject_id = 1};"
render(sql, x = 2)## [1] "SELECT * FROM cohort WHERE subject_id = 1;"9.1.2 다른 SQL 언어로의 변환
SqlRender 패키지의 또 다른 기능은 OHDSI SQL에서 다른 SQL 언어로 변환하는 것이다. 예를 들면 다음과 같다:
sql <- "SELECT TOP 10 * FROM person;"
translate(sql, targetDialect = "postgresql")## [1] "SELECT * FROM person LIMIT 10;"targetDialect 매개 변수는 다음과 같은 값을 가질 수 있다: “oracle”, “postgresql”, “pdw”, “redshift”, “impala”, “netezza”, “bigquery”, “sqlite”, “sql server”.
최선의 노력에도 불구하고, 지원되는 모든 플랫폼에서 오류 없이 실행될 OHDSI SQL을 작성할 때 고려해야 할 사항이 몇 가지 있다. 다음은 이러한 고려 사항에 대해 자세히 설명한다.
변환에 의해 지원되는 기능 및 구조
다음과 같은 SQL Server 함수는 테스트 되었으며 다양한 언어로 올바르게 변환되는 것으로 확인되었다:
| Function | Function | Function |
|---|---|---|
| ABS | EXP | RAND |
| ACOS | FLOOR | RANK |
| ASIN | GETDATE | RIGHT |
| ATAN | HASHBYTES* | ROUND |
| AVG | ISNULL | ROW_NUMBER |
| CAST | ISNUMERIC | RTRIM |
| CEILING | LEFT | SIN |
| CHARINDEX | LEN | SQRT |
| CONCAT | LOG | SQUARE |
| COS | LOG10 | STDEV |
| COUNT | LOWER | SUM |
| COUNT_BIG | LTRIM | TAN |
| DATEADD | MAX | UPPER |
| DATEDIFF | MIN | VAR |
| DATEFROMPARTS | MONTH | YEAR |
| DATETIMEFROMPARTS | NEWID | |
| DAY | PI | |
| EOMONTH | POWER |
* Oracle은 특별 권한이 필요하다. SQLite에는 해당하는 것이 없다.
마찬가지로 많은 SQL 구문 구조가 지원된다. 다음은 우리가 잘 번역할 수 있는 표현식의 전체 목록이다:
-- Simple selects:
SELECT * FROM table;
-- Selects with joins:
SELECT * FROM table_1 INNER JOIN table_2 ON a = b;
-- Nested queries:
SELECT * FROM (SELECT * FROM table_1) tmp WHERE a = b;
-- Limiting to top rows:
SELECT TOP 10 * FROM table;
-- Selecting into a new table:
SELECT * INTO new_table FROM table;
-- Creating tables:
CREATE TABLE table (field INT);
-- Inserting verbatim values:
INSERT INTO other_table (field_1) VALUES (1);
-- Inserting from SELECT:
INSERT INTO other_table (field_1) SELECT value FROM table;
-- Simple drop commands:
DROP TABLE table;
-- Drop table if it exists:
IF OBJECT_ID('ACHILLES_analysis', 'U') IS NOT NULL
DROP TABLE ACHILLES_analysis;
-- Drop temp table if it exists:
IF OBJECT_ID('tempdb..#cohorts', 'U') IS NOT NULL
DROP TABLE #cohorts;
-- Common table expressions:
WITH cte AS (SELECT * FROM table) SELECT * FROM cte;
-- OVER clauses:
SELECT ROW_NUMBER() OVER (PARTITION BY a ORDER BY b)
AS "Row Number" FROM table;
-- CASE WHEN clauses:
SELECT CASE WHEN a=1 THEN a ELSE 0 END AS value FROM table;
-- UNIONs:
SELECT * FROM a UNION SELECT * FROM b;
-- INTERSECTIONs:
SELECT * FROM a INTERSECT SELECT * FROM b;
-- EXCEPT:
SELECT * FROM a EXCEPT SELECT * FROM b;문자열 연결
문자열 연결은 SQL Server가 다른 언어보다 덜 구체적인 영역이다. SQL Server에서는 SELECT first_name + ' ' + last_name AS full_name FROM table과 같이 작성하지만 Postgres와 Oracle에서는 SELECT first_name || ' ' || last_name AS full_name FROM table이라고 작성한다. SqlRender는 연결되는 값이 문자열인지 추측하려고 한다. 위의 예에서 명시적인 문자열 (작은따옴표로 묶인 공백) 이 있으므로 번역은 정확할 것이다. 그러나 SELECT first_name + last_name AS full_name FROM table과 같이 작성한다면 SqlRender는 두 필드가 문자열이라는 단서가 없으며, 잘못된 더하기 기호를 남겼다. 값이 문자열이라는 또 다른 단서는 “VARCHAR”에 대한 명시적 형 변환이므로 SELECT last_name + CAST(age AS VARCHAR(3)) AS full_name FROM table도 올바르게 변환된다. 모호성을 피하려면 CONCAT() 함수를 사용하여 두 개 이상의 문자열을 연결하는 것이 가장 좋다.
테이블 별칭과 AS 키워드
많은 SQL 언어는 테이블 별칭을 정의할 때 AS 키워드를 사용할 수 있지만, 키워드 없이도 잘 동작 한다. 예를 들어, 이 두 SQL 문은 SQL Server, PostgreSQL, RedShift 등에 적합하다:
-- Using AS keyword
SELECT *
FROM my_table AS table_1
INNER JOIN (
SELECT * FROM other_table
) AS table_2
ON table_1.person_id = table_2.person_id;
-- Not using AS keyword
SELECT *
FROM my_table table_1
INNER JOIN (
SELECT * FROM other_table
) table_2
ON table_1.person_id = table_2.person_id;그러나 Oracle에서는 AS 키워드를 사용하면 오류가 발생한다. 위의 예제 중 첫 번째 쿼리는 실패한다. 따라서 테이블 별칭을 지정할 때 AS 키워드를 사용하지 않는 것이 좋다. (참고로 Oracle에서 AS를 사용할 수 없는 테이블 별칭과 이 AS를 사용해야 하는 필드 별칭을 쉽게 구별할 수 없기 때문에 SqlRender가 이것을 처리하도록 만들 수 없다)
임시 테이블
임시 테이블은 중간 결과를 저장하는 데 매우 유용할 수 있으며 올바르게 사용하면 쿼리 성능을 크게 향상할 수 있다. 대부분의 데이터베이스 플랫폼에서 임시 테이블이라는 매우 좋은 기능을 가지고 있다. 현재 사용자에게만 보이며 세션이 끝나면 자동으로 삭제되고 사용자에게 쓰기 권한이 없어도 생성할 수 있다. 불행히도, Oracle에서는 임시테이블은 기본적으로 영구적인 테이블이며, 데이터의 내부는 현재 사용자에게만 보인다는 차이점만 있다. 이것이 Oracle에서 SqlRender가 다음과 같이 임시 테이블을 에뮬레이션하려고 시도하는 이유이다.
- 테이블 이름에 임의의 문자열을 추가하여 다른 사용자의 테이블이 충돌하지 않도록 한다.
- 사용자가 임시 테이블이 작성될 스키마를 지정할 수 있도록 허용한다.
예를 들면:
sql <- "SELECT * FROM #children;"
translate(sql, targetDialect = "oracle", oracleTempSchema = "temp_schema")## [1] "SELECT * FROM temp_schema.fiu76ja0children ;"사용자는 temp_schema에 대한 쓰기 권한이 있어야 한다.
또한 Oracle은 테이블 이름이 30자로 제한되어 있다. 세션 아이디를 추가한 후 이름이 너무 길어지기 때문에 임시 테이블 이름은 최대 22자까지만 허용된다.
그뿐만 아니라 Oracle의 임시 테이블은 자동 삭제되지 않으므로 Oracle에 임시 테이블 스키마가 쌓이는 것을 방지하기 위해 모든 임시 테이블을 사용한 후에는 명시적으로 TRUNCATE 및 DROP을 해야 한다.
암묵적인 형 변환
SQL Server가 다른 언어보다 덜 명시적인 몇 가지 점 중 하나는 암묵적인 형 변환을 허용한다는 것이다. 예를 들어 이 코드는 SQL Server에서 작동한다:
CREATE TABLE #temp (txt VARCHAR);
INSERT INTO #temp
SELECT '1';
SELECT * FROM #temp WHERE txt = 1;비록 txt는 VARCHAR 필드이고 이것을 정수와 비교하고 있다. 즉, 이는 논리적으로 비교할 수 없는 (문자열과 정수의 비교) 비교이지만, SQL Server는 비교를 허용하기 위해 두 가지 중 하나를 자동으로 올바른 타입으로 변환한다 (보통 정수를 문자열로 ‘암묵적으로’ 변환한다). 이와 대조적으로, PostgreSQL과 같은 다른 언어는 VARCHAR과 INT를 비교하려고 할 때 오류를 일으킬 것이다.
따라서 형 변환은 항상 명시적으로 해야 한다. 위의 마지막에 있는 예는
SELECT * FROM #temp WHERE txt = CAST(1 AS VARCHAR);또는 아래와 같이 대체되어야 한다.
SELECT * FROM #temp WHERE CAST(txt AS INT) = 1;문자열 비교의 대소문자 구분
SQL Server와 같은 일부 DBMS 플랫폼은 대소문자를 구분하지 않는 비교를 수행하는 반면, PostgreSQL과 같은 다른 플랫폼은 대소문자를 구분한다. 따라서 항상 대소문자를 구분하는 비교를 가정하고 명확하게 모르는 경우 명시적으로 대소문자를 구분하지 않도록 하는 명령을 추가하여 비교하기 추천한다. 예를 들어,
SELECT * FROM concept WHERE concep_class_id = 'Clinical Finding'대신, 다음과 같이 사용하는 것이 좋다.
SELECT * FROM concept WHERE LOWER(concep_class_id) = 'clinical finding'스키마와 데이터베이스
SQL Server에서 테이블은 스키마 안에 있으며 스키마는 데이터베이스 안에 있다. 예를 들면, cdm_data.dbo.person은 cdm_data 데이터베이스의 dbo 스키마 안에 있는 person 테이블을 말한다. 다른 언어에서는 비슷한 계층 구조가 종종 존재하더라도 매우 다르게 사용된다. SQL Server에는 일반적으로 데이터베이스 당 하나의 스키마 (dbo라고 함), 가 있으며 사용자는 다른 데이터베이스의 데이터를 쉽게 사용할 수 있다. Postgres와 같은 다른 플랫폼에서는 단일 세션에서 데이터베이스 간 데이터를 사용할 수 없지만, 데이터베이스 안에는 많은 스키마를 가지고 있다. SQL Server의 데이터베이스는 PostgreSQL에서 스키마라고 할 수 있다.
따라서 SQL Server의 데이터베이스와 스키마를 단일 매개변수로 연결할 것을 권장한다. 이 매개 변수는 일반적으로 @databaseSchema 라고 한다. 예를 들면 우리는 매개 변수화된 SQL을 가질 수 있다.
SELECT * FROM @databaseSchema.personSQL Server에서 databaseSchema = "cdm_data.dbo"값에 데이터베이스와 스키마 이름을 모두 포함할 수 있다. 다른 플랫폼에서는 같은 코드를 사용할 수 있지만, 스키마 매개 변수값은 다음과 같이 지정한다: databaseSchema = "cdm_data"
이것이 실패하는 한 가지 상황은 에러를 발생시키는 USE cdm_data.dbo;, 즉 USE 명령어를 사용했기 때문이다. 따라서 USE 명령어를 사용하지 말고 항상 테이블이 있는 데이터베이스 및 스키마를 지정하는 것이 바람직하다.
매개 변수화된 SQL 디버깅하기
매개 변수화된 SQL을 디버깅하는 것은 약간 복잡할 수 있다. 렌더링 된 SQL만 데이터베이스 서버에 대해 테스트할 수 있지만 매개 변수화된 (사전 렌더링 된) SQL에서 코드를 변경해야 한다.
SqlRender 패키지에는 대화형으로 SQL 소스를 편집하여 SQL을 렌더링하거나 반대로 번역할 수 있는 Shiny 앱이 포함되어 있다. 이 앱은 다음과 같이 시작한다:
launchSqlRenderDeveloper()그러면 그림 9.1에 표시된 앱으로 기본 브라우저가 열린다. 이 앱은 웹에서도 공개적으로 사용할 수 있다.49
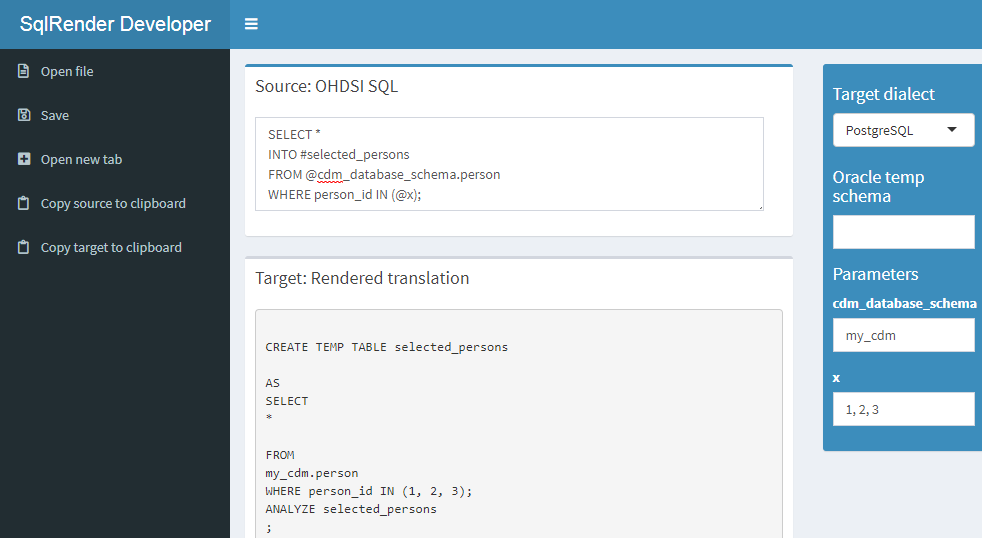
Figure 9.1: The SqlDeveloper Shiny 앱.
앱에서 OHDSI SQL을 입력하고 대상 언어를 선택하고 SQL에 매개 변수값을 제공하면 자동으로 번역된 SQL이 하단에 나타난다.
9.2 DatabaseConnector
DatabaseConnector 는 Java의 JDBC 드라이버를 사용하여 다양한 데이터베이스 플랫폼에 연결하기 위한 R 패키지이다. DatabaseConnector 패키지는 CRAN (종합 R 아카이브 네트워크)에서 사용할 수 있으므로 다음을 사용하여 설치할 수 있다:
install.packages("DatabaseConnector")DatabaseConnector는 기존 데이터베이스 시스템 (PostgreSQL, Microsoft SQL Server, SQLite 및 Oracle), 병렬 데이터웨어 하우스 (Microsoft APS, IBM Netezza 및 Amazon RedShift) 및 빅데이터 플랫폼 (Hadoop through Impla 및 Google BigQuery) 을 포함한 다양한 기술 플랫폼을 지원한다. 패키지에는 이미 대부분의 드라이버가 포함되어 있지만, 라이센스 문제로 인해 BigQuery, Netezza 및 Impla 용 드라이버는 포함되어 있지 않아서 사용자가 구해야 한다. 이러한 드라이버를 다운로드하는 방법에 대한 지침을 보려면 ?jdbcDrivers 를 입력한다. 다운로드한 후 connect, dbConnect, and createConnectionDetails 함수의 pathToDriver 인수로 사용할 수 있다.
9.2.1 연결 생성하기
데이터베이스에 연결하려면 데이터베이스 플랫폼, 서버의 위치, 사용자 이름 및 비밀번호와 같은 많은 세부 사항을 지정해야 한다. connect 함수를 호출하여 다음 세부 사항을 직접 지정할 수 있다:
conn <- connect(dbms = "postgresql",
server = "localhost/postgres",
user = "joe",
password = "secret",
schema = "cdm")## Connecting using PostgreSQL driver각 플랫폼에 필요한 세부 사항에 대한 정보는 ?connect 를 참조하라. 나중에 작업을 마치고 연결 끊는 것을 잊지 말라:
disconnect(conn)서버 이름을 제공하는 대신 JDBC connecting string을 사용하는 것이 더 편리할 경우 이를 제공할 수도 있다는 점에 유의하라:
connString <- "jdbc:postgresql://localhost:5432/postgres"
conn <- connect(dbms = "postgresql",
connectionString = connString,
user = "joe",
password = "secret",
schema = "cdm")## Connecting using PostgreSQL driver때때로 먼저 세부 사항을 지정하고 나중에 연결할 때까지 연결을 연기해야 할 수 있다. 예를 들어, 함수 내에서 연결이 설정되고 세부 사항은 인수로 전달해야 하는 경우에 편리할 수 있다. 이를 목적으로 createConnectionDetails 함수를 사용할 수 있다:
details <- createConnectionDetails(dbms = "postgresql",
server = "localhost/postgres",
user = "joe",
password = "secret",
schema = "cdm")
conn <- connect(details)## Connecting using PostgreSQL driver9.2.2 쿼리하기
데이터베이스 쿼리를 위한 주요 함수는 querySql과 executeSql 이다. 이러한 함수의 차이점은 querySql은 데이터베이스가 데이터를 반환할 것으로 예상하며, 한 번에 하나의 SQL 문만 처리할 수 있다는 것이다. 이와 대조적으로 executeSql은 데이터를 반환할 것을 예상하지 않으며, 단일 SQL 문자열에서 복수의 SQL 문을 수용한다.
몇 가지 예시:
querySql(conn, "SELECT TOP 3 * FROM person")## person_id gender_concept_id year_of_birth
## 1 1 8507 1975
## 2 2 8507 1976
## 3 3 8507 1977executeSql(conn, "TRUNCATE TABLE foo; DROP TABLE foo;")두 함수 모두 광범위한 오류 보고 기능을 제공한다: 서버에서 오류가 발생하면 오류 메시지와 문제가 되는 SQL 부분이 텍스트 파일에 기록되어 더 나은 디버깅을 돕는다. 기본적으로 executeSql 함수도 실행된 SQL 문의 백분율을 나타내는 진행 표시줄을 보여준다. 이러한 속성이 필요하지 않은 경우 패키지는 lowLevelQuerySql과 lowLevelExecuteSql 함수도 제공한다.
9.2.3 Ffdf 객체를 사용하여 쿼리하기
데이터베이스에서 가져올 데이터가 너무 커서 종종 메모리에 들어갈 수 없는 경우도 있다. 8.4.2절에서 언급했듯이, 그러한 경우 ff 패키지를 사용하여 R 데이터 객체를 디스크에 저장하고 메모리에서 사용하듯이 사용할 수 있다. DatabaseConnector는 객체에 데이터를 직접 다운로드할 수 있다:
x <- querySql.ffdf(conn, "SELECT * FROM person")x는 이제 ffdf 객체이다.
9.2.4 같은 SQL을 사용하여 다른 플랫폼 쿼리하기
SqlRender 패키지의 render 및 translate 함수를 먼저 호출하는 다음과 같은 편의 함수를 사용할 수 있다: renderTranslateExecuteSql, renderTranslateQuerySql, renderTranslateQuerySql.ffdf. 예를 들면:
x <- renderTranslateQuerySql(conn,
sql = "SELECT TOP 10 * FROM @schema.person",
schema = "cdm_synpuf")SQL Server 관련 ‘TOP 10’ 구문은 PostgreSQL에서 예를 들어 ‘LIMIT 10’으로 변환되며 SQL 매개변수 @schema는 제공된 값 ‘cdm_synpuf’로 인스턴스화 되는 것에 주의해야 한다.
9.2.5 테이블 삽입하기
executeSql 함수를 사용하여 SQL 문을 전송하여 데이터베이스에 데이터를 삽입할 수도 있지만, insertTable 함수를 사용하는 것이 더 편리하고 빠르다 (일부 최적화로 인해):
data(mtcars)
insertTable(conn, "mtcars", mtcars, createTable = TRUE)이 예는 mtcars 데이터 프레임을 자동으로 서버의 ’mtcars’라는 테이블로 업로드하고 생성한다.
9.3 CDM 쿼리 실행
다음 예시에서는 CDM이 적용된 데이터베이스를 쿼리 실행하기 위해 OHDSI SQL을 사용한다. 이러한 쿼리는 CDM의 데이터를 찾을 수 있는 데이터베이스 스키마를 나타내기 위해 @cdm을 사용한다.
데이터베이스에 얼마나 많은 사람이 있는지 쿼리 실행하는 것부터 시작할 수 있다:
SELECT COUNT(*) AS person_count FROM @cdm.person;| PERSON_COUNT |
|---|
| 26299001 |
그렇지 않으면 observation period의 평균에 관심이 있을 수도 있다:
SELECT AVG(DATEDIFF(DAY,
observation_period_start_date,
observation_period_end_date) / 365.25) AS num_years
FROM @cdm.observation_period;| NUM_YEARS |
|---|
| 1.980803 |
테이블을 조인하여 추가 통계를 생성할 수 있다. 조인은 일반적으로 테이블의 특정 필드가 동일한 값을 갖도록 하여 여러 테이블의 필드를 결합한다. 예를 들어 두 테이블 모두 가지고 있는 PERSON_ID 필드로 PERSON 테이블과 OBSERVATION_PEROPD 테이블을 조인할 수 있다. 즉, 조인의 결과는 두 테이블의 모든 필드를 갖는 새로운 테이블과 같은 집합이지만, 모든 행에서 두 테이블의 PERSON_ID는 동일한 값을 가져야 한다. 예를 들어 PERSON 테이블의 YEAR_OF_BIRTH 필드와 함께 OBSERVATION_PERIOD 테이블의 OBSERVATION_PERIOD_END_DATE 필드를 사용하여 관찰 종료 시 환자의 최고 나이를 계산할 수 있다:
SELECT MAX(YEAR(observation_period_end_date) -
year_of_birth) AS max_age
FROM @cdm.person
INNER JOIN @cdm.observation_period
ON person.person_id = observation_period.person_id;| MAX_AGE |
|---|
| 90 |
관찰 시작 당시 연령 분포를 결정하려면 훨씬 더 복잡한 쿼리가 필요하다. 이 쿼리에서는 먼저 PERSON 테이블과 OBSERVATION_PERIOD을 조인하여 관찰 당시 연령을 계산한다. 또한 연령을 기준으로 이 조인된 집합의 순서를 정렬하고 order_nr로 저장한다. 이 조인의 결과를 여러 번 사용하고 싶기 때문에 “ages”라고 하는 common table expression(CTE) (WITH ... AS를 사용하여 정의된) 으로 정의한다. 즉, 연령을 기존 테이블인 것처럼 나타낼 수 있다. “ages”의 행 수를 세어 “n”을 생성하고 각 사분위 수에 대해 order_nr이 분수 시간 “n” 보다 작은 최소 연령을 찾는다. 예를 들어, 중앙값을 찾기 위해 \(order\_nr < .50 * n\)인 최소 연령을 사용한다. 최소 및 최대 연령은 별도로 계산된다:
WITH ages
AS (
SELECT age,
ROW_NUMBER() OVER (
ORDER BY age
) order_nr
FROM (
SELECT YEAR(observation_period_start_date) - year_of_birth AS age
FROM @cdm.person
INNER JOIN @cdm.observation_period
ON person.person_id = observation_period.person_id
) age_computed
)
SELECT MIN(age) AS min_age,
MIN(CASE
WHEN order_nr < .25 * n
THEN 9999
ELSE age
END) AS q25_age,
MIN(CASE
WHEN order_nr < .50 * n
THEN 9999
ELSE age
END) AS median_age,
MIN(CASE
WHEN order_nr < .75 * n
THEN 9999
ELSE age
END) AS q75_age,
MAX(age) AS max_age
FROM ages
CROSS JOIN (
SELECT COUNT(*) AS n
FROM ages
) population_size;| MIN_AGE | Q25_AGE | MEDIAN_AGE | Q75_AGE | MAX_AGE |
|---|---|---|---|---|
| 0 | 6 | 17 | 34 | 90 |
SQL을 사용하는 대신 R에서 더 복잡한 계산을 수행할 수도 있다. 예를 들어, 이 코드를 사용하여 동일한 결과를 얻을 수 있다:
sql <- "SELECT YEAR(observation_period_start_date) -
year_of_birth AS age
FROM @cdm.person
INNER JOIN @cdm.observation_period
ON person.person_id = observation_period.person_id;"
age <- renderTranslateQuerySql(conn, sql, cdm = "cdm")
quantile(age[, 1], c(0, 0.25, 0.5, 0.75, 1))## 0% 25% 50% 75% 100%
## 0 6 17 34 90서버에서 연령을 계산하고 모든 연령을 다운로드한 다음 연령 분포를 계산한다. 그러나 이를 위해서는 데이터베이스 서버에서 수백만 행의 데이터를 다운로드해야 하므로 효율성이 떨어진다. 계산이 SQL에서 가장 잘 수행되는지 R에서 가장 잘 수행되는지를 사례별로 결정해야 한다.
쿼리는 CDM의 source value를 사용할 수도 있다. 예를 들어, 다음을 사용하여 가장 빈번한 상위 10개의 condition source code를 검색할 수 있다:
SELECT TOP 10 condition_source_value,
COUNT(*) AS code_count
FROM @cdm.condition_occurrence
GROUP BY condition_source_value
ORDER BY -COUNT(*);| CONDITION_SOURCE_VALUE | CODE_COUNT |
|---|---|
| 4019 | 49094668 |
| 25000 | 36149139 |
| 78099 | 28908399 |
| 319 | 25798284 |
| 31401 | 22547122 |
| 317 | 22453999 |
| 311 | 19626574 |
| 496 | 19570098 |
| I10 | 19453451 |
| 3180 | 18973883 |
여기서 CONDITION_OCCURRENCE 테이블의 행을 CONDITION_SOURCE_VALUE 필드의 값으로 그룹화하고 각 그룹의 행 수를 세었다. 우리는 CONDITION_SOURCE_VALUE, count, 그리고 count의 역순을 검색했다.
9.4 쿼리 작성 시 OMOP 용어 사용하기
많은 작업에서 용어 vocabulary는 유용하다. Vocabulary 테이블은 CDM의 일부이므로 SQL 쿼리를 사용하여 이용할 수 있다. Vocabulary에 대한 쿼리가 CDM에 대한 쿼리와 어떻게 결합할 수 있는지 보여준다. CDM의 많은 필드에는 CONCEPT 테이블을 사용하여 확인할 수 있는 concept ID가 포함되어 있다. 예를 들어, 데이터베이스에서 성별에 따라 계층화된 인원수를 세려고 할 때, GENDER_CONCEPT_ID를 개념 이름으로 찾아 바꾸어 사용하는 것이 더 편리할 것이다:
SELECT COUNT(*) AS subject_count,
concept_name
FROM @cdm.person
INNER JOIN @cdm.concept
ON person.gender_concept_id = concept.concept_id
GROUP BY concept_name;| SUBJECT_COUNT | CONCEPT_NAME |
|---|---|
| 14927548 | FEMALE |
| 11371453 | MALE |
용어의 매우 강력한 특징은 계층구조에 있다. 특정 개념과 그에 속하는 모든 하위 개념을 찾는 쿼리를 사용하는 경우가 빈번하다. 예를 들어, ibuprofen 성분이 들어 있는 모든 약의 처방 개수를 세고 싶다고 상상해보라:
SELECT COUNT(*) AS prescription_count
FROM @cdm.drug_exposure
INNER JOIN @cdm.concept_ancestor
ON drug_concept_id = descendant_concept_id
INNER JOIN @cdm.concept ingredient
ON ancestor_concept_id = ingredient.concept_id
WHERE LOWER(ingredient.concept_name) = 'ibuprofen'
AND ingredient.concept_class_id = 'Ingredient'
AND ingredient.standard_concept = 'S';| PRESCRIPTION_COUNT |
|---|
| 26871214 |
9.5 QueryLibrary
QueryLibrary는 CDM에 대해 일반적으로 사용되는 SQL 쿼리의 라이브러리이다. 그림 9.2에 표시된 응용 프로그램50 및 R 패키지로 제공된다.51
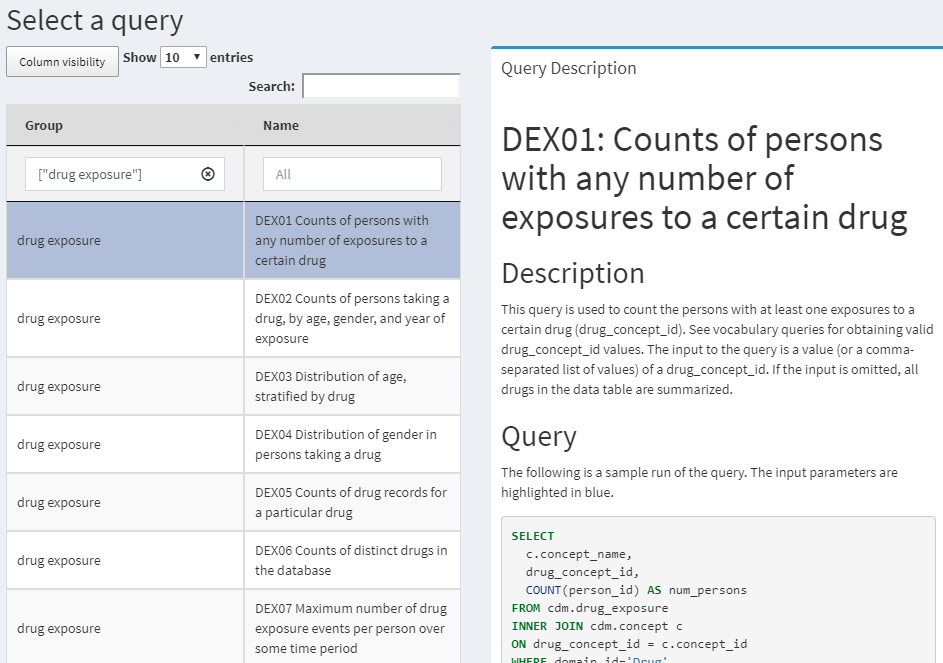
Figure 9.2: QueryLibrary: CDM에 대한 SQL 조회 라이브러리.
라이브러리의 목적은 새로운 사용자가 CDM에 쿼리하는 방법을 배우도록 돕는 것이다. 라이브러리의 쿼리는 OHDSI 커뮤니티에서 검토하고 승인하였다. 쿼리 라이브러리는 주로 교육 목적으로 사용되지만 숙련된 사용자에게 유용한 자원이기도 하다.
QueryLibrary는 SqlRender를 사용하여 선택한 SQL 언어로 쿼리를 출력한다. 사용자는 CDM 데이터베이스 스키마, vocabulary 데이터베이스 스키마 (별도의 경우) 및 Oracle 임시 스키마 (필요한 경우)를 지정할 수 있으므로 이러한 설정으로 쿼리가 자동으로 렌더링 된다.
9.6 간단한 연구 구성하기
9.6.1 문제 정의
혈관 부종Angioedema은 ACE inhibitor(ACEi)의 잘 알려진 부작용이다. (Slater et al. 1988) ACEi 치료 첫 주에 혈관 부종의 발생률이 주당 3,000명의 환자당 1건인 것으로 추정하였다. 여기서 우리는 이 결론을 모방하고 나이와 성별에 따라 계층화한다. 간단하게 하기 위해서 우리는 하나의 ACEi: lisinopril에 중점을 둔다. 따라서 우리는 질문에 대답한다.
Lisinopril 치료 개시 후 첫 주에 나이와 성별에 따라 계층화되는 혈관 부종의 비율은 얼마인가?
9.6.2 노출
노출 Exposure은 lisinopril에 대한 첫 번째 노출로 정의한다. 먼저 이전에 lisinopril에 노출되지 않았음을 의미한다. 첫 노출 전에 365일의 연속 관찰 기간이 필요하다.
9.6.3 결과
입원 또는 응급실 방문 중 혈관 부종 진단 코드의 발생으로 혈관 부종을 정의한다.
9.6.4 위험 노출 기간 Time-at-risk
환자가 일주일 동안 노출되었는지와 관계없이 이 치료 시작 후 첫 주에 발생률을 계산한다.
9.7 SQL과 R을 사용하여 연구 구현
OHDSI 툴 규약에 구속되지 않지만 동일한 원칙을 따르는 것은 도움이 된다. 이 경우 OHDSI 툴의 작동 방식과 유사하게 SQL을 사용하여 코호트 테이블을 채운다. 코호트 테이블은 CDM에 정의되어 있으며 사전 정의된 필드 집합도 있다. 먼저 쓰기 접근 권한이 있는 데이터베이스 스키마에 COHORT 테이블을 만들어야 하는데, 이는 CDM 데이터를 저장하는 데이터베이스 스키마와 동일하지 않을 수 있다.
library(DatabaseConnector)
conn <- connect(dbms = "postgresql",
server = "localhost/postgres",
user = "joe",
password = "secret")
cdmDbSchema <- "cdm"
cohortDbSchema <- "scratch"
cohortTable <- "my_cohorts"
sql <- "
CREATE TABLE @cohort_db_schema.@cohort_table (
cohort_definition_id INT,
cohort_start_date DATE,
cohort_end_date DATE,
subject_id BIGINT
);
"
renderTranslateExecuteSql(conn, sql,
cohort_db_schema = cohortDbSchema,
cohort_table = cohortTable)여기서는 데이터베이스 스키마 및 테이블 이름을 매개 변수화하여 다른 환경에 쉽게 적용할 수 있다. 결과는 데이터베이스 서버의 빈 테이블이다.
9.7.1 노출 코호트
다음으로 노출 코호트를 만들어 COHORT 테이블에 삽입한다:
sql <- "
INSERT INTO @cohort_db_schema.@cohort_table (
cohort_definition_id,
cohort_start_date,
cohort_end_date,
subject_id
)
SELECT 1 AS cohort_definition_id,
cohort_start_date,
cohort_end_date,
subject_id
FROM (
SELECT drug_era_start_date AS cohort_start_date,
drug_era_end_date AS cohort_end_date,
person_id AS subject_id
FROM (
SELECT drug_era_start_date,
drug_era_end_date,
person_id,
ROW_NUMBER() OVER (
PARTITION BY person_id
ORDER BY drug_era_start_date
) order_nr
FROM @cdm_db_schema.drug_era
WHERE drug_concept_id = 1308216 -- Lisinopril
) ordered_exposures
WHERE order_nr = 1
) first_era
INNER JOIN @cdm_db_schema.observation_period
ON subject_id = person_id
AND observation_period_start_date < cohort_start_date
AND observation_period_end_date > cohort_start_date
WHERE DATEDIFF(DAY,
observation_period_start_date,
cohort_start_date) >= 365;
"
renderTranslateExecuteSql(conn, sql,
cohort_db_schema = cohortDbSchema,
cohort_table = cohortTable,
cdm_db_schema = cdmDbSchema)여기에서는 DRUG_EXPOSURE 테이블에서 자동으로 파생되는 CDM의 표준 테이블인 DRUG ERA 테이블을 사용한다. DRUG ERA 테이블에는 성분 수준에서 연속 노출 기간이 기록되어 있다. 따라서 lisinopril을 검색할 수 있으며, 이는 lisinopril을 함유한 모든 약물에 대한 노출을 자동으로 식별할 수 있다. 사람당 첫 번째 약물 노출을 취한 다음 OBSERVATION_PERIOD 테이블과 조인한다. 청구 데이터의 경우 한 사람이 여러 개의 관찰 기간을 가질 수 있으므로 약물 노출이 포함된 기간과 겹치는 OBSERVATION PERIOD를 조인해야 한다. 그런 다음 OBSERVATION_PERIOD_START_DATE와 COHORT_START_DATE 사이에 적어도 365일이 필요함을 명시한다.
9.7.2 결과 코호트
마지막으로, 우리는 결과outcome 코호트를 만들어야 한다:
sql <- "
INSERT INTO @cohort_db_schema.@cohort_table (
cohort_definition_id,
cohort_start_date,
cohort_end_date,
subject_id
)
SELECT 2 AS cohort_definition_id,
cohort_start_date,
cohort_end_date,
subject_id
FROM (
SELECT DISTINCT person_id AS subject_id,
condition_start_date AS cohort_start_date,
condition_end_date AS cohort_end_date
FROM @cdm_db_schema.condition_occurrence
INNER JOIN @cdm_db_schema.concept_ancestor
ON condition_concept_id = descendant_concept_id
WHERE ancestor_concept_id = 432791 -- Angioedema
) distinct_occurrence
INNER JOIN @cdm_db_schema.visit_occurrence
ON subject_id = person_id
AND visit_start_date <= cohort_start_date
AND visit_end_date >= cohort_start_date
WHERE visit_concept_id IN (262, 9203,
9201) -- Inpatient or ER;
"
renderTranslateExecuteSql(conn, sql,
cohort_db_schema = cohortDbSchema,
cohort_table = cohortTable,
cdm_db_schema = cdmDbSchema)CONDITION OCCURRECE 테이블과 CONCEPT ANCESTOR 테이블을 조인하여 모든 혈관 부종과 그 자손을 찾는다. 같은 날에 여러 혈관 부종의 진단이 여러 혈관 부종 발생이 아닌 동일한 사건일 가능성이 높기 때문에 DISTINCT를 사용하여 하루에 하나의 행만 선택하도록 한다. 이러한 발생을 VISIT_OCCURRENCE 테이블과 조인하여 입원이나 응급실 환경에서 진단되었는지 확인한다.
9.7.3 발생률 계산
코호트가 준비되었으므로 연령과 성별에 따라 계층화되는 발생률을 계산할 수 있다:
sql <- "
WITH tar AS (
SELECT concept_name AS gender,
FLOOR((YEAR(cohort_start_date) -
year_of_birth) / 10) AS age,
subject_id,
cohort_start_date,
CASE WHEN DATEADD(DAY, 7, cohort_start_date) >
observation_period_end_date
THEN observation_period_end_date
ELSE DATEADD(DAY, 7, cohort_start_date)
END AS cohort_end_date
FROM @cohort_db_schema.@cohort_table
INNER JOIN @cdm_db_schema.observation_period
ON subject_id = observation_period.person_id
AND observation_period_start_date < cohort_start_date
AND observation_period_end_date > cohort_start_date
INNER JOIN @cdm_db_schema.person
ON subject_id = person.person_id
INNER JOIN @cdm_db_schema.concept
ON gender_concept_id = concept_id
WHERE cohort_definition_id = 1 -- Exposure
)
SELECT days.gender,
days.age,
days,
CASE WHEN events IS NULL THEN 0 ELSE events END AS events
FROM (
SELECT gender,
age,
SUM(DATEDIFF(DAY, cohort_start_date,
cohort_end_date)) AS days
FROM tar
GROUP BY gender,
age
) days
LEFT JOIN (
SELECT gender,
age,
COUNT(*) AS events
FROM tar
INNER JOIN @cohort_db_schema.@cohort_table angioedema
ON tar.subject_id = angioedema.subject_id
AND tar.cohort_start_date <= angioedema.cohort_start_date
AND tar.cohort_end_date >= angioedema.cohort_start_date
WHERE cohort_definition_id = 2 -- Outcome
GROUP BY gender,
age
) events
ON days.gender = events.gender
AND days.age = events.age;
"
results <- renderTranslateQuerySql(conn, sql,
cohort_db_schema = cohortDbSchema,
cohort_table = cohortTable,
cdm_db_schema = cdmDbSchema,
snakeCaseToCamelCase = TRUE)우선 적절한 위험 노출 기간으로 모든 노출을 포함하는 CTE인 “tar”를 만든다. OBSERVATION_PERIOD_END_DATE에서 위험 관찰 기간을 단축한다는 점에 유의한다. 또한 10년 단위로 나이를 계산하고 성별을 파악한다. CTE를 사용하면 동일한 중간 결과 집합을 쿼리에서 여러 번 사용할 수 있다는 장점이 있다. 이 경우 위험 관찰 기간 동안 발생하는 혈관 부종 사건의 수와 총 위험 관찰 기간의 양을 계산하는 데 사용된다.
SQL에서는 필드 이름에 snake_case(대소문자를 구분하지 않는)를 사용하는 반면 R에서는 camelCase(대소문자를 구분하는)를 사용하는 경향이 있기 때문 snakeCaseToCamelCase = TRUE로 한다. results 데이터 프레임 열 이름은 이제 camelCase이다.
ggplot2 패키지의 도움을 받아 다음과 같은 결과를 쉽게 표시할 수 있다:
# Compute incidence rate (IR) :
results$ir <- 1000 * results$events / results$days / 7
# Fix age scale:
results$age <- results$age * 10
library(ggplot2)
ggplot(results, aes(x = age, y = ir, group = gender, color = gender)) +
geom_line() +
xlab("Age") +
ylab("Incidence (per 1,000 patient weeks)")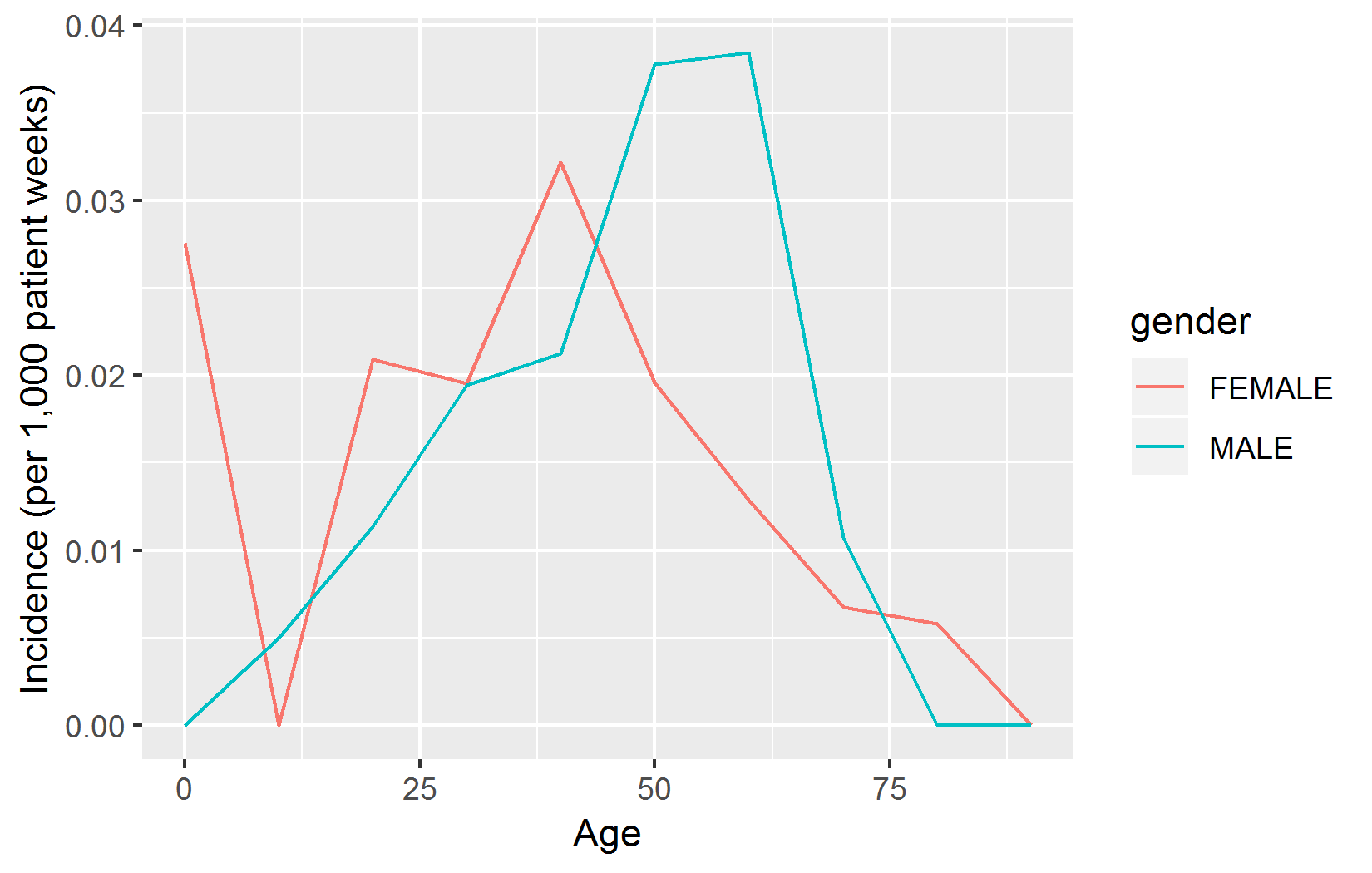
9.7.4 마무리하기
생성한 테이블을 정리하고 연결을 닫는 것을 잊지 말자.
sql <- "
TRUNCATE TABLE @cohort_db_schema.@cohort_table;
DROP TABLE @cohort_db_schema.@cohort_table;
"
renderTranslateExecuteSql(conn, sql,
cohort_db_schema = cohortDbSchema,
cohort_table = cohortTable)
disconnect(conn)9.7.5 호환성
OHDSI SQL을 DatabaseConnector 및 SQLRender와 함께 사용하기 때문에 여기서 검토한 코드는 OHDSI가 지원하는 모든 데이터베이스 플랫폼에서 실행된다.
여기에서는 예시 목적으로 수작업으로 만든 SQL 쿼리를 사용하여 코호트를 만들었다는 점에 유의하라. ATLAS에서 코호트 정의를 구성하고 ATALS에서 생성된 SQL을 사용하여 코호트를 생성하는 것이 훨씬 더 편리했을 것이다. ATLAS는 OHDSI SQL을 생성하기 때문에, SqlRender 및 DatabaseConnector와 함께 쉽게 사용할 수 있다.
9.8 요약
SQL은 공통 데이터 모델을 따르는 데이터베이스를 포함하여 데이터베이스를 조회하기 위한 표준 언어이다.
데이터베이스 플랫폼마다 SQL 언어가 다르며 각 플랫폼별 쿼리를 작성하기 위해서는 다른 많은 작업이 필요하다.
SqlRender 및 DatabaseConnector R 패키지는 CDM에서 데이터를 쿼리하는 통합된 방법을 제공하므로 동일한 분석 코드를 수정 없이 다른 환경에서 실행할 수 있다.
R과 SQL을 함께 사용하면 OHDSI 툴에서 지원하지 않는 사용자 맞춤 분석 연구를 구현할 수 있다.
- QueryLibrary 는 CDM용으로 재사용 가능한 SQL 쿼리 모음을 제공한다.
9.9 예제
전제조건
이 연습문제에서는 8.4.5절에서 설명된 대로 R, R-Studio, Java가 설치되었다고 가정한다. 또한 다음을 사용하여 설치할 수 있는 SqlRender, DatabaseConnector 및 Eunomia 패키지도 필요하다:
install.packages(c("SqlRender", "DatabaseConnector", "devtools"))
devtools::install_github("ohdsi/Eunomia", ref = "v1.0.0")Eunomia 패키지는 로컬 R 세션 내에서 실행될 CDM의 시뮬레이션 된 다른 데이터 세트를 제공한다. 연결 세부 사항은 다음을 사용하여 얻을 수 있다:
connectionDetails <- Eunomia::getEunomiaConnectionDetails()CDM 데이터베이스 스키마는 “main”이다.
Exercise 9.1 SQL과 R을 사용하여 데이터베이스에 몇 사람이 있는지 계산하십시오.
Exercise 9.2 SQL과 R을 사용하여 celecoxib을 적어도 한 번 이상 처방 한 사람을 계산하십시오.
Exercise 9.3 SQL과 R을 사용하여 celecoxib에 노출되는 동안 얼마나 많은 위장 출혈gastrointestinal hemorrhage이 있는지 진단한다. (힌트: 위장 출혈의 개념 ID는 192671이다.)
제안된 답변은 부록 E.5에서 찾을 수 있다.
References
Slater, Eve E., Debora D. Merrill, Harry A. Guess, Peter J. Roylance, Warren D. Cooper, William H. W. Inman, and Pamela W. Ewan. 1988. “Clinical Profile of Angioedema Associated With Angiotensin Converting-Enzyme Inhibition.” JAMA 260 (7): 967–70. doi:10.1001/jama.1988.03410070095035.