Chapter 10 코호트 만들기
Chapter lead: Kristin Kostka
실세계 데이터 Real world data라고도 불리는 관찰 건강 정보 Observational health data는 다양한 출처에서 꾸준하게 수집되는 환자의 건강 상태나 환자에게 제공되는 의료서비스에 관한 정보이다. CDM 데이터 유지를 위해 노력하는 OHDSI 공동 연구자는 전자 의무 기록(EHR), 보험 청구 자료, 결제 자료, 제품 및 질병 정보 등을 포함하는 다양한 출처의 데이터를 활용하며, 환자 개인이 가정 내에서, 혹은 핸드폰 등의 다른 기기를 통해 생성한 건강 정보를 활용하기도 한다. 이러한 데이터는 연구 목적으로 수집된 데이터가 아니기 때문에 우리가 보고자 하는 임상 정보를 명료하게 담고 있지 못할 수도 있다.
예를 들어, 건강 보험 청구 자료 데이터베이스는 특정 질병 (예를 들어 혈관성 부종)이 있는 환자에게 제공된 의료 서비스를 파악하고 그 치료비용을 의료기관에 상환해주기 위해 설립되었기 때문에, 청구목적에 맞는 정보만 부분적으로 담고 있다. 우리가 그런 데이터를 연구 목적으로 사용하기를 원한다면, 우리가 데이터를 사용할 때 실제로 관심 있는 것을 추론해야 하며, 타당한 추론을 가능케 하는 적절한 코호트를 설정해서 연구를 진행해야 한다. 그러므로 만약 우리가 보험 청구 자료 데이터베이스에서 새로 발생한 혈관성 부종만을 확인하고 싶다면, 코호트를 만들 때 이미 치료하고 있는 혈관성 부종 환자를 제외하기 위해서 응급실로 방문한 혈관성 부종 환자만을 코호트에 포함한다는 논리를 세워야 할 것이다. 전자 의무 기록에 담긴 임상 정보를 사용할 경우에도 비슷하다. 데이터를 이차적인 목적으로 사용하는 것이기 때문에 데이터베이스가 설립된 일차적인 목적을 인식하고 있어야 한다. 연구를 설계할 때마다 다양한 데이터베이스 환경에서 우리가 설정한 코호트가 어떻게 존재하고 있는지에 대한 뉘앙스를 항상 생각해야 한다.
이 장에서는 코호트를 생성하고 공유하는 것이 가지는 의미와 코호트를 개발하는 방법, 그리고 ATLAS와 SQL을 이용해 당신만의 코호트를 생성하는 방법에 관해서 설명할 것이다.
10.1 코호트란 무엇인가?
OHDSI 연구에서는 특정 기간 안에 하나 이상의 포함 기준에 속하는 사람의 집단을 코호트라고 정의한다. 때로 표현형 phenotype이라는 용어로 코호트를 대신 사용하기도 한다. 코호트는 OHDSI 분석 도구를 사용하거나 연구를 시작하기 위한 첫 단계로 사용된다. 예를 들어 고혈압 치료약물인 ACE inhibitor를 복용하기 시작한 사람 중에서 혈관성 부종이 일어날 위험을 예측하기 위한 연구를 진행할 때, 우리는 다음 두 가지 코호트를 지정해야 한다: 결과 코호트 outcome cohort(혈관성 부종이 발생한 사람), 그리고 대상 코호트 target cohort(ACE inhibitor를 복용하기 시작한 사람). OHDSI에서 사용되는 코호트라는 개념이 가지는 중요한 특성은, 연구 내에서 지정된 각각의 코호트는 연구 내의 다른 코호트와 상호 독립적이기 때문에 (그 연구만이 아닌 다른 연구에서도) 재사용이 가능하다는 것이다. 앞서 제시된 혈관성 부종 코호트를 예로 들어 보면, 이 코호트는 관찰되는 인구 내의 모든 혈관성 부종 발생 예를 담게 되며, 이는 혈관성 부종 코호트에는 ACE inhibitor를 사용한 사람과 사용하지 않은 사람 모두가 포함된다는 것을 의미한다. 우리의 분석 도구는 두 코호트의 교집합을 분석할 것이다. 이것이 가지는 장점은, ACE inhibitor를 복용함으로써 발생하는 다른 결과를 분석할 때에도 이번에 만든 동일한 혈관성 부종 코호트를 재사용할 수 있다는 것이다.
코호트는 특정 시간 내에 하나 이상의 포함 기준을 만족시키는 사람의 집합이다.
OHDSI에서 사용되는 코호트의 정의가 다른 분야에서 사용되는 코호트의 정의와 다를 수 있다는 것을 인지하는 것이 중요하다. 예를 들어 동료 심사 peer review를 거친 많은 과학 논문에서, 코호트는 특정 임상 코드 집합 (예를 들어 ICD-9/ICD-10, NDC, HCPCS 등)과 동일한 의미로 사용되었다. 코드 집합은 코호트를 설정하는 데 중요한 부분을 담당하지만, 코호트는 코드 집합에 의해서만 정의되는 것은 아니다. 코호트는 기준에 맞도록 코드 집합을 사용하는 특정 논리가 필요하다. (예를 들어 그 환자에게 첫 번째로 발생된 ICD-9/ICD-10 코드인가?). 잘 정의된 코호트는 환자가 어떻게 코호트에 포함되고 제외되는지를 구체적으로 설명한다.
OHDSI가 코호트를 정의하는 방식에는 다음과 같은 독특한 특징이 있다:
- 한 사람은 여러 개의 코호트에 속할 수도 있다.
- 한 사람이 동일한 코호트 여러 다른 기간에 걸쳐 속할 수도 있다.
- 한 사람이 같은 기간에 동일한 코호트에 여러 번 속하지 않을 수도 있다
- 코호트에는 0명 혹은 그 이상의 구성원을 가질 수도 있다.
코호트를 만드는 방법에는 두 가지 주요 방법이 있다:
- 규칙 기반 코호트 정의는 언제 환자가 코호트 내에 속하는지에 관한 명확한 포함 규칙을 가진다. 이 포함 규칙을 정하는 것은 코호트를 디자인하는 사람의 전문적인 지식에 상당히 의존한다.
- 확률적 코호트 정의는 확률 모델을 사용하여 환자가 코호트에 속할 확률 (0~100%)을 계산한다. 이 확률은 역치 값을 사용하여 ‘예-아니오’ 분류로 전환할 수도 있고, 그대로 사용할 수도 있다. 확률 모델은 일반적으로 예측 가능한 관련 환자 특성을 자동으로 식별하기 위해 일부 기계학습모델 (예를 들어 로지스틱 회귀)의 학습을 통해 만들어진다.
다음으로 이 두 가지의 방법에 대해서 구체적으로 알아보겠다.
10.2 규칙 기반 코호트 정의
규칙 기반 코호트 정의는 특정 기간 내에 (예를 들어 “지난 6개월 이내 해당 질병이 발생한 사람”) 하나 혹은 그 이상의 포함 기준 (예를 들어 “혈관 부종을 앓는 환자”)을 명확히 제시함으로써 시작한다.
이러한 기준을 만드는 데 사용되는 표준 구성 요소는 다음과 같다:
도메인: CDM 도메인 (예를 들어 “Procedure Occurrence”, “Drug Exposure”)은 데이터가 저장되는 곳인데, 도메인의 종류에 따라 어떤 유형의 임상 정보가 담길지, 어떤 개념이 담길지가 결정된다. 도메인에 관한 세부사항은 4.2.4절에서 확인할 수 있다.
개념 모음 Concept set: 우리가 관심을 가지는 임상적 개념을 대변하는 하나 이상의 표준화된 개념의 모음을 의미한다. concept set은 표준 용어 (임상에서 쓰이는 용어는 국가나 병원, 사람에 따라 동일한 개념도 조금씩 다른 용어로 사용되는데 이를 표준 용어로 매핑함)로 구성되어 있기 때문에 다양한 관찰 의료 데이터에서 상호 운용이 가능하다. concept set에 관하여 10.3절에 자세한 설명이 있다.
도메인별 속성: 관심 있는 임상 실체와 연관된 추가적인 속성 (예를 들어 DRUG_EXPOSURE의 DAYS_SUPPLY, MEASUREMENT의 VALUE_AS_NUMBER와 RANGE_HIGH)
시간의 설정: 선정 기준과 임상사건 발생 간의 시간 간격 (예를 들어 노출 시작 또는 노출 시작 후 365일 이내에 특정 조건이 발생해야 함)
코호트 정의를 작성할 때, 코호트 속성을 나타내는 도메인을 빌딩 블록 (그림 10.1 참조)과 유사하게 생각하면 도움이 될 수 있다. 각 도메인에서 허용 가능한 구성 요소에 대해 혼란스럽다면 언제든지 공통 데이터 모델 4장을 참조한다.

Figure 10.1: 코호트 정의를 위한 빌딩 블록
코호트 정의를 작성할 때, 다음과 같은 질문에 답할 수 있어야 한다:
- 코호트 진입 시간을 정의하는 초기 이벤트는 무엇인가?
- 초기 이벤트에는 어떤 포함 기준이 적용되는가?
- 코호트 종료 시간을 정의하는 것은 무엇인가?
코호트 진입 이벤트 cohort entry event: 코호트 진입 이벤트 (초기 이벤트)는 환자가 코호트에 진입하는 코호트 기준 시점 cohort index date으로 정의된다. 코호트 진입 이벤트는 약물 노출 Drug exposure, 질병 상태 conditions, 절차 procedures, 측정 measurements 및 방문 visits과 같은 CDM에 기록된 모든 사건일 수 있다. 초기 이벤트는 데이터가 저장되는 CDM 도메인 (예를 들어 PROCEDURS_OCCURRENCE, DRUG_EXPOSURE 등), 임상 활동을 식별하기 위해 구축된 concept set (예를 들어 질병 상태에 대한 SNOMED 코드, 약물에 대한 RxNorm 코드) 및 기타 특정 속성 (예를 들어 발생 연령, 첫 진단 / 절차 등, 지정된 시작 및 종료 날짜, 방문 유형 등)에 의해 정의된다. 진입 이벤트를 가진 사람의 집합을 초기 사건 코호트 initial event cohort라고 한다.
포함 기준 inclusion criteria: 포함 기준은 초기 이벤트 코호트에 적용되어 코호트에 진입할 사람을 추가로 제한한다. 각 포함 기준을 만들 때는 데이터가 저장되는 CDM 도메인, concept set, 도메인별 속성 (예를 들어 days supply, 방문 유형) 및 코호트 색인 날짜에 관한 시간 논리를 결정해야 한다. 적격 코호트 qualifying cohort는 초기 이벤트 코호트에서 모든 포함 기준을 충족하는 사람의 집합으로 정의한다.
코호트 종료 기준 cohort exit criteria: 코호트 종료 이벤트는 한 사람이 더 이상 코호트 자격 요건을 갖추지 못했을 때를 의미한다. 코호트 종료는 관찰 기간이 끝났을 때, 초기 진입 이벤트로부터 일정한 시간이 지났을 때 혹은 마지막 이벤트가 발생했을 때 등 여러 방법으로 정의할 수 있다. 코호트 종료 기준에 따라 한 사람의 오랜 시간에 걸친 기록 중에서 특정한 기간이 선정기준에 맞아 코호트에 한 번 포함된 후에 또 다른 기간이 코호트 선정 기간에 맞아 다시 코호트에 포함되는 등 한 사람의 관찰이 하나의 코호트에 여러 번 속할 수 있다.
OHDSI 도구에는 포함 기준과 제외 기준이 구분되지 않는다. 모든 기준은 포함 기준으로 설정해야 한다. 예를 들어 ‘이전 고혈압 환자 제외’라는 제외 기준을 ‘이전 고혈압 발생이 0인 사람 포함’이라는 포함 기준으로 설정해야 한다.
10.3 개념 모음
개념 모음 concept set을 구성하는 개념은 다양한 다른 분석에서 재사용이 가능하다. concept set은 관찰 연구에서 종종 사용되는 표준화된 컴퓨터 코드라고 생각해도 된다. concept set은 다음 특성을 포함하고 있다:
- Exclude: concept set으로부터 해당 개념과 해당 개념의 하위 개념을 제외한다.
- Descendants: 이 개념뿐만 아니라 모든 하위 항목 개념을 고려한다.
- Mapped: 표준화되지 않은 개념도 검색한다.
예를 들어 표 10.1과 같이 concept set은 두 개의 개념을 포함할 수 있다. 여기서 우리는 4329847 (“심근경색 Myocardial infarction”)과 그 모든 하위 개념을 포함했고, 314666 (“과거 심근경색 Old myocardial infarction”)과 그 모든 하위 개념은 제외했다.
| Concept Id | Concept Name | Excluded | Descendants | Mapped |
|---|---|---|---|---|
| 4329847 | Myocardial infarction | NO | YES | NO |
| 314666 | Old myocardial infarction | YES | YES | NO |
그림 10.2에서 볼 수 있다시피, “심근경색”과 그 모든 하위 개념을 포함할 것이고, 하위 개념 중에서 “과거 심근경색”과 그 모든 하위 개념은 제외할 것이다. 결과적으로 거의 100개 정도의 표준 개념 standard concept을 포함한 concept set이 만들어졌다. 이 standard concept은 다양한 데이터베이스에서 사용되는 수백 개의 원천 코드 (예를 들어 ICD-9, ICD-10)를 반영한다.
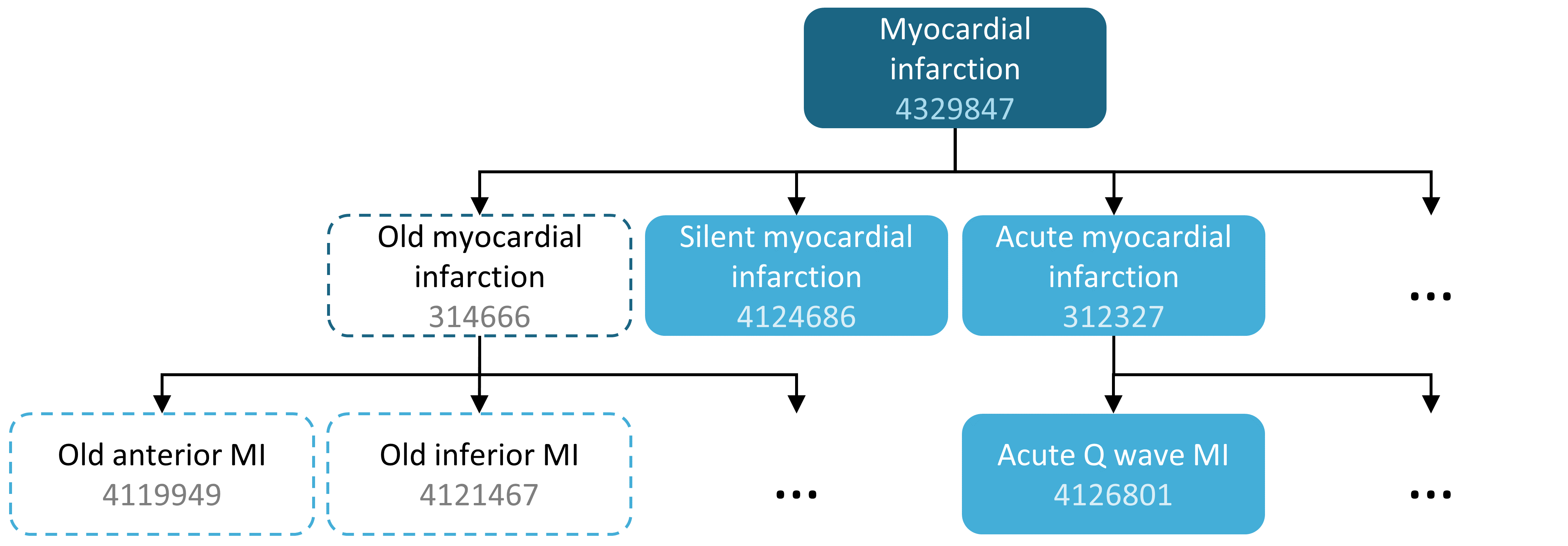
Figure 10.2: “심근경색”과 그 하위 개념을 포함하지만 “과거 심근경색”과 그 하위 개념은 제외하는 concept set
10.4 확률적 코호트 정의
규칙 기반 코호트 정의는 코호트 정의를 수행할 때 널리 사용되는 방법이다. 그러나 코호트를 만들기 위해 전문가끼리 합의를 이루는 것은 매우 많은 시간이 소요되는 일이다. 확률적 코호트 정의는 코호트 속성의 효율적인 선택을 위한 대안적인 기계 구동 방식이다. 이 접근법에서, 레이블이 붙은 증례를 이용하여 지도 기계학습이 코호트 설계 알고리즘을 학습한다. 이 알고리즘은 전반적인 연구의 정확도의 균형 (민감도/특이도 간의 균형 trade-off)를 향상시킬 수 있도록 추가 수정을 거칠 수 있다.
이 접근 방법을 CDM의 데이터에 적용한 예는 아프로디테 (APHRODITE: Automated PHenotype Routine for Observational Definition, Identification, Training and Evaluation) R 패키지이다. 이 패키지는 불완전하게 레이블이 붙은 데이터로부터 학습하는 능력을 결합한 코호트 구축 프레임워크를 제공한다. (Banda et al. 2017)
10.5 코호트 정의 유효성
코호트를 구축할 때, 다음 중 더 중요한 것이 무엇인지 고려하는 것이 필요하다: 코호트 조건에 해당하는 환자를 모두 찾는 것이 더 중요한가? 아니면 확신이 가는 환자만 찾는 것이 더 중요한가?
코호트를 구축할 때 당신의 전략은 전문가가 질병을 얼마나 엄격하게 정의하는지에 의존할 것이다. 얻을 수 있는 모든 것을 사용하거나, 최소 공통분모를 사용하거나 이 둘을 절충하는 코호트 정의를 작성할 수 있다. 관심 코호트를 적절하게 연구하기 위해 얼마나 엄격한 임계값을 사용할지는 궁극적으로 연구자의 재량에 달려 있다.
이 장의 시작 부분에서 언급했듯이 코호트 정의는 데이터로부터 무엇인가 관찰하고자 하는 것을 유추하려는 시도이다. 그러면 그러한 시도에서 코호트를 얼마나 잘 정의했는지 의문을 품게 된다. 일반적으로, 규칙 기반의 코호트 정의나 확률적 알고리즘의 검증은 작성한 코호트를 ‘절대 표준 gold standard’ 참고 값 (즉 수작업으로 차트를 검토한 것)과 비교함으로써 검증할 수 있다. 이에 대해서는 16장 (“임상적 타당성”)에서 자세히 설명한다.
10.5.1 OHDSI 절대 표준 표현형 라이브러리
커뮤니티를 지원하기 위해서 OHDSI 절대 표준 표현형 라이브러리 OHDSI Gold Standard Phenotype Library(GSPL) 그룹이 형성되었다. GSPL 그룹의 목표는 규칙 기반 및 확률적 방법으로 커뮤니티 기반의 코호트 라이브러리를 개발하는 것이다. GSPL은 OHDSI 커뮤니티의 멤버가 각자의 연구를 위해 커뮤니티가 검증한 코호트를 찾아서 실행시킬 수 있게 하였다. 이 ‘절대 표준 gold standard’ 코호트는 라이브러리 안에 들어 있다. GSPL과 관련된 추가적인 정보를 얻으려면 OHDSI 워크그룹 페이지에 문의한다. 이전에 소개되었던 APHRODITE (Banda et al. 2017) 와 PheValuator tool (Swerdel, Hripcsak, and Ryan 2019) 뿐만 아니라 OHDSI 네트워크에서 EHR과 유전 정보를 공유하기 위해 만들어진 Electronic Medical Records and Genomics (eMERGE) Phenotype Library (Hripcsak et al. 2019) 도 해당 작업 그룹에서 다루고 있다. 당신이 코호트를 설계하는 데 관심이 많다면, 이 워크 그룹에 참여한다.
10.6 고혈압 환자 코호트 작성하기
규칙 기반의 접근 방법으로 코호트를 작성해보자. 이번 예제에서는, 고혈압의 초기 치료를 위해 ACE inhibitors 단일 치료를 시작한 환자를 찾을 것이다.
이 연습을 진행하면서 표준 소모 도표 attrition chart와 비슷한 코호트를 작성하게 될 것이다. 그림 10.3은 우리가 어떤 논리로 코호트를 작성할지 보여준다.
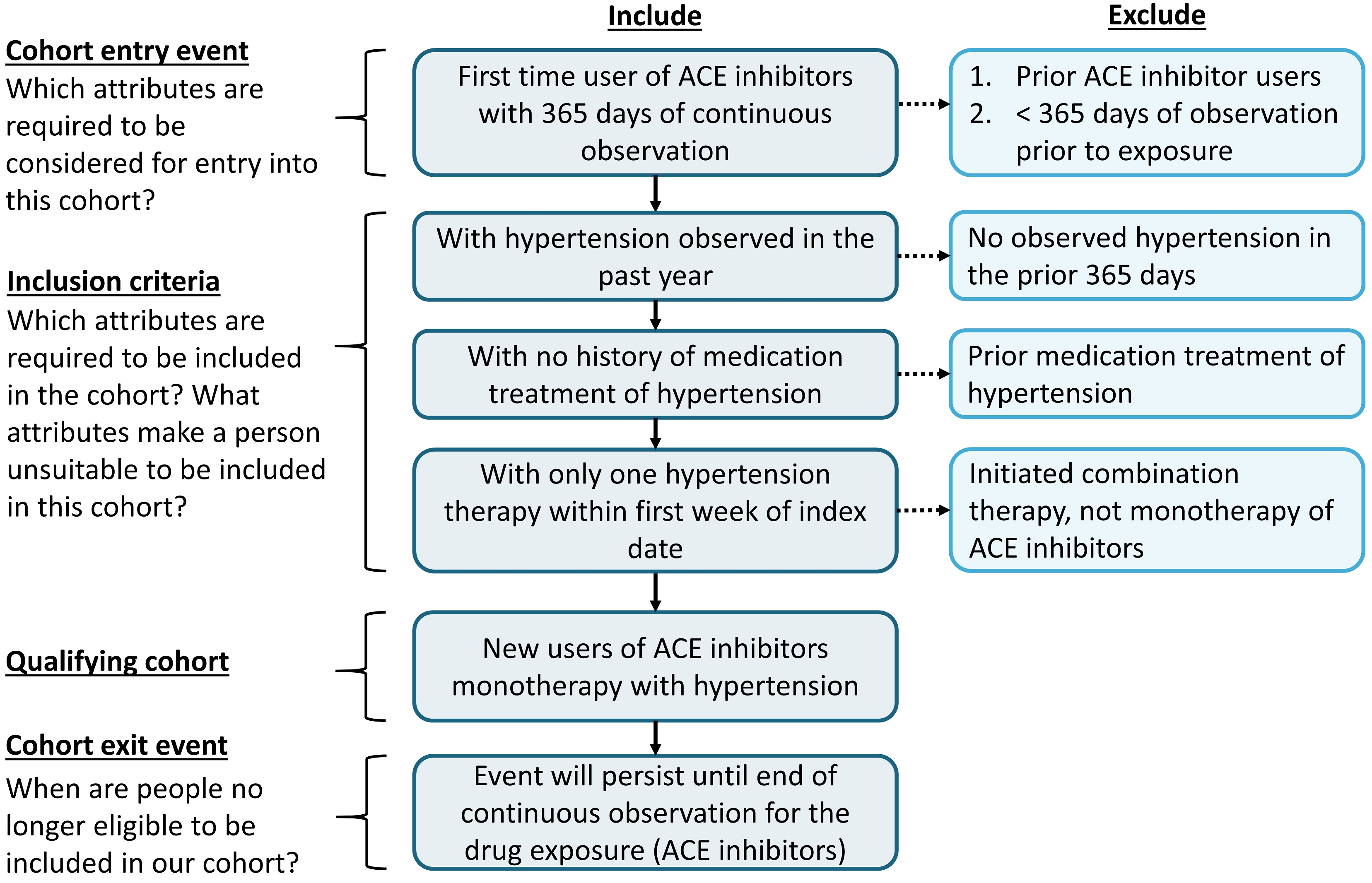
Figure 10.3: 만들고자 하는 코호트의 논리적 구성도
ATLAS 유저 인터페이스를 사용해서 코호트를 작성해도 되고, 쿼리를 직접 작성해도 된다. 이 장에서는 두 가지 방법 모두를 간단히 소개하겠다.
10.7 ATLAS를 이용해 코호트 작성하기
ATLAS를 시작하기 위해  버튼을 클릭한다. 다음으로 ‘New cohort’ 버튼을 클릭한다. 다음 화면에서 비어 있는 코호트를 확인할 수 있을 것이다. 그림 10.4에서 당신이 현재 보고 있는 화면을 확인한다.
버튼을 클릭한다. 다음으로 ‘New cohort’ 버튼을 클릭한다. 다음 화면에서 비어 있는 코호트를 확인할 수 있을 것이다. 그림 10.4에서 당신이 현재 보고 있는 화면을 확인한다.
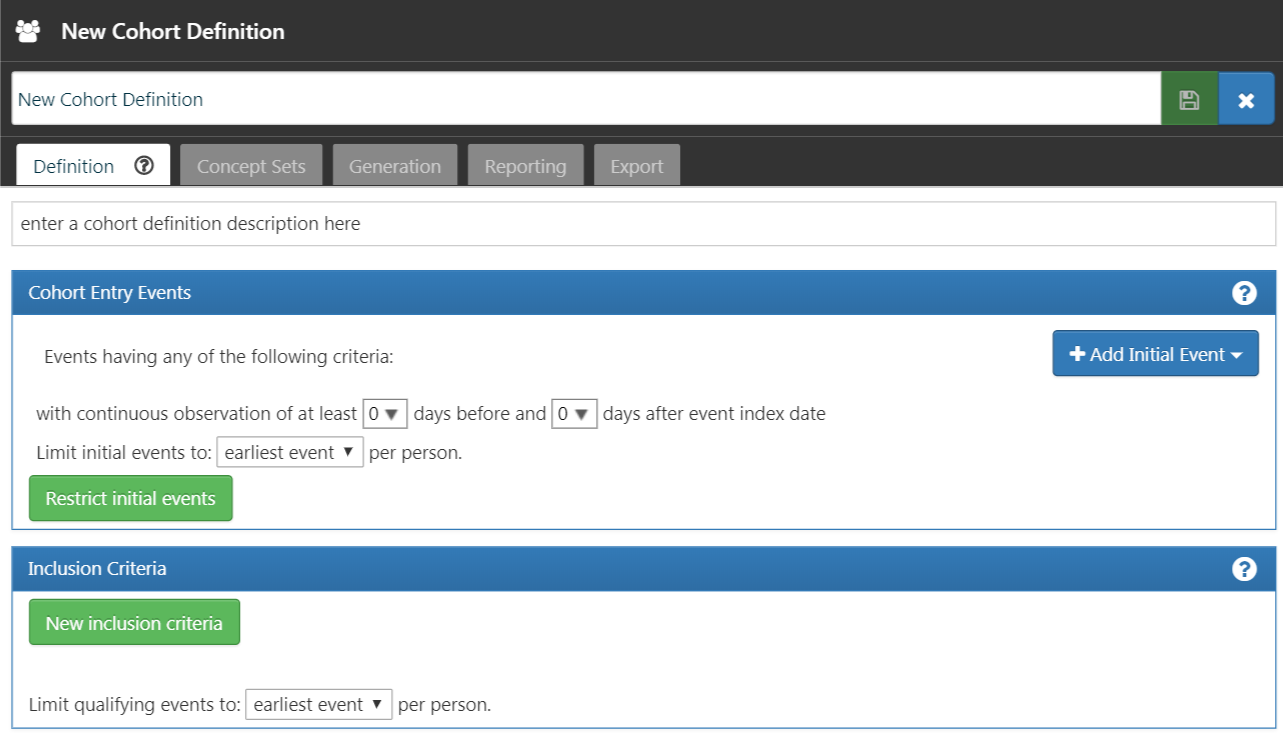
Figure 10.4: 새로운 코호트 정의
먼저 “New Cohort Definition”로 지정된 코호트 이름을 다른 이름으로 바꿔 지어 주기를 추천한다. ’New users of ACE inhibitors as first-line monotherapy for hypertension’라고 지으면 적당할 것이다.
이름을 정했으면,  을 눌러서 코호트를 저장하라.
을 눌러서 코호트를 저장하라.
10.7.1 초기 이벤트 기준 Initial Event Criteria
이제 우리는 초기 코호트 이벤트를 정의해야 한다. “Add initial event”를 클릭한다. 어떤 도메인 내에서 기준을 설정할지 결정해야 한다. 초기 코호트 이벤트를 정의하기 위해 어떤 도메인이 필요한지 어떻게 알 수 있을까? 함께 알아보자.
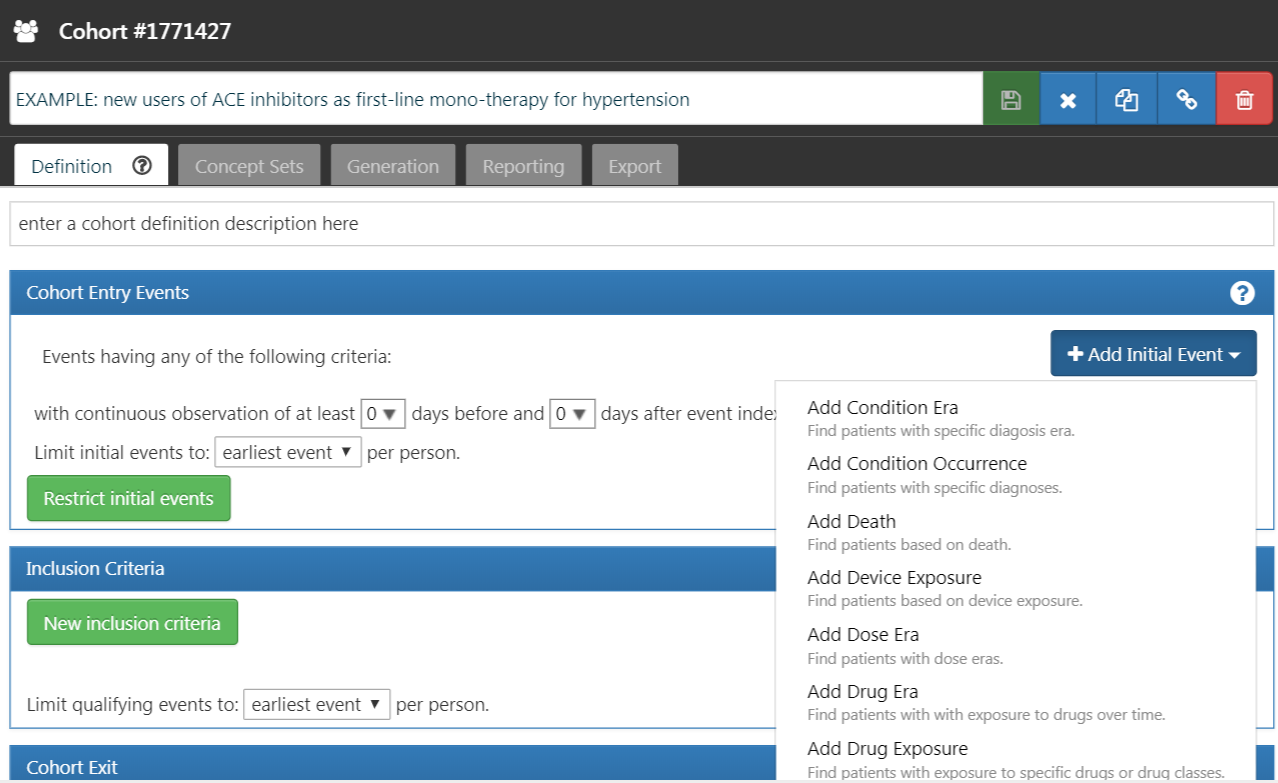
Figure 10.5: 초기 이벤트 추가하기
그림 10.5에서 볼 수 있듯이 ATLAS는 각 기준 아래에 설명을 제공한다. 우리가 만약 특정 질병을 진단받은 환자를 찾으려 한다면 CONDITION_OCCURRENCE 도메인에서 기준을 만들어야 한다. 특정 약물이나 특정 계열의 약물을 복용한 환자를 찾고 싶다면 DRUG_EXPOSURE 도메인에서 기준을 만들어야 한다. 우리는 고혈압의 초 치료로 ACE inhibitors 단독요법을 시행한 환자를 찾고 싶기 때문에 DRUG_EXPOSURE 도메인에서 기준을 만들어야 한다. 그런데 고혈압을 진단받은 환자도 찾아야 하지 않는가? 맞다! 고혈압 진단과 관련해서는 다른 기준을 만들 것이다. 하지만 고혈압 약물을 복용하기 시작한 날짜가 코호트 시작 날짜, 즉 시작 이벤트 initial event가 될 것이다. 고혈압 진단은 소위 추가적 적격 기준 additional qualifying criteria이 된다. 이에 관해서는 뒤에서 다시 설명하겠다. 이제 ’Add Drug Exposure’를 클릭한다.
화면은 당신이 선택한 기준에 따라 업데이트되겠지만, 아직 끝난 것은 아니다. 그림 10.6에서 볼 수 있다시피 ATLAS는 우리가 어떤 약물을 찾고자 하는지 아직 모른다. ATLAS에게 어떤 concept set이 ACE inhibitors와 연관이 있는지 알려주어야 한다.
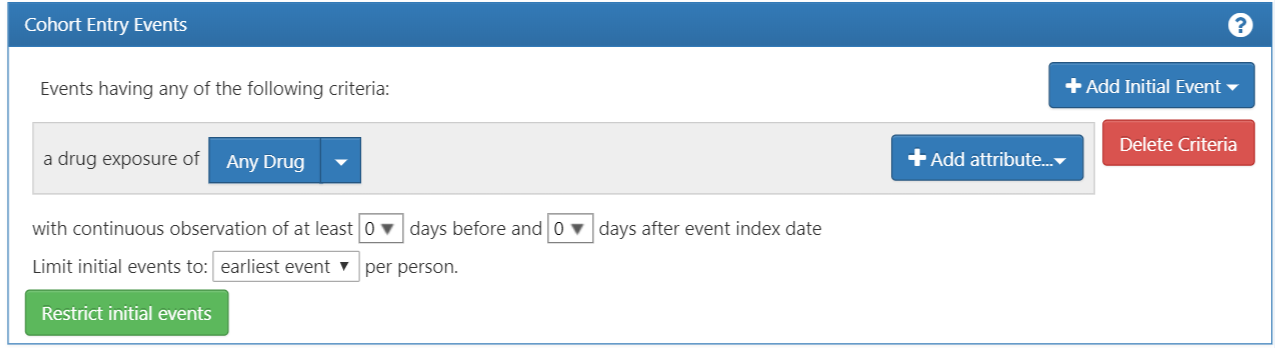
Figure 10.6: 약물 복용에 관하여 정의하기
10.7.2 concept set 정의하기
ACE inhibitors를 정의하기 위한 대화 상자를 열기 위해  을 클릭한다.
을 클릭한다.
시나리오 1: 당신은 아직 concept set을 만들지 않았다
아직 당신의 코호트에 추가할 concept set을 만들지 않았다면, 이것을 먼저 진행해야 한다. ‘Concept set’ 탭의 ’New Concept Set’을 클릭하여 코호트를 작성하는 데 쓰일 concept set을 만들 수 있다. concept set의 이름을 ’Unnamed Concept Set’에서 새로 만들어 주어야 한다. 이제  모듈을 통해 ACE inhibitors를 나타내는 개념을 찾아보자. (그림 10.7)
모듈을 통해 ACE inhibitors를 나타내는 개념을 찾아보자. (그림 10.7)
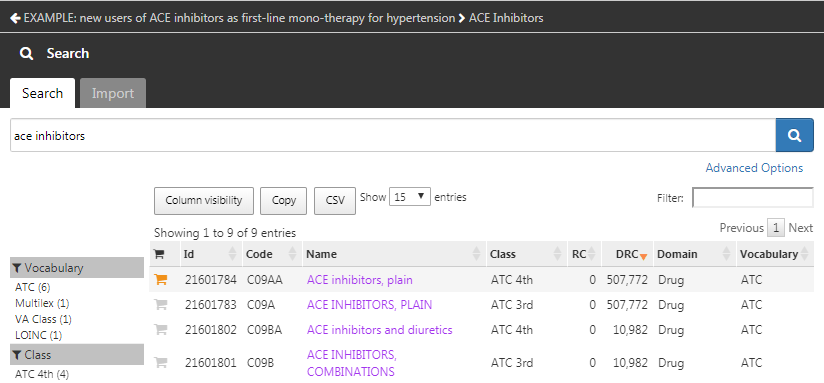
Figure 10.7: ACE Inhibitors 용어 찾기
필요한 용어를 찾았다면,  을 클릭함으로써 그 개념을 선택할 수 있다. 그림 10.7의 좌상단의 왼쪽을 향하는 화살표 버튼을 클릭하여 코호트 작성 페이지로 돌아갈 수 있다. 적절한 용어를 찾기 위한 방법은 5장 (“OMOP 표준 용어”)을 참고한다.
을 클릭함으로써 그 개념을 선택할 수 있다. 그림 10.7의 좌상단의 왼쪽을 향하는 화살표 버튼을 클릭하여 코호트 작성 페이지로 돌아갈 수 있다. 적절한 용어를 찾기 위한 방법은 5장 (“OMOP 표준 용어”)을 참고한다.
그림 10.8에서 우리가 선택한 concept set의 구성을 확인할 수 있다. 우리는 모든 ACE inhibitors 성분을 선택했으며, 하위 개념도 포함했다. ’Included concepts’를 클릭하여 포함된 21,536개의 모든 개념을 확인할 수 있고, ’Included Source Codes’를 클릭하여 모든 원천 코드를 확인할 수도 있다.
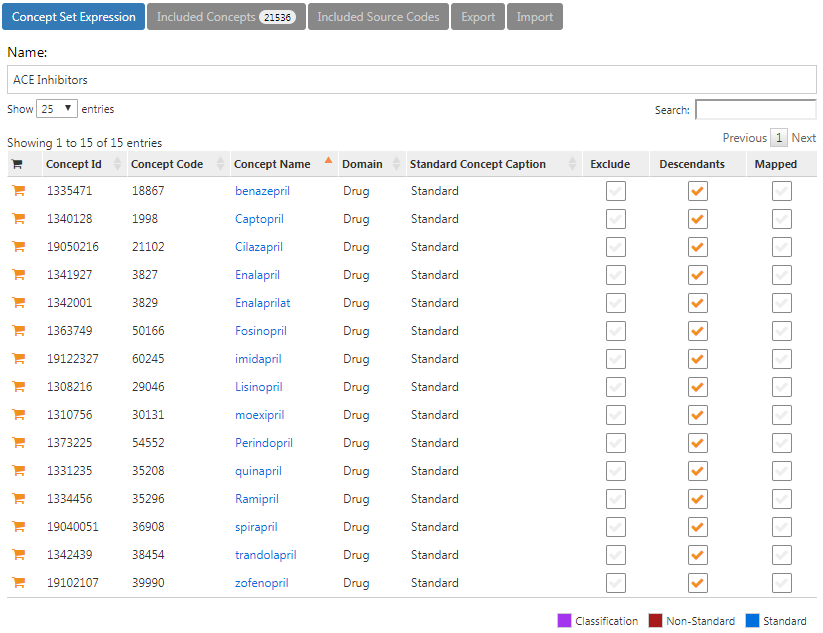
Figure 10.8: ACE inhibitor를 포함한 약물의 concept set
시나리오 2: 당신은 이미 concept set을 만들었다
만약 당신이 이미 concept set을 만들었고, ATLAS에 저장했다면, ’Import Concept Set’을 클릭한다. 그러면 그림 10.9에서 볼 수 있다시피 ATLAS의 concept set 저장소에서 당신의 concept set을 찾을 수 있는 대화창이 뜬다. 이번 예시에서는 사용자가 ATLAS에 저장되어 있던 concept set을 이용한다고 가정하자. 사용자는 검색 창에 ’ACE inhibitors’를 검색하였고, 검색 내용이 이름에 포함된 concept set을 볼 수 있을 것이다. 사용자는 해당하는 concept set을 클릭하여 선택할 수 있다 (참고로 당신이 concept set을 선택하면 대화창은 사라진다). Any Drug 칸이 당신이 선택한 concept set의 이름으로 바뀌어 있다면 성공한 것이다.
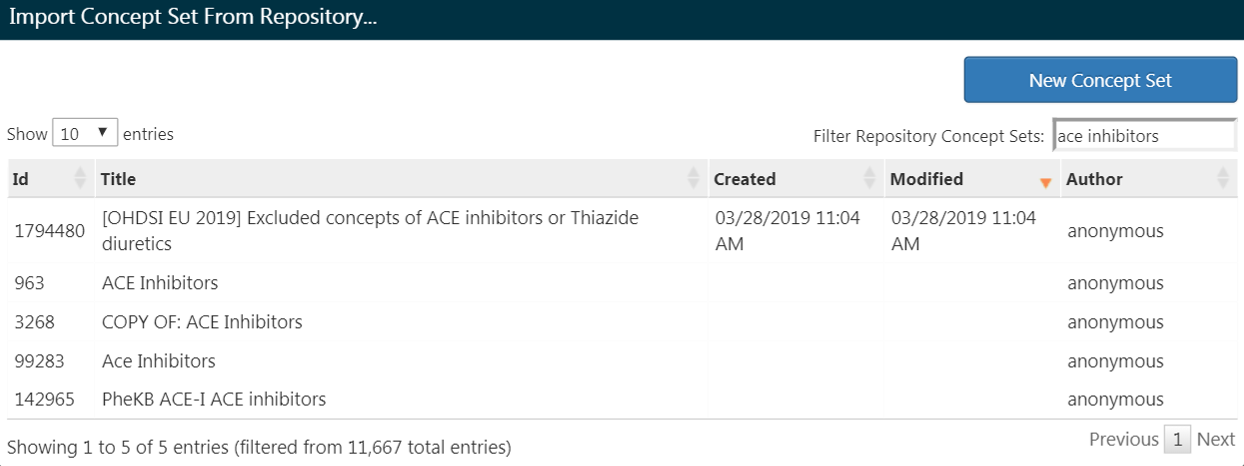
Figure 10.9: ATLAS 저장소에서 concept set 가져오기
10.7.3 추가적 초기 이벤트 기준 Additional Initial Event Criteria
이제 코호트에 concept set을 만들어 붙였지만, 아직 끝난 것이 아니다. 우리는 ACE inhibitors를 태어나서 처음 복용한 사람을 찾고 있다. 이는 ACE inhibitors를 처음 복용한 환자 기록을 찾는 것을 의미한다. 이를 지정하기 위해 당신은 ’+Add attribute’를 클릭하여 ’Add first exposure criteria’를 선택해야 한다. 당신이 만든 기준에 다른 특성도 지정할 수 있다는 것을 참고한다. 약물을 복용한 날짜나 나이, 성별 혹은 약물과 관련한 다른 특성을 지정할 수 있다. 각 도메인에 따라 선택할 수 있는 특성이 다르다.
선택했으면, 창은 자동으로 닫힌다. 선택된 특성은 초기 기준과 같은 칸 안에서 볼 수 있을 것이다 (그림 10.10 참조).
현재 ATLAS 디자인은 활용하기에 약간 혼란스러울 수 있다. 생김새와는 다르게 버튼  는 ’NO’를 의미하는 것이 아니다. 이는 사용자가 해당 기준을 삭제할 수 있도록 만들어진 버튼이다. 만약
는 ’NO’를 의미하는 것이 아니다. 이는 사용자가 해당 기준을 삭제할 수 있도록 만들어진 버튼이다. 만약  를 클릭한다면, 해당 기준은 사라질 것이다. 그러므로 그 기준을 그대로 활성화하고 싶으면,
를 클릭한다면, 해당 기준은 사라질 것이다. 그러므로 그 기준을 그대로 활성화하고 싶으면,  버튼을 그대로 놔두어야 한다.
버튼을 그대로 놔두어야 한다.
이제 만족스러운 초기 이벤트를 설정했다. 환자가 처음으로 약물을 복용했다는 사실을 보증하기 위해, 환자의 그 이전 기록을 확인할 수 있는 충분한 기간을 설정해 주면 좋을 것이다. 짧은 관찰 기간을 가진 환자는 우리가 확인할 수 없는 다른 곳에서 약물을 복용하였을 수도 있다. 우리가 이것을 강제적으로 막을 수는 없지만, 기준일자 index date 이전에 관찰 기간을 설정함으로써 최소한 해당 관찰 기간 동안에는 약물 복용이 이루어지지 않았음을 보증할 수 있다. 이를 위해 관찰 기간을 설정하는 부분이 있으며, 구체적인 관찰 기간을 직접 설정할 수도 있다. 우리는 초기 이벤트 이전에 365일 동안 관찰된 환자를 필요로 한다. 그림 10.10처럼 관찰 기간을 다음과 같이 설정하라: with continuous observation of 365 days before. 당신 연구팀의 재량껏 관찰 기간을 설정하면 된다. 다른 코호트에서는 관찰 기간을 다르게 설정해서 다양한 시도를 해볼 수 있다. 이는 환자의 과거력에 관한 기간이며, 기준일자 이후의 시간은 포함하지 않는다. 그러므로 우리는 0 dates after index date라고 설정해야 한다. 우리는 생에 처음 ACE inhibitors를 복용한 환자를 찾고 싶어서 limit initial events to the “earliest event” per person (한 환자에서 발생한 여러 번의 ACE inhibitor 복용 중, 첫 번째 복용을 초기 이벤트로 설정하는 것)으로 설정한다.
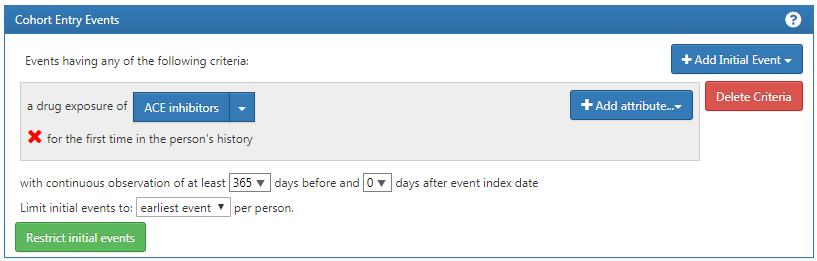
Figure 10.10: Index date 이전에 필요로 하는 관찰 기간 설정하기.
지금껏 설정한 논리를 한눈에 보기 위해서 환자의 타임라인을 설정해볼 수 있다.
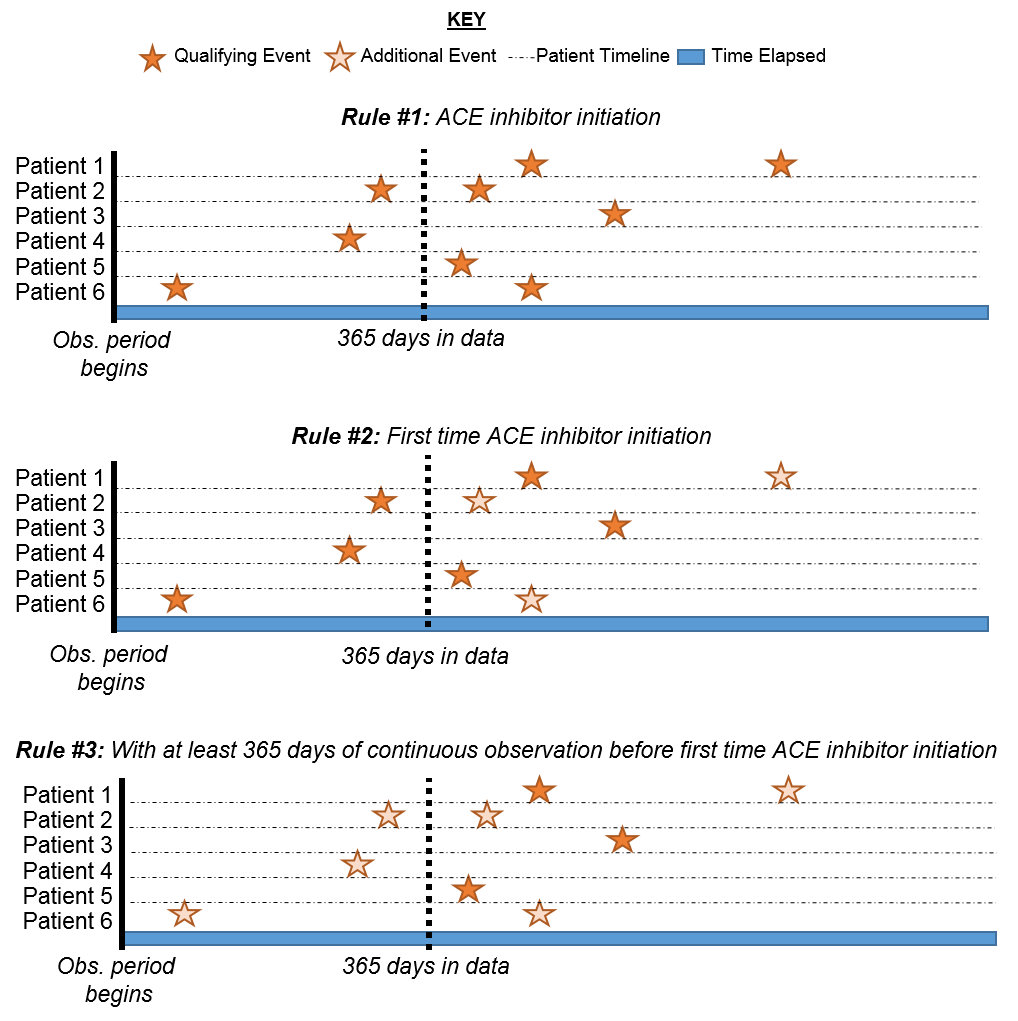
Figure 10.11: 기준이 적용됨에 따라 환자가 코호트에 적합한지 살펴보기
그림 10.11에서 각 행은 코호트에 들어올 자격을 갖출 수 있는 환자 개개인을 나타낸다. 그리고 진한 별은 환자가 특정 기준을 만족했던 시간을 나타낸다. 추가 기준이 설정될수록 진한 별 대신 연한 별이 그려진 것을 볼 수 있다 (즉, 추가 기준에 의해서 코호트에 포함되지 못하고 탈락). 이는 환자가 조건을 모두 만족하는 이벤트도 가지고 있지만, 그렇지 않은 이벤트도 가지고 있음을 의미한다. 마지막 기준을 그린 그림을 보면 ACE inhibitors를 처음으로 복용하였으며, 복용 이전에 최소 365일의 관찰 기간을 가진 환자 (환자 1번, 환자 3번, 환자 5번의 진한 별은 관찰에 포함; 환자 1번의 연한 별은 관찰에서 탈락)를 확인할 수 있다. 당신의 코호트를 설계할 때 OHDSI 포럼에 참여하는 연구자의 의견을 참고하면 더 좋을 것이다.
10.7.4 포함 기준 Inclusion Criteria
코호트 진입 이벤트를 설정했으면, 다음 두 옵션을 통해 추가적 이벤트를 설정할 수 있다: ‘Restrict initial events’, 그리고 ‘New inclusion criteria’. 이 두 옵션 사이에는 ATLAS가 사용자에게 어떤 임시 정보를 제공하는가의 차이가 있다. 만약 당신이 기준을 추가하기 위해 ’Restrict initial events’를 사용한다면, ATLAS에서 조건에 맞는 대상 환자 수를 셀 때, 모든 기준을 충족시키는 사람의 숫자만을 얻게 될 것이다. ’New inclusion criteria’를 통해 기준을 추가한다면, 추가 포함 기준을 적용하여 손실된 환자 수를 보여주는 감소 차트를 확인할 수 있을 것이다. 당신이 추가한 기준에 의해 얼마나 큰 손실이 발생하는지 단계별로 보여주는 감소 차트를 확인하는 것은 중요하기 때문에 ’New inclusion criteria’를 통해 기준을 추가하는 것을 권장한다. 이를 통해 코호트에 포함되는 환자 수를 급격하게 감소시키는 기준이 무엇인지 확인할 수 있다. 당신은 해당 기준을 완화하여 더욱 큰 코호트를 얻을 수 있다. 이것은 궁극적으로 이 코호트를 설계하는 전문가의 재량에 달려있다.
이제 ’New inclusion criteria’를 통해 기준을 추가해보자. 이는 위에서 코호트 기준을 설정한 것과 동일한 방법으로 하면 된다. 특정 기준을 만들어서 넣은 다음, 특정 속성을 추가할 수 있을 것이다. 우리가 첫 번째로 추가할 기준은 다음과 같다: ACE inhibitors 약물을 복용한 시점 이후 0~365일 이내에 최소 1회 고혈압이 발생한 사람. ’New inclusion criteria’를 클릭한 다음, 그 기준을 설명해줄 수 있는 이름을 정한다. 그래야 나중에 이 코호트를 다시 보았을 때 자신이 무엇을 만들었는지 헷갈리지 않을 것이다.
이 새로운 기준에 이름을 달고 난 다음, “+Add criteria to group” 버튼을 클릭하여 여러 규칙을 담은 기준을 설계한다. 이 버튼은 “Add Initial Event”와 비슷한데, 다만 “+Add criteria to group” 버튼은 초기 이벤트를 설계하고 수정하는 버튼이 아니다. 우리는 여기서 여러 개의 기준을 추가할 수 있다. 예를 들어 질병의 발생을 확인하는 여러 가지 방법을 가지고 있다고 가정하자 (예를 들어 CONDITION_OCCURRENCE, 혹은 DRUG_EXPOSURE, 혹은 MEASUREMENT를 사용한 방법). 모두 다른 도메인이고 각각 다른 기준이 필요하겠지만 특정 조건을 찾는 하나의 기준으로 그룹화할 수 있다. 이 경우에는, 우리는 고혈압의 진단을 찾고 싶기 때문에 “Add condition occurrence”를 선택한다. 여기에 적절한 concept set을 붙이는 등 초기 이벤트를 설정할 때와 비슷하게 하면 된다. 또한, ACE inhibitor를 처음 복용한 날 index date로 이후 0~365일의 기간을 설정한다. 그림 10.12와 같이 작성될 수 있을 것이다.
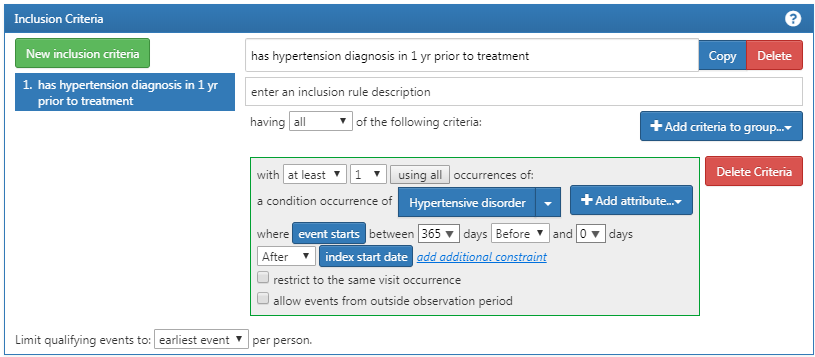
Figure 10.12: 추가적 포함 기준 1
아마도 환자를 탐색할 또 다른 기준을 추가하고 싶을 것이다: with exactly 0 occurrences of hypertension drugs ALL days before and 1 day before index start date (ACE inhibitor 투여 이전에 어떠한 고혈압 약물도 복용하지 않은 사람). (역자 주: xx before and yy 구문은 항상 혼란스럽지만, from xx to yy로 고쳐서 생각하면 이해하기 쉽다. 즉 해당 조건의 시작과 끝을 지정하는 용법이다. 앞선 예라면 “과거 전체로부터 시작해서 (ACE inhibitor가 최초 투여된) 기준 날짜 index date 바로 하루 전까지 고혈압치료제를 한 번도 복용하지 않은 경우”가 된다) 먼저 “New inclusion criteria”를 클릭해 당신의 기준을 설정한 다음, “+Add criteria to group”을 클릭한다. 이는 DRUG_EXPOSURE의 영역이니 “Add Drug Exposure”를 클릭한 다음, 고혈압 약물의 concept set을 붙인다. 그리고, index date로부터 ALL days before and 0 days after라는 시간을 설정해준다 (역자 주: “ALL days before and 0 days after” 는 “ALL days before and 0 days before”와 같은 의미이며 기준 날짜 index date를 포함하여 그날까지의 의미이다. 그림에는 “ALL days before and 1 days before”로 표현했는데 과거 전체로부터 기준 날짜 index date 하루 전까지의 의미이다. 본인이 원하는 기준이 무엇인지에 따라 구분하여 사용하라). exactly 0 occurrence를 선택하였는지 다시 한번 확인하고 그림 10.13과 같이 잘 만들어졌는지 확인한다.
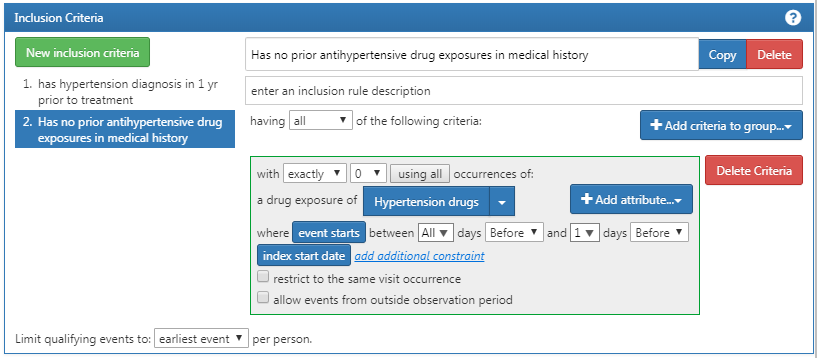
Figure 10.13: 추가적 포함 기준 2
“having no occurrences (발생하지 않았다)”라는 말이 왜 “exactly 0 occurrences (발생 횟수 0회)”라고 쓰이는지 혼란스러울 수 있다. 이는 ATLAS가 사용하는 규칙이다. ATLAS는 오직 포함 기준만을 사용하고, 제외 기준을 사용하지 않는다. 만약 당신이 어떤 특성을 가진 환자를 제외하고 싶다면 해당 특성을 0회 가지는 환자를 포함한다는 말로 대체하여야 한다. 처음에는 헷갈릴 수 있지만 계속 사용하다 보면 이러한 논리가 익숙해질 것이다.
마지막으로 목표 환자군 설정을 위한 기준을 하나 더 추가해야 한다: with exactly 1 occurrence of hypertension drugs between 0 days before and 7 days after index start date AND can only start one HT drug (an ACE inhibitor) – index date 이후 0~7일 동안 정확히 1회의 항고혈압제 처방이 발생했으며, 반드시 ACE inhibitor로 고혈압 약물치료를 시작해야 한다. 먼저 “New inclusion criteria”를 클릭해 당신의 기준을 설정한 다음, “+Add criteria to group”을 클릭한다. DRUG_ERA 를 사용하여 “Add Drug Era”를 클릭한 다음, 고혈압 약물의 concept set을 붙였다. (역자 주: Drug era는 9.7.1에 간략히 설명되어 있는데 약물 노출 테이블에서 계산된 것으로 연속으로 처방된 동일한 성분의 여러 약물 노출을 합쳐서 하나의 기간으로 표현한 것이다. 동일한 성분의 약물 노출 간에 30일 이상의 공백이 있으면 다른 drug era로 계산된다. 이 점은 condition era도 마찬가지이다) 그리고 index date 이후 0~7일이라는 시간을 설정해준다. 그림 10.14를 통해 진행된 모습을 확인한다.
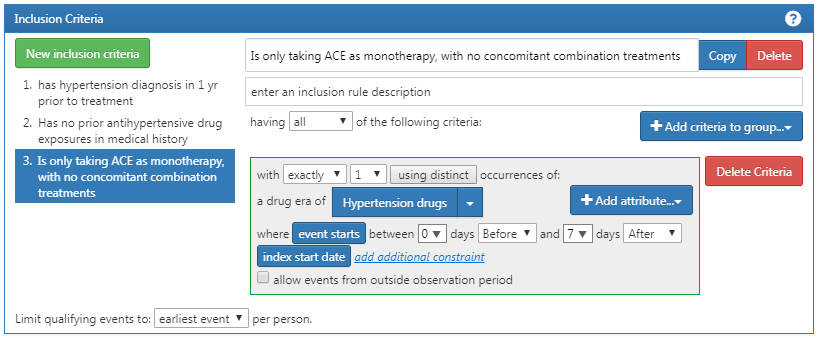
Figure 10.14: 추가적 포함 기준 3
10.7.5 코호트 종료 기준 Cohort Exit Criteria
이제 모든 적절한 포함 기준을 추가했다. 다음으로 코호트 종료 기준을 정해야 한다. 사람이 더 이상 이 코호트에 포함될 자격이 없어질 때는 언제일지 생각해보아야 할 것이다. 우리는 이 코호트에서 약물을 처음 복용한 사람을 추적한다. 즉, 약물 복용을 중단한 시점에 환자는 코호트에서 나오게 하면 된다. 약물 복용이 중단되는 동안에는 해당 환자에게 무슨 일이 일어나는지 확인할 수 없기 때문이다. 또한 약물 복용 사이에 허용되는 공백 기간을 지정하기 위해 persistence 창에서 기준을 설정할 수 있다. 이 연구에서 전문가는 약물 복용 사이에 최대 30일의 공백 기간은 허용된다고 결론지었다.
왜 공백 기간이 허용되는가? 데이터 모음에서 실제로 이루어지는 일의 일부만 관찰할 수 있을 뿐이다. 특히 환자의 약물 복용에 관한 정보는 처방전의 기록으로 확인한다. 그리고 처방전을 통해 하루 치 이상의 약을 처방하기 때문에 기록이 비어 있는 시간 동안에도 환자가 약을 복용하고 있다는 합리적 추론이 가능하다.
Event will persist “end of a continuous drug exposure”를 선택하고, persistence 창에 “allow for a maximum of 30 days”를 추가한 다음 ‘ACE inhibitor’ concept 모음을 추가로 지정해 주면 된다. 그림 10.15를 통해 이를 확인한다.
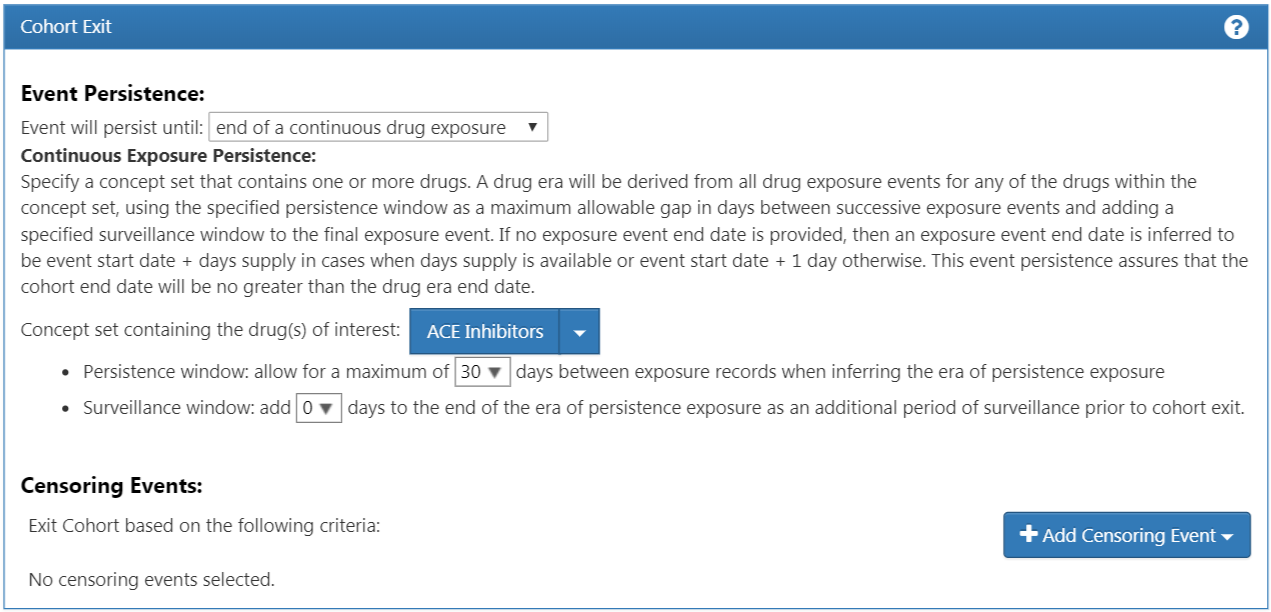
Figure 10.15: 코호트 종료 기준
이 코호트의 경우 다른 중도 절단 사건 Censoring event는 선택되지 않았다. 하지만 Censoring event를 추가해야 하는 다른 코호트를 만들어야 한다면 다른 속성을 추가했던 것과 비슷하게 진행하면 된다. 이제 코호트를 성공적으로 만들었다. 반드시  버튼을 눌러야 한다. 축하한다! 코호트를 만드는 것은 OHDSI가 제공하는 도구를 이용할 때 가장 중요한 부분이다. 이제 ‘Export’ 탭을 클릭하면 ATLAS에 당신이 정의한 코호트가 SQL 코드와 JSON 파일로 저장되어 다른 연구자와 공유할 수 있다.
버튼을 눌러야 한다. 축하한다! 코호트를 만드는 것은 OHDSI가 제공하는 도구를 이용할 때 가장 중요한 부분이다. 이제 ‘Export’ 탭을 클릭하면 ATLAS에 당신이 정의한 코호트가 SQL 코드와 JSON 파일로 저장되어 다른 연구자와 공유할 수 있다.
10.8 SQL을 사용하여 코호트 구현하기
여기서는 동일한 코호트를 SQL과 R을 이용하여 작성하는 방법을 설명할 것이다. 9장에서 설명하였듯이 OHDSI는 SqlRender, DatabaseConnector라는 두 개의 R 패키지를 제공하는데, 이는 SQL의 코드가 다양한 플랫폼에서 실행될 수 있게끔 SQL문을 자동으로 번역해준다.
자세한 설명을 위해 SQL 코드를 여러 개의 단계로 나눌 것이고, 각 단계에서는 다음 단계에 필요한 임시 테이블이 생성될 것이다. 이런 설명 방법이 가장 효율적이지는 않겠지만 매우 긴 단일 명령문을 읽는 것보단 쉬울 것이다.
10.8.1 데이터베이스에 연결하기
먼저 R이 어떻게 서버에 접속하는지 알려주어야 한다. createConnectionDetails 라는 기능을 가진 DatabaseConnector 패키지를 사용할 것이다. 다양한 데이터베이스 관리 시스템(DBMS)에 연결하는 데 필요한 설정을 확인하려면 ?createConnectionDetails 과 같이 입력한다. 예를 들어 아래의 코드를 이용해 PostgreSQL에 연결할 수 있다:
library(CohortMethod)
connDetails <- createConnectionDetails(dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
cdmDbSchema <- "my_cdm_data"
cohortDbSchema <- "scratch"
cohortTable <- "my_cohorts"마지막 3줄은 변수 cdmDbSchema, cohortDbSchema, 그리고 cohortTable을 정의한다. 나중에 이 변수를 R에게 CDM 포맷의 데이터가 어디에 있으며, 우리가 만든 코호트가 어디에 생성되어야 하는지 알려주기 위해 사용할 것이다. Microsoft SQL Server에서는 cdmDbSchema <- "my_cdm_data.dbo"의 예시와 같이 데이터베이스와 스키마 모두를 지정해 주어야 함을 참고한다.
10.8.2 개념 결정하기
가독성을 위해 R에 필요한 개념 ID를 정의하고 SQL에 전달한다:
aceI <- c(1308216, 1310756, 1331235, 1334456, 1335471, 1340128, 1341927,
1342439, 1363749, 1373225)
hypertension <- 316866
allHtDrugs <- c(904542, 907013, 932745, 942350, 956874, 970250, 974166,
978555, 991382, 1305447, 1307046, 1307863, 1308216,
1308842, 1309068, 1309799, 1310756, 1313200, 1314002,
1314577, 1317640, 1317967, 1318137, 1318853, 1319880,
1319998, 1322081, 1326012, 1327978, 1328165, 1331235,
1332418, 1334456, 1335471, 1338005, 1340128, 1341238,
1341927, 1342439, 1344965, 1345858, 1346686, 1346823,
1347384, 1350489, 1351557, 1353766, 1353776, 1363053,
1363749, 1367500, 1373225, 1373928, 1386957, 1395058,
1398937, 40226742, 40235485)10.8.3 약물을 처음 복용한 환자 찾기
먼저 각 환자에 대한 ACE inhibitor의 첫 복용을 찾을 것이다:
conn <- connect(connectionDetails)
sql <- "SELECT person_id AS subject_id,
MIN(drug_exposure_start_date) AS cohort_start_date
INTO #first_use
FROM @cdm_db_schema.drug_exposure
INNER JOIN @cdm_db_schema.concept_ancestor
ON descendant_concept_id = drug_concept_id
WHERE ancestor_concept_id IN (@ace_i)
GROUP BY person_id;"
renderTranslateExecuteSql(conn,
sql,
cdm_db_schema = cdmDbSchema,
ace_i = aceI)DRUG_EXPOSURE 테이블을 CONCEPT_ANCESTOR 테이블에 조인함으로써 ACE inhibitor를 포함하는 모든 약물을 찾았다는 것을 참고한다.
10.8.4 약물 복용 이전 최소 365일 동안 관찰될 수 있었던 환자
OBSERVATION_PERIOD 테이블을 조인하여 약물 복용 이전 최소 365일 동안 관찰될 수 있었던 환자를 선택해야 한다:
sql <- "SELECT subject_id,
cohort_start_date
INTO #has_prior_obs
FROM #first_use
INNER JOIN @cdm_db_schema.observation_period
ON subject_id = person_id
AND observation_period_start_date <= cohort_start_date
AND observation_period_end_date >= cohort_start_date
WHERE DATEADD(DAY, 365, observation_period_start_date) < cohort_start_date;"
renderTranslateExecuteSql(conn, sql, cdm_db_schema = cdmDbSchema)10.8.5 이전에 고혈압을 진단받은 환자
기준 날짜 index date로부터 365일 이내에 고혈압 진단을 받은 환자여야 한다:
sql <- "SELECT DISTINCT subject_id,
cohort_start_date
INTO #has_ht
FROM #has_prior_obs
INNER JOIN @cdm_db_schema.condition_occurrence
ON subject_id = person_id
AND condition_start_date <= cohort_start_date
AND condition_start_date >= DATEADD(DAY, -365, cohort_start_date)
INNER JOIN @cdm_db_schema.concept_ancestor
ON descendant_concept_id = condition_concept_id
WHERE ancestor_concept_id = @hypertension;"
renderTranslateExecuteSql(conn,
sql,
cdm_db_schema = cdmDbSchema,
hypertension = hypertension)SELECT DISTINCT를 사용하여 과거에 여러 번의 고혈압 진단을 받은 환자가 여러 번의 코호트 진입을 하지 않도록 했다.
10.8.6 사전에 받은 치료가 없어야 함
이전에 어떠한 고혈압 약물이라도 복용해서는 안 된다:
sql <- "SELECT subject_id,
cohort_start_date
INTO #no_prior_ht_drugs
FROM #has_ht
LEFT JOIN (
SELECT *
FROM @cdm_db_schema.drug_exposure
INNER JOIN @cdm_db_schema.concept_ancestor
ON descendant_concept_id = drug_concept_id
WHERE ancestor_concept_id IN (@all_ht_drugs)
) ht_drugs
ON subject_id = person_id
AND drug_exposure_start_date < cohort_start_date
WHERE person_id IS NULL;"
renderTranslateExecuteSql(conn,
sql,
cdm_db_schema = cdmDbSchema,
all_ht_drugs = allHtDrugs)Left join을 사용했으며, DRUG_EXPOSURE 테이블의 person_id 행이 NULL인 (일치하는 기록이 없음을 의미) 행만 찾을 수 있도록 했다는 점에 유의한다.
10.8.7 단독 요법
코호트에 진입하고 첫 1주일 동안은 고혈압 치료 약물에 단 한 번만 노출되도록 설정할 필요가 있다 (역자 주: 아래 코드는 입원환자에게는 정확히 적용되지 않을 수 있다. 만일 입원하여 하루 단위로 고혈압 처방이 이루어진다면 기준 날짜 index date로부터 1주일 이내에 여러 번의 고혈압 처방 start date가 생성되며 아래 코드에 의하면 해당 환자의 두 번째 처방으로 인해 그 환자는 코호트에서 탈락한다. 이러한 점을 피하려면 drug_exposure 테이블 대신에 drug_era table을 이용하면 될 것이다. drug_era 테이블에서는 30일 이내에 처방된 같은 동일 성분명의 약물 노출은 서로 합쳐서 하나의 노출로 만들어 준다. 정확히는 약 처방일 + 처방된 기간 (day supply) + 30을 판단 기준으로 한다. 예를 들어 14일 처방을 냈을 경우 처방 낸 날 + 14 + 30 이내에 같은 성분명의 약물이 다시 처방되면 같은 약물 처방으로 간주하여 하나의 drug_dra로 그 두 처방 (혹은 이후 계속되는 동일 성분명 처방 전체)을 묶어준다. 10.8.8 코호트 종료에서 drug era로 묶는 코드가 제시되고 있다):
sql <- "SELECT subject_id,
cohort_start_date
INTO #monotherapy
FROM #no_prior_ht_drugs
INNER JOIN @cdm_db_schema.drug_exposure
ON subject_id = person_id
AND drug_exposure_start_date >= cohort_start_date
AND drug_exposure_start_date <= DATEADD(DAY, 7, cohort_start_date)
INNER JOIN @cdm_db_schema.concept_ancestor
ON descendant_concept_id = drug_concept_id
WHERE ancestor_concept_id IN (@all_ht_drugs)
GROUP BY subject_id,
cohort_start_date
HAVING COUNT(*) = 1;"
renderTranslateExecuteSql(conn,
sql,
cdm_db_schema = cdmDbSchema,
all_ht_drugs = allHtDrugs)10.8.8 코호트 종료 Cohort Exit
이제 코호트 종료 일자를 제외하고 코호트를 완전히 지정했다. 코호트는 노출이 중단되면 종료되도록 정의되며, 노출 사이에 최대 30일의 간격까지는 허용된다. 즉, 약물의 복용 시작뿐만 아니라 ACE inhibitor의 후속 복용에 대해서도 고려한다는 말이다. SQL을 통해 약물의 후속 복용을 고려하여 약물 복용 기간을 정의하는 것은 매우 복잡하다. 운이 좋게도 약물 복용 기간을 효율적으로 만들 수 있는 표준 코드가 작성되었다. 이 코드는 Chris Knoll이 작성했으며 OHDSI 내에서 종종 마법이라고 불리는 코드이기도 하다. 먼저 병합하려는 모든 약물 복용을 포함하는 임시 테이블을 만든다:
sql <- "
SELECT person_id,
CAST(1 AS INT) AS concept_id,
drug_exposure_start_date AS exposure_start_date,
drug_exposure_end_date AS exposure_end_date
INTO #exposure
FROM @cdm_db_schema.drug_exposure
INNER JOIN @cdm_db_schema.concept_ancestor
ON descendant_concept_id = drug_concept_id
WHERE ancestor_concept_id IN (@ace_i);"
renderTranslateExecuteSql(conn,
sql,
cdm_db_schema = cdmDbSchema,
ace_i = aceI)그런 다음 순차적 복용을 병합하기 위한 표준 코드를 실행한다:
sql <- "
SELECT ends.person_id AS subject_id,
ends.concept_id AS cohort_definition_id,
MIN(exposure_start_date) AS cohort_start_date,
ends.era_end_date AS cohort_end_date
INTO #exposure_era
FROM (
SELECT exposure.person_id,
exposure.concept_id,
exposure.exposure_start_date,
MIN(events.end_date) AS era_end_date
FROM #exposure exposure
JOIN (
--cteEndDates
SELECT person_id,
concept_id,
DATEADD(DAY, - 1 * @max_gap, event_date) AS end_date
FROM (
SELECT person_id,
concept_id,
event_date,
event_type,
MAX(start_ordinal) OVER (
PARTITION BY person_id ,concept_id ORDER BY event_date,
event_type ROWS UNBOUNDED PRECEDING
) AS start_ordinal,
ROW_NUMBER() OVER (
PARTITION BY person_id, concept_id ORDER BY event_date,
event_type
) AS overall_ord
FROM (
-- select the start dates, assigning a row number to each
SELECT person_id,
concept_id,
exposure_start_date AS event_date,
0 AS event_type,
ROW_NUMBER() OVER (
PARTITION BY person_id, concept_id ORDER BY exposure_start_date
) AS start_ordinal
FROM #exposure exposure
UNION ALL
-- add the end dates with NULL as the row number, padding the end dates by
-- @max_gap to allow a grace period for overlapping ranges.
SELECT person_id,
concept_id,
DATEADD(day, @max_gap, exposure_end_date),
1 AS event_type,
NULL
FROM #exposure exposure
) rawdata
) events
WHERE 2 * events.start_ordinal - events.overall_ord = 0
) events
ON exposure.person_id = events.person_id
AND exposure.concept_id = events.concept_id
AND events.end_date >= exposure.exposure_end_date
GROUP BY exposure.person_id,
exposure.concept_id,
exposure.exposure_start_date
) ends
GROUP BY ends.person_id,
concept_id,
ends.era_end_date;"
renderTranslateExecuteSql(conn,
sql,
cdm_db_schema = cdmDbSchema,
max_gap = 30)이 코드는 모든 후속 복용을 병합하며, max_gap의 변수를 통해 약물 복용 사이에 허용되는 최대기간을 정의할 수 있다. 그 결과로 작성된 약물 복용 기간은 임시 테이블 #exposure_era라고 불리는 임시 테이블에 기록된다.
다음으로 ACE inhibitor 복용 기간을 우리의 기존 코호트에 조인하기만 하면, ACE inhibitor 복용 종료 날짜를 코호트 종료 날짜로써 사용할 수 있게 된다.
sql <- "SELECT ee.subject_id,
CAST(1 AS INT) AS cohort_definition_id,
ee.cohort_start_date,
ee.cohort_end_date
INTO @cohort_db_schema.@cohort_table
FROM #monotherapy mt
INNER JOIN #exposure_era ee
ON mt.subject_id = ee.subject_id
AND mt.cohort_start_date = ee.cohort_start_date;"
renderTranslateExecuteSql(conn,
sql,
cohort_db_schema = cohortDbSchema,
cohort_table = cohortTable)이제 정의한 최종 코호트를 스키마와 테이블에 저장해야 한다. 코호트 정의 ID를 1로 설정하여 동일한 테이블에 저장된 다른 코호트와 구별할 것이다.
10.8.9 정리하기
마지막으로, 작성된 임시 테이블을 정리하고 데이터베이스 서버와의 연결을 끊는 것이 좋다:
sql <- "TRUNCATE TABLE #first_use;
DROP TABLE #first_use;
TRUNCATE TABLE #has_prior_obs;
DROP TABLE #has_prior_obs;
TRUNCATE TABLE #has_ht;
DROP TABLE #has_ht;
TRUNCATE TABLE #no_prior_ht_drugs;
DROP TABLE #no_prior_ht_drugs;
TRUNCATE TABLE #monotherapy;
DROP TABLE #monotherapy;
TRUNCATE TABLE #exposure;
DROP TABLE #exposure;
TRUNCATE TABLE #exposure_era;
DROP TABLE #exposure_era;"
renderTranslateExecuteSql(conn, sql)
disconnect(conn)10.9 요약
코호트는 일정 기간 동안 하나 이상의 포함 기준을 만족시키는 사람의 집합이다.
코호트 정의는 특정 코호트를 식별하는 데 사용되는 논리에 대한 설명이다.
코호트는 OHDSI 분석 도구 전체에서 사용 (및 재사용) 될 수 있다.
코호트를 작성하기 위한 두 가지 주요 접근 방법이 있다: 규칙 기반 정의, 확률적 정의
- 규칙 기반의 코호트 정의는 ATLAS나 SQL을 통해 작성할 수 있다.
10.10 예제
전제조건
첫 번째 예제로, ATLAS에 접근이 필요하다. http://atlas-demo.ohdsi.org를 통해 접속하거나, 다른 접속 방법을 이용해도 된다.
Exercise 10.1 ATLAS를 이용하여 아래의 기준에 따라 코호트를 작성하라:
- diclofenac을 복용하기 시작한 환자
- 16세 이상의 환자
- 약물 복용 이전 최소 365일간 관찰이 되어 있던 환자
- 이전에 Non-Steroidal Anti-Inflammatory Drug(NSAID)를 복용하지 않은 환자
- 이전에 암을 진단받지 않은 환자
- 약물 복용 중단을 코호트 종료로 정의 (30일 이하의 약물 미복용 기간은 허용)
전제조건
두 번째 예제를 수행하기 위해서 8.4.5절에서 설명된 것처럼 R과 R-Studio, 그리고 JAVA가 설치되어 있다고 가정한다. 또한 아래의 코드를 사용하여 SqlRender, DatabaseConnector, 그리고 Eunomia 패키지를 설치하라:
install.packages(c("SqlRender", "DatabaseConnector", "devtools"))
devtools::install_github("ohdsi/Eunomia", ref = "v1.0.0")Eunomia 패키지는 로컬 R 세션에서 수행될 CDM 데이터를 제공한다. 아래의 코드를 이용하여 연결할 수 있다.
connectionDetails <- Eunomia::getEunomiaConnectionDetails()CDM 데이터베이스 스키마는 ‘main’ 이다.
Exercise 10.2 다음 기준에 따르도록 현재 존재하는 코호트 테이블 안에서 급성 심근경색 Acute Myocardial Infarction 코호트를 SQL과 R을 이용하여 만들어 보자:
- 심근 경색을 진단받은 사람 (개념 4329847 ’심근경색 Myocardial infarction’과 그 하위 개념에서 개념 314666 ’과거 심근경색 Old myocardial infarction’과 그 모든 하위 개념을 제외하기)
- 입원환자 혹은 응급실 방문 환자만 선택 (개념 9201 ‘Inpatient Visit’, 9203 ‘Emergency Room Visit’, 262 ‘Emergency Room and Inpatient Visit’)
제안된 답변은 부록 E.6에서 찾을 수 있다.
References
Banda, J. M., Y. Halpern, D. Sontag, and N. H. Shah. 2017. “Electronic phenotyping with APHRODITE and the Observational Health Sciences and Informatics (OHDSI) data network.” AMIA Jt Summits Transl Sci Proc 2017: 48–57.
Hripcsak, G., N. Shang, P. L. Peissig, L. V. Rasmussen, C. Liu, B. Benoit, R. J. Carroll, et al. 2019. “Facilitating phenotype transfer using a common data model.” J Biomed Inform, July, 103253.
Swerdel, J. N., G. Hripcsak, and P. B. Ryan. 2019. “PheValuator: Development and Evaluation of a Phenotype Algorithm Evaluator.” J Biomed Inform, July, 103258.