Chapter 16 임상적 타당성
Chapter leads: Joel Swerdel, Seng Chan You, Ray Chen & Patrick Ryan
사람이 질량을 에너지로 바꾸는 일의 가능성은 눈을 가리고 총을 쏘아 나는 새를 맞출 가능성과 마찬가지다. 알베르트 아인슈타인, 1935
OHDSI의 이상은 ‘관찰형 연구를 통하여 세계에 건강과 질병에 대한 포괄적인 이해를 제공’ 하는 것이다. 후향적 연구는 이미 존재하는 데이터를 이용할 수 있다는 편리함이 있지만, 이전 14장에서 기술한 바와 같이 다양한 타당성의 한계를 지니고 있다. 데이터의 질과 통계적 방법론과 별개로 임상적 타당성 clinical validity을 논하기는 쉽지 않지만 이번 장에서는 보건의료 데이터의 특성, 코호트 정의 검증, 근거의 일반화, 이 세 가지에 집중해보려 한다. 인구 수준 추정(PLE) 12장의 예제로 돌아가 보자. 앞장에서 ’ACE inhibitor가 thiazide 또는 thiazide-like diuretics에 비해 혈관부종 위험성이 높은가?’에 대해 대답하려 했었고, ACE inhibitor가 thiazide 또는 thiazide-like diuretics에 비해 혈관부종을 더 많이 야기함을 증명했었다. 이번 장은 다음의 질문에 답하기 위해 쓰였다: “수행한 분석이 어느 정도로 임상적 의도와 일치하는가?”
16.1 보건의료 데이터의 특성
우리가 확인했던 것이 사실 ACE inhibitor 사용과 혈관부종의 관계가 아니라, ACE inhibitor의 처방과 혈관부종 간의 관계일 수도 있다. 이전 15장에서 데이터의 질에 대해서 이미 다루었다. CDM으로 변환된 데이터의 질은 원본 데이터의 질을 결코 넘어설 수 없다. 여기서 우리는 대부분의 의료 서비스 사용 데이터의 특성에 대해 다룬다. OHDSI의 많은 데이터베이스는 청구 데이터 또는 전자의무기록에서 유래된다. 청구데이터와 전자의무기록 모두 연구를 위해서 만들어진 데이터베이스가 아니며, 둘은 서로 다른 데이터 수집 과정을 거친다. 청구 데이터는 보험금 청구 및 환급을 위하여 만들어진 데이터베이스로, 데이터 요소는 제공된 의료 서비스의 청구를 정당화하기 위한 목적하에 만들어진다. EHR의 데이터 요소는 임상 의료 행위 및 운영을 뒷받침하기 위하여 수집되며, 주어진 의료 시스템 기반에서 현재의 의료 서비스 및 추후 필요하리라 예상되는 정보 위주로 수집된다. 이들 모두 환자의 완전한 병력 medical history을 반영하거나 서로 다른 의료 기관 간의 데이터를 통합하지 못한다.
관찰 데이터로부터 믿을만한 근거를 생성하기 위해서 연구자는 환자가 의료 서비스를 찾는 그 순간부터 의료 서비스에 대한 데이터가 만들어져 분석에 사용되기까지의 전 과정에 대해 이해할 필요가 있다. 예를 들어 다양한 원천 관찰형 데이터에서 “약물 노출”은 의사의 처방 기록, 약국 조제 기록, 병원에서의 직접 주입, 또는 환자가 자가보고한 약물 복용 기록까지 여러 가지를 의미할 수 있다. 데이터가 어디에서 유래했는지는 환자가 약을 실제로 복용했는지 안 했는지 뿐 아니라 약물 복용 기간에 대한 신빙성에 영향을 미칠 수 있다. 데이터 수집 과정은 의사 처방 없이 복용할 수 있는 일반의약품 over-the-count(OTC) 등의 약물 노출을 과소평가할 수 있고, 환자가 처방만 받고 실제로 약국에서 조제 받지 않거나, 복용하지 않은 경우 약물 노출을 과대평가할 수도 있다. 치료 노출과 결과 간의 가능한 비뚤림을 이해하고, 보다 이상적으로는 이러한 오류를 정량화하고 보정하는 것은 가용한 데이터로부터 만들어 낸 근거의 신뢰성을 향상할 수 있다.
16.2 코호트 유효성 검사
Hripcsak and Albers (2017) 은 “표현형 phenotype은 유기체의 유전적 구성에서 파생된 유전형 genotype과 구별되는, 관찰할 수 있고 잠재적으로 변화하는 유기체의 상태를 나타내는 것”이라고 설명했다. 표현형이라는 용어는 전자 의무 기록(EHR) 데이터로부터 추정되는 환자 특성에 적용될 수 있다. 연구자는 정보학이 시작된 이후 구조화된 데이터와 서술적 데이터 모두에서 EHR phenotyping을 수행해 왔다. 목표는 원 EHR 데이터, 청구 데이터 또는 기타 임상 관련 데이터를 기반으로 대상 개념에 대한 결론을 도출하는 것이다. 표현형 알고리즘 (예를 들어, 표현형을 식별하거나 특성화하는 알고리즘)은 공학적인 최근의 연구 또는 다양한 형태의 머신 러닝을 통해 분야의 전문가나 분야 지식을 가지고 있는 엔지니어에 의해 생성될 수 있다.
이 설명은 임상적 타당성 고려 시 보강에 유용한 몇 가지 속성을 강조한다. 1) 그것은 관찰할 수 있는 어떤 것에 대해 말하고 있다는 것을 분명히 한다. (따라서 관찰형 데이터에서 수집이 가능함을 의미한다) 2) 그것은 표현형 정의에 시간의 개념을 포함한다. (사람의 상태가 변할 수 있기 때문에) 3) 원하는 의도인 표현형과 의도에 대한 구현인 표현형 알고리즘을 구별한다.
OHDSI는 “코호트”라는 용어를 채택하여 일정 기간 하나 이상의 포함 기준을 충족하는 사람 집합을 정의했다. “코호트 정의”는 관찰 데이터베이스에 대해 코호트를 구현하는데 필요한 논리를 나타낸다. 이와 관련하여 코호트 정의 (또는 표현형 알고리즘) 는 관찰 가능한 관심 임상 상태에 속하는 환자의 표현형을 반영하기 위한 코호트 생성에 사용된다.
임상적 특성 분석, 인구 수준 추정, 환자 수준 예측을 포함한 대부분의 관찰형 분석의 연구 프로세스의 일부로서 하나 이상의 코호트를 설정해야 한다. 이러한 분석에 의해 도출된 근거의 타당성을 평가하기 위해, 반드시 각각의 코호트에 대해 다음의 질문을 던져야 한다: ‘코호트 정의 따라 가용한 관찰 데이터에서 식별된 코호트의 피험자가 의도했던 표현형에 실제로 얼마나 부합하는가?’
다시 12장의 예제로 돌아가 보자. ’ACE 억제제가 thiazide 계열 이뇨제와 비교해서 혈관부종 위험성을 높이는가?’에 대한 질문에 대답하기 위하여 우리는 3가지 코호트를 생성했다. ACE 억제제를 처음 사용한 표적 코호트 target cohort, Thiazide 계열 이뇨제를 처음 사용한 대조 코호트 comparator cohort, 혈관부종이 발생한 결과 코호트 outcome cohort. ACE 억제제와 thiazide 이뇨제를 사용한 사람을 모두 식별하였다고 얼마나 자신 있게 말할 수 있는가? 처음 사용한 환자 new user라는 조건에 이전 (관찰되지 않은) 사용이 모두 배제되었다고 믿을 수 있는가? 약물에 노출되었다는 기록이 있는 환자가 실제로 약물에 노출되었고, 기록이 없는 환자는 실제로 약물에 노출되지 않았다고 믿을 수 있는가? ACE 억제제를 복용한다고 했을 때, 코호트 시작 시점과 종료 시점이 실제 약물을 시작하고, 종료한 시점과 일치할까? “혈관부종” 발생의 기록이 있는 환자가 다른 알레르기 피부 질환과 다른, 실제 피하조직의 부종을 경험했을 것인가? 실제 혈관 부종을 겪은 환자 중 얼마나 많은 숫자의 환자가 실제로 의사에게 진찰을 받고 해당 질환을 진단받고 기록되었을까? 우리가 약물에 의한 것으로 의심한 혈관부종이 음식 알레르기나 바이러스 감염 등에 의한 다른 원인과 얼마나 구별 지어질 수 있는가? 질병의 발생 시기가 약물 노출과 부작용 발생 간의 시계열적 연관성을 도출하기에 확신을 가질 수 있을 만큼 잘 포착되었는가? 이러한 유형의 질문에 답하는 것이 임상적 타당성의 핵심이다.
이 장에서는 코호트 정의를 검증하는 방법에 대해 논의한다. 먼저 코호트 정의의 타당성을 측정하는 데 사용되는 측정기준을 설명한다. 다음으로, 이러한 측정기준을 추정하는 두 가지 방법을 설명한다. 1) 원천 기록 검증을 통한 임상 판정과 2) PheValuator, 진단적 예측 모델링을 이용하는 반자동화 방법이다.
16.2.1 코호트 판단 측정기준
연구에 대한 코호트 정의가 결정되면 정의의 타당성을 평가할 수 있다. 타당성을 평가하는 일반적인 접근방식은 정의된 코호트의 일부 또는 모든 사람을 ’절대 표준 gold standard’과 비교하고 그 결과를 코호트 정의 내의 절대 표준 분류 및 자격 검정에 따라 계층화하여 2x2의 혼동 행렬 confusion matrix로 표현하는 것이다. 그림 16.1은 혼동 행렬의 요소를 보여준다.

Figure 16.1: 혼동 행렬.
코호트 정의의 참과 거짓 결과는 그 정의를 사람들의 집합에 적용함으로써 결정된다. 정의에 포함된 사람은 특정 건강 상태에 대해 양성으로 간주하며 “양성 true”으로 표시된다. 코호트 정의에 포함되지 않은 사람은 건강 상태에 대해 음성으로 간주하며 “음성 false”으로 표시된다. 코호트 정의에서 고려된 개인의 건강 상태에 대한 절대적 진리는 알기 어렵지만, 기준이 되는 절대 표준을 확립하는 방법은 여러 가지가 있는데, 그중 두 가지는 이번 장 후반부에 기술할 것이다. 사용한 방법과 관계없이, 이러한 사람에 대한 라벨링은 코호트 정의에 설명된 것과 동일하다.
표현형 지정의 이항 표시 오류 외에도, 건강 상태의 시점도 부정확할 수 있다. 예를 들어, 코호트 정의는 어떤 사람이 특정 표현형에 속한다고 올바르게 정의할 수 있지만, 언제부터 해당 건강 조건을 갖게 되었는지 시점을 정하는 데에는 부정확할 수 있다. 이 오류는 생존 분석 결과 (예를 들어 위험비 hazard ratio를 효과 측정으로 effect measure 이용하는 분석에서 비뚤림을 유발할 수 있다.
이 과정의 다음 단계는 절대 표준과 사용자가 정의한 코호트 정의 사이의 일치성을 평가하는 것이다. 절대 표준과 코호트 정의에 의해 “양성”이라고 표기된 사람을 “진양성 true positive”이라고 부른다. 절대 표준에 의해 “음성”으로, 코호트 정의에 의해 “양성”으로 분류된 사람을 “위양성 false positive”이라고 부른다. 예를 들어, 코호트 정의가 실제 특정 상태가 없는 환자를 잘못하여 특정 상태가 있다고 판단할 수 있다. 절대 표준과 코호트 정의에 의해 모두 “음성”으로 정의된 환자는 “진음성 true negative”이라고 부른다. 절대 표준에 의해 “양성”으로 분류되었으나 코호트 정의에 의해 “음성”으로 분류된 환자는 “위음성 false negative”이라고 부른다. 예를 들어 실제로는 환자가 특정 건강 상태를 지니고 있으나, 코호트 정의에서는 그렇지 않게 분류될 수 있다. 혼동 행렬의 네 칸의 숫자를 세어, 우리는 코호트 정의가 사람을 실제 표현형으로 분류하는 데 얼마나 정확한지 여부를 정량화할 수 있다. 다음은 코호트 정의 성능을 측정하기 위한 표준 성능 평가 기준이다 (역자 주: 민감도, 특이도는 절대 표준을 분모로 하는 성능평가도구이고, 양성예측도와 음성예측도는 측정도구의 평가를 분모로 하는 성능평가도구이다).
코호트 정의 민감도 sensitivity – 실제 표현형에 속하는 피험자 중 얼마나 많은 비율의 피험자가 코호트 정의에 의해 양성으로 분류되는가? 다음의 공식으로 구한다:
민감도 = 진양성 / (진양성 + 위음성)
코호트 정의 특이도 specificity – 실제 표현형에 속하지 않는 피험자 중 얼마나 많은 비율의 피험자가 코호트 정의에 의해 음성으로 분류되는가? 다음의 공식으로 구한다:
특이도 = 진음성 / (진음성 + 위양성)
코호트 정의 양성 예측도 Positive predictive value(PPV) – 코호트 정의에 의해 양성으로 분류되는 환자 중 표현형을 실제로 가지고 있는 환자가 얼마나 되는가? 다음의 공식으로 구한다:
양성 예측도 = 진양성 / (진양성 + 위양성)
코호트 정의 음성 예측도 Negative predictive value(NPV) – 코호트 정의에 의해 음성으로 분류되는 환자 중 표현형을 실제로 가지고 있지 않은 환자가 얼마나 되는가? 다음의 공식으로 구한다:
음성예측도 = 진음성 / (진음성 + 위음성)
위의 측정기준에서 만점은 100%이다. 관측 데이터의 특성상 만점은 보통 평균과 거리가 멀다. Rubbo et al. (2015) 은 심근경색에 대한 코호트 정의를 검증하는 연구를 검토했다. 그들이 조사한 33개의 연구 중, 오직 하나의 데이터 집합에서 하나의 코호트 정의만이 양성 예측도에 대해 만점을 얻었다. 전체적으로 33개 연구 중 31개에서 70% 이상의 양성 예측도가 보고되었다. 그러나 그들은 또한 33개 연구 중 11개만이 민감도를 보고했고 5개만이 특이도를 보고했다는 것을 발견했다. 양성예측도는 민감성, 특이성, 유병률의 함수다. 유병률에 대한 값이 다른 데이터에서는 민감도와 특이도가 일정하게 유지되더라도 양성예측도에 대해서는 다른 값을 생성한다 (역자 주: 같은 검사법이라 할지라도 질병의 유병률이 바뀌면 PPV, NPV가 달라진다). 민감도와 특이도가 없다면 불완전한 코호트 정의로 인한 비뚤림을 수정할 수 없다. 게다가, 건강 상태의 오분류가 수행될 수 있는데, 즉 표적 및 대조 코호트 정의 수행 시 오분류의 정도가 비슷할 수도 있지만, 때로는 그 정도가 두 그룹 간에 매우 다를 수도 있다는 점이다. 이전의 코호트 정의 검증 연구는 실제 추정치에 강한 비뚤림을 초래할 수 있음에도 불구하고 대상 및 대조 코호트 간의 오분류의 잠재적 가능성에 대해 시험을 하지 않았다.
코호트 정의에 대한 성능 평가 기준이 마련되면, 이러한 정의를 사용하여 연구 결과를 조정하는 데 사용될 수 있다. 이론적으로, 이러한 측정 오차 추정치를 이용해 연구 결과를 보정하는 방법은 잘 확립되어 있다. 그러나 실제로는 성능 평가 점수를 얻기 어렵기 때문에, 이러한 보정은 거의 고려되지 않고 있다. 절대 표준을 결정하는 방법은 이 절의 나머지 부분에 설명되어 있다.
16.3 원천 기록 검증
코호트 정의를 검증하는 데 사용되는 일반적인 방법은 원천 기록 확인을 통한 임상적 판단이다. 즉 관심 임상 조건이나 특성을 분류할 수 있는 충분한 지식을 갖춘 하나 분야 이상의 전문가가 개인의 기록을 철저히 검사하는 것이다. 차트 검토는 보통 다음의 단계를 따른다:
- 기관생명윤리위원회 institutional review board(IRB) 또는 환자 개인에게 직접 차트 검토 및 연구에 대한 승인을 받는다.
- 평가할 코호트 정의를 사용하여 코호트를 생성한다. 수동으로 전체 코호트를 판단할 만큼 시간적, 인적 자원이 충분하지 않은 경우 검토 대상 중 일부를 표본으로 추출한다.
- 환자 차트를 검토할 수 있는 충분한 임상 전문 지식을 가진 사람 한 명 이상을 섭외한다.
- 환자가 원하는 임상 조건이나 특성이 있는지에 대해 양성 및 음성을 판단하기 위한 지침을 결정한다.
- 임상 전문가는 각 환자가 표현형에 속하는지 여부를 분류하기 위해 표본 내의 사람에 대한 모든 가용 데이터를 검토하고 판단한다.
- 코호트 정의 분류 및 임상 판정 분류에 따라 혼동 행렬로 도표화하고 수집된 데이터에서 가능한 성능을 평가한다.
차트 검토의 결과는 일반적으로 하나의 성능 지표인 양성예측도 평가로 제한된다. 평가 대상이 되는 코호트 정의는 원하는 조건이나 특성을 가진 것으로 생각하는 사람만 생성하기 때문이다. 따라서 코호트의 표본에 있는 각 개인은 임상적 판단에 근거하여 진양성 또는 위양성 중 하나로 분류된다. 전체 모집단의 표현형 (코호트 정의에 의해 식별되지 않은 사람을 포함)에 있는 모든 사람에 대한 지식이 없으면, 위음성의 식별이 불가능하며, 따라서 혼동행렬의 나머지 부분을 채워 나머지 성능 특성을 채울 수 없다. 모집단 전체의 표현형을 식별하는 가능한 방법에는 모집단이 작지 않은 경우 불가능한 전체 데이터에 대한 차트 검토 또는 이미 충분한 임상 정보와 특정 표현형 유무가 결정된 임상 레지스트리 (예로 암 레지스트리)의 활용 등이 포함되나 이는 일반적으로는 실행이 불가능하다 (아래 예시 참조). 또는 코호트 정의에 적합하지 않은 사람을 표본으로 추출하여 예측된 음성 집단의 표본을 생성한 다음, 위 차트 검토의 3~6단계를 반복하여 이러한 환자가 진정으로 관심의 임상 조건이나 특성이 없었는지 여부를 확인할 수 있다. 이렇게 하면 음성예측도를 추정할 수 있으며, 표현형의 유병률에 대한 적절한 추정치를 구할 수 있다면, 민감도와 특이도를 추정할 수 있다.
원천 기록 확인, 차트 검토를 통한 임상적 판단에는 여러 가지 한계가 있다. 앞서 언급했듯이, 차트 검토는 양성예측도와 같은 단일 지표의 평가에도 매우 많은 시간이 소요되고 자원이 많이 소요되는 과정이 될 수 있다. 이러한 한계는 혼동 행렬을 완전히 채우기 위해서 전체 모집단을 평가해야 하므로 실용성을 크게 저해한다. 또한 상기 프로세스의 다중 단계는 연구의 결과를 편향시킬 수 있는 잠재력을 가지고 있다. 예를 들어, EHR에서 기록에 동일하게 접근할 수 없거나, EHR이 없거나, 개별적인 환자 동의가 필요한 경우, 추출된 평가 대상의 표본은 정말로 무작위적이지 않을 수 있으며, 선택비뚤림을 야기할 수 있다. 또한 수동적인 프로세스는 인간의 실수나 잘못된 분류에 취약하므로 완벽하게 정확한 측정 기준을 나타내지 못할 수 있다. 환자의 기록이 불충분하거나, 결정이 주관적이거나, 충분한 전문성이 없는 등의 이유로 종종 임상 전문가들 사이에 의견 불일치가 있을 수 있다. 많은 연구에서, 전문가들 사이의 불일치에 대해 충분히 고려하지 않는 다수결의 원칙을 통해 일방적으로 해결된다.
16.3.1 원천 기록 검증에 대한 예시
차트 검토를 활용한 코호트 정의 유효성을 검사하는 과정의 예시는 Columbia University Irving Medical Center(CUIMC)에 수행하는 연구로부터 제공되는데, 그 연구는 National Cancer Institute(NCI)에서 행하는 타당성 연구의 일환인 다수의 암에 대한 코호트 정의를 검수하는 것이다. 이 과정은 이러한 암 중 하나인 전립선암을 검증하는 데 사용되었는데, 과정은 다음과 같다.
- 제안서를 제출하고, OHDSI 암 표현형 연구를 위한 IRB의 동의를 얻었다.
- 전립선암에 대한 코호트 정의를 개발하였다: 어휘를 탐구하기 위해 ATHENA와 ATLAS를 사용해서, 전립선 2차 신경세포 (concept ID 4314337) 또는 Non-Hodgkin’s의 전립선 림프종 (concept ID 4048666) 을 제외한 전립선 악성 종양 (concept ID 4163261) 을 가지고 있는 모든 환자를 포함한 코호트 정의를 생성했다.
- ATLAS를 사용하여 코호트를 생성하고, 수동 검토를 위한 100명의 임의 환자를 지정하여 개개인의 PERSON_ID를 환자 MRN을 매핑 테이블을 사용하여 매핑하였다. PPV의 성능 측정 기준에 맞는 원하는 통계적 정밀도를 달성하기 위하여 100명의 환자가 선정되었다.
- 위의 임의로 선정한 각각의 환자가 참 혹은 거짓 양성 반응을 보이는지 밝히기 위하여 입원 환자와 외래 환자를 모두 포함한 다양한 EHR의 기록을 수동으로 검토하였다.
- 한 의사가 수동 검토와 임상 판정을 실행하였다. (비록 이상적으로는 미래에 합의와 계층 간의 신뢰성을 평가하기 위해 더욱 엄격한 검증 연구가 더욱더 많은 검토자에 의해 실행되어야 할 것이다)
- 참고 기준의 결정은 임상 문서, 병리학 보고서, 실험실, 의약품, 사용 가능한 전자 환자기록 전체 문서가 기반이 되었다.
- 환자들은 1) 전립선암 2) 비 전립선암 3) 인식 불가로 분류되었다.
- PPV의 보수적인 추정치는 다음과 같이 계산되었다: 전립선암 / (비 전립선암 + 인식 불가)
- 그리고, CUIMC 전체 인구를 아우르는 참조 기준을 식별하기 위한 절대 표준과 같은 종양 레지스트리를 사용하여, 코호트 정의에 정확히 식별되지 않은 종양 레지스트리 안의 환자 수를 세었다. 이 방법은 우리가 이 값을 진양성 혹은 위음성으로 사용하여 민감도를 측정할 수 있게 했다.
- 측정된 민감도, PPV, 유병률을 사용하여, 코호트 정의에 대한 특이도를 측정할 수 있었다. 위에서 언급한 바와 같이, 이 과정은 각각의 코호트 정의가 수동 차트 검토를 통하여 개별적으로 평가되어야 하고, 또한 모든 성능 지표를 확인하기 위해 CUIMC 종양 레지스트리와 연관되어야 하므로 상당한 시간과 노동력이 필요하다. 최대한 신속히 종양 레지스트리의 접근 권한을 얻는 검토를 이행하지만, IRB 승인 과정 만으로 몇 주가 소요되고, 수동 차트 검토의 과정 자체로도 몇 주가 더 소요된다.
Rubbo et al. (2015) 는 심근경색(MI) 코호트 정의에 대한 검증 노력의 검토는 연구에 사용된 코호트 정의뿐만이 아니라 검증 방법과 보고된 결론에서도 상당한 이질성이 존재한다는 것을 확인하였다. 그들은 급성 심근경색에 대한 절대 표준의 코호트 정의가 존재하지 않다고 결론 지었다. 그들은 이 과정이 상당한 비용과 시간이 소모되었다고 언급하였다. 이러한 제약 때문에, 대부분의 연구는 그들의 검증에 대한 작은 표본 크기를 가졌고 이는 곧 성능 특성에 대한 추정치의 큰 변화를 가져왔다. 그들은 또한 33개의 연구 중에서, 모든 연구가 양성 예측도에 대해 보고하는 가운데 오직 11개의 연구가 민감도에 대해 보고하였고, 오직 5개의 연구만이 특이도에 대해 보고하였다. 위에서 언급한 바와 같이 민감도와 특이도의 예측 없이, 분류비뚤림에 대한 통계적 교정은 시행될 수 없다.
16.4 PheValuator
OHDSI 공동체는 진단 예측 모델을 사용하여 절대 표준을 구축하는 다른 접근방식을 개발하였다. (Swerdel, Hripcsak, and Ryan 2019) 일반적인 생각은 outcome의 확인을 의료진이 원천 기록 검증을 수행하는 방식과 비슷하게 모방하지만, 규모에 맞게 자동화된 방법으로 시행된다. 이 도구는 PheValuator라고 불리는 오픈 소스 R 패키지로 개발되었다.54 PheValuator은 환자 수준 예측(PLP) 기능을 사용한다.
과정은 다음과 같다:
- 극도로 특이도가 높은(“xSpec”) 코호트를 생성한다: 진단 예측 모델을 학습할 때 관심 결과가 생길 가능성이 아주 높은 사람으로서 noisy positive label로 사용될 집단을 결정한다.
- 극도로 민감도가 높은(“xSens”) 코호트를 생성한다: 결과가 생길 가능성이 있는 모든 이를 포함하는 집단을 결정한다. 이 코호트는 그 집단의 상반되는 집단을 식별하는데 사용될 것이다. 즉, 이는 진단 예측 모델을 학습할 때 noisy negative labels로 사용될 결과를 갖지 않을 것이라고 확신하는 사람 집단이다.
- xSpec과 xSens 코호트를 사용하여 예측 모델을 학습시킨다: 13장에서 설명된 바와 같이, 다양한 환자 특징을 예측자로 사용하여 모델을 적합시키고, 어떤 개인이 xSpec 코호트에 포함되는지 (결과가 있다고 생각되는 사람들) 혹은 xSens 코호트에 상반되는 집단 (결과가 없다고 생각되는 사람들)에 포함되어 있는지에 대한 여부를 예측하는 것을 목표로 한다.
- 코호트 정의 성능을 평가하는데 사용될 독립적인 hold-out 집단을 이용하여 outcome 발생의 확률을 예측하는데 적용한다: 모델의 예측 인자는 개인이 표현형에 포함되리라 예측된 확률을 추정하기 위해서 적용될 수 있다. 우리는 이 예측치를 확률적 절대 표준 probabilistic gold standard로 사용한다.
- 코호트 정의의 성능 특성을 평가하라: 우리는 예측된 확률을 사용자 코호트 정의의 이진 분류와 비교한다 (혼동 행렬의 시험 조건). 시험 조건과 참 조건의 예측을 사용하여, 우리는 혼동 행렬을 완전히 채울 수 있고 성능 특성, 다시 말하면 민감도, 특이도, 예측값의 전부를 계산할 수 있다. 이 접근 방식을 사용하는 데 있어 주요 제약은 outcome 발생이 데이터베이스 내 일부 데이터에 의해 제약을 받는 사람의 확률을 예측하는 것이다. 임상 노트 clinical note 에만 outcome 발생을 확인할 수 있는 주요 인자가 존재하는 경우, 임상 노트 사용이 불가능한 데이터의 경우 이와 같은 방식이 유효하지 않을 수 있다.
진단 예측 모델링에서, 병이 있는 사람과 없는 사람을 식별하는 모델을 생성한다. 환자 수준 예측 장에서 언급된 바와 같이, 예측 모델은 표적 target 코호트와 결과 outcome 코호트를 사용하여 개발되었다. 표적 코호트는 outcome를 소유한 개인과 그렇지 않은 개인 모두를 포함한다; 결과 코호트는 표적 코호트 안의 개인 중에서 outcome가 생긴 개인을 식별한다. PheValuator 과정에서 우리는 극도의 명확한 코호트 정의, 즉 “xSpec” 코호트를 예측 모델을 위한 결과 코호트를 결정하는 데 사용한다. xSpec 코호트는 관심 질병에 걸릴 확률이 매우 높은 (특이도가 높은) 집단을 찾기 위한 정의를 사용한다. xSpec 코호트는 관심 outcome에 대해 발병 기록이 여러 번 발생한 환자들로 정의할 수 있다. 심방세동을 예로 들자면 심방세동 진단 코드를 10번 이상 가진 사람으로 선정할 수 있다. 심근 경색에 대해서는 5번의 심근경색이 생겼고 그 중 최소 2번은 입원 중에 생긴 것으로 정의할 수 있다. 예측 모델에 사용할 표적 코호트는 outcome이 있을 가능성이 낮은 집단의 그룹과 xSpec 코호트 안의 집단을 합쳐서 구축한다. 실제로는 outcome이 발생 했을 가능성이 낮은 집단을 결정하기 위해서, 전체 데이터베이스에서 표본을 추출하여 outcome 표현형에 속하리라 생각되는 환자를 제외하는데, 보통 xSpec 코호트를 정의하는 데 사용된 concept을 포함하고 있는 기록을 가진 개인을 제외함으로써 얻을 수 있다. 이 방법에는 제약이 있다. 이 xSpec 코호트 내 사람들이 병을 가진 이와 다른 특성을 가질 가능성이 있기 때문이다. xSpec 에 포함된 환자들은 첫 진단 후 보통의 환자와 비교해 관찰 시간이 더욱 길었을 수도 있다. 우리는 확률적 절대 표준을 생성하는데 사용되는 예측 모델을 생성하는데 LASSO 로지스틱 회귀를 사용한다. (Suchard et al. 2013) 이 알고리즘은 간명한 모델을 생성하고 일반적으로 데이터 집합 전체에서 존재할 수 있는 많은 공변량을 제거한다. 현재 PheValuator 소프트웨어 버전에서, 결과 상태 (예/아니오) 는 개인에 대한 모든 데이터에 근거하여 평가되고 (모든 관측 시간), 코호트 시작 날짜의 정확도를 평가하지 않는다.
16.4.1 PheValuator에 의한 검증 예시
PheValuator를 이용해 코호트 정의의 성능 특성을 완전히 평가할 수 있는데, 예시로 심근 경색 코호트를 검증해 보도록 한다.
다음은 PheValuator를 사용하여 심근경색 코호트 정의를 검토하는 과정이다:
첫 번째 단계 : xSpec 코호트 정의
높은 확률 (높은 특이도)로 심근경색을 가지고 있을 환자를 정의하자 (xSpec). 심근경색을 뜻하는 concept 또는 그것의 하위 concept이 입원 5일 이내에 발생하거나, 1년 이내 4번 이상 condition occurrence 테이블에 기록된 환자가 필요하다. 그림 16.2가 ATLAS에서 해당 코호트의 정의가 어떻게 표현되는지 보여준다.
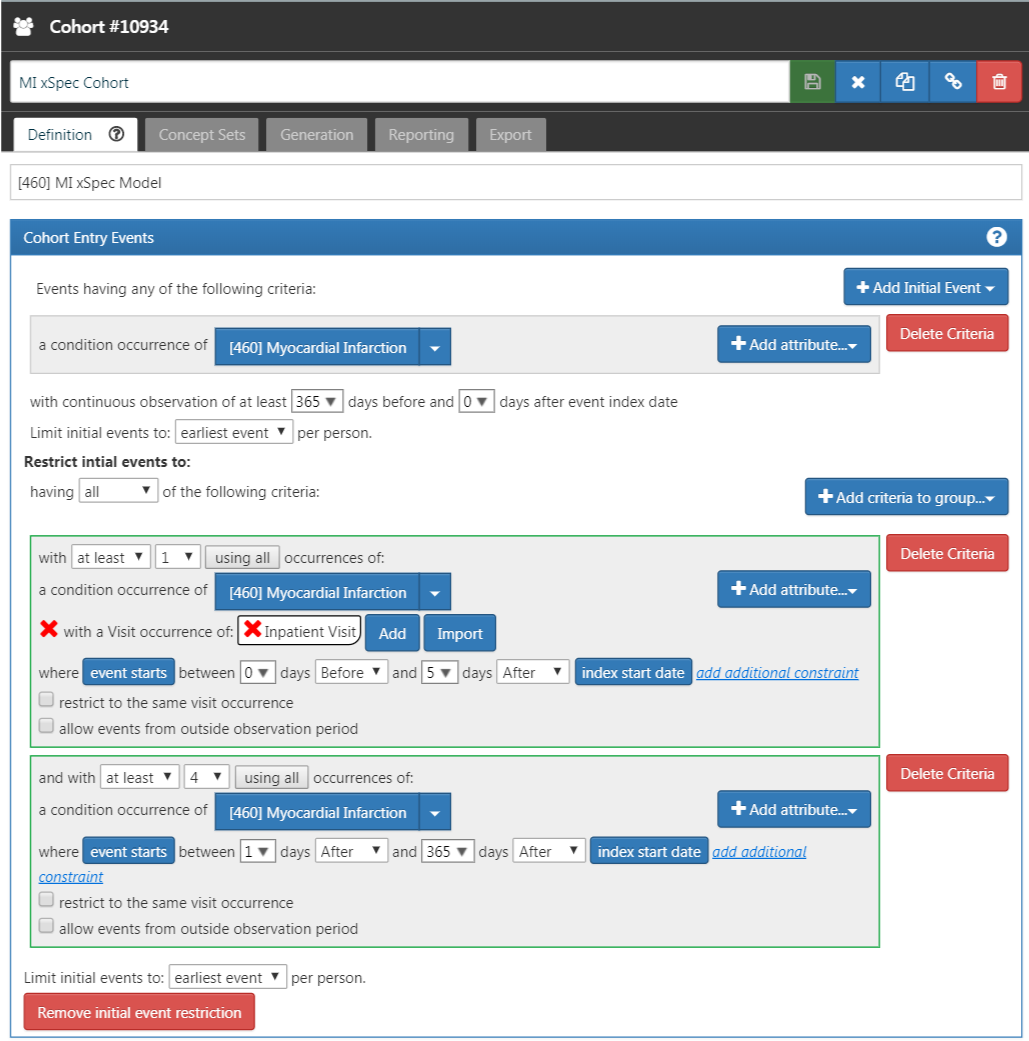
Figure 16.2: 극도로 특이도가 높은(xSpec) 심근경색 코호트 정의
두 번째 단계 : xSens 코호트 정의
그리고 극도로 민감도가 높은 코호트 (xSens)를 정의하자. 이 코호트는 한 번이라도 심근경색이라는 concept에 해당하는 CONDITION OCCURRENCE 기록을 가지고 있는 환자로 정의할 수 있다. 그림 16.3은 심근경색의 xSens 코호트를 ATLAS에서 어떻게 만드는지 보여준다.
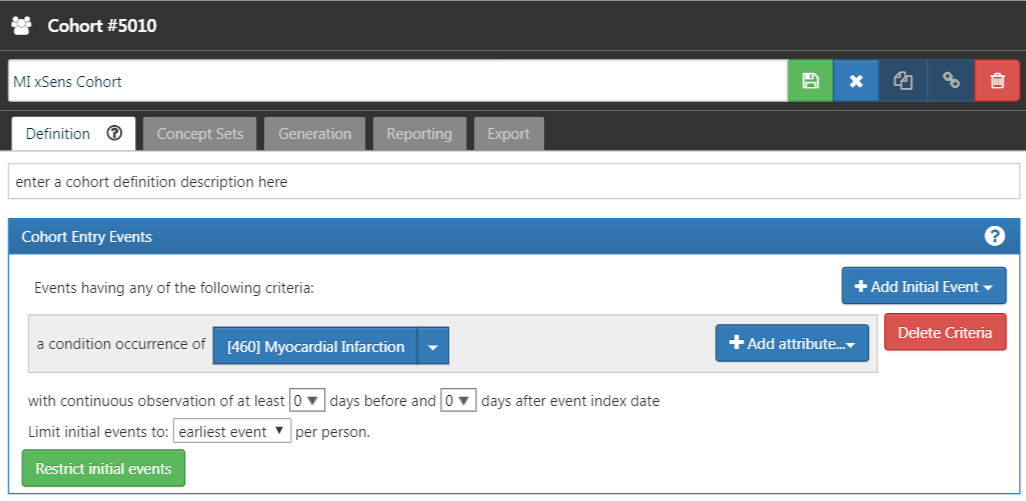
Figure 16.3: 극도로 민감도가 높은 (xSens) 심근경색 코호트 정의
세 번째 단계 : 예측 모델에 적합
‘createPhenoModel’ 함수는 평가용 코호트 환자가 우리가 관심 있는 임상 결과를 실제로 가지고 있을 확률을 평가하기 위한 진단 예측 모델을 개발한다. 이 기능을 사용하기 위해 1단계와 2단계에서 개발한 xSpec 및 xSens 코호트를 사용한다. xSpec 코호트는 함수에서 xSpecCohort 매개변수로 입력된다. xSens 코호트는 xSens 코호트에 있는 코호트 모델링 프로세스 중에 사용될 대상 코호트에서 제외되어야 하기 때문에 ‘exclCohort’ 매개변수로 입력된다. 이 배제 방법을 사용하여 실제로 임상 결과가 있을 가능성이 낮은 사람을 결정할 수 있다. 우리는 이 그룹을 “불확실 음성 noisy negative” 그룹, 즉 해당 임상결과가 있는 사람을 소수 포함하여 대부분은 해당 outcome이 없을 가능성이 큰 사람이 포함된 그룹으로 생각할 수 있다. 함수에서 xSens 코호트를 ‘prevCohort’ 매개변수로 사용할 수도 있다. 이 매개변수는 해당 임상 결과에 대한 모집단에서의 대략적인 유병률을 결정하기 위해서 사용된다. 일반적으로 데이터베이스에서 무작위로 추출한 표본에서의 유병률은 모수의 유병률과 비례해야 한다. 설명한 방법을 사용하면, 더 이상 임의 추출 표본을 사용하지 않고, outcome 발생 환자 대비 outcome 발생하지 않은 환자의 비율을 기반으로 예측 모형의 재교정 re-calibration이 필요하다.
xSpec 코호트를 정의하는 데 사용된 모든 concept은 모델링 과정에서 제외해야 한다. 이를 위해 excludedConcepts 매개변수를 xSpec 정의에 사용된 concept 목록으로 설정한다. 예를 들어 심근경색의 경우 심근 경색에 대한 concept과 모든 하위 concept을 사용하여 ATLAS에 concept set을 만들었다. 예제에서는 excludedConcepts 매개변수를 심근 경색의 concept ID인 4329847로 설정하고 addDescendantsToExclude 매개변수를 TRUE로 설정해서, 모든 하위 컨셉이 제외되도록 하였다.
모델링에 포함될 환자의 임상 특성을 지정할 수 있는 몇 가지 매개변수가 있다. lowerAgeLimit 및 upAgeLimit 을 이용하여 모델링에 포함될 환자의 연령의 하한과 상한을 설정할 수 있다. 계획한 연구가 특정 연령군에 대한 연구일 경우 이를 이용할 수 있다. 예를 들어, 연구에 사용될 코호트 정의가 소아의 제1형 당뇨병에 대한 것이라면, 진단 예측 모델을 개발하는 데 사용되는 연령을 5~17 세로 제한할 수 있다. 이를 통해 테스트할 코호트 정의에서 선택한 환자와 더 밀접하게 관련 있는 모델을 생성할 수 있다. 또한 gender 매개변수를 남성 또는 여성의 concept ID로 설정하여 모델에 포함할 성별을 지정할 수 있다. 기본적으로 이 매개변수는 남성과 여성 모두를 포함하도록 설정되어 있다. 이 기능은 전립선암과 같은 특정 성별에만 발생하는 질병에 유용 할 수 있다. 환자별 의료기관 최초 방문 일자를 기준으로 startDate및 endDate 매개변수를 각각 날짜 범위의 하한 및 상한으로 설정하여 대상 환자에 대한 제한을 설정할 수 있다. 마지막으로 mainPopnCohort 매개변수를 사용하여 대상 및 결과 코호트에 있는 모든 사람을 선택할 대규모 집단 코호트를 지정할 수 있다. 대부분의 경우 이 값은 0으로 설정되어 대상 및 결과 코호트에 대한 사람을 선택하는 데 제한이 없음을 나타낸다. 그러나 때에 따라 outcome의 유병률이 매우 낮거나 0.01% 이하인 경우, 이 매개변수가 더 나은 모델을 구축하는 데 유용한 경우가 있을 수 있다. 예를 들면
setwd("c:/temp")
library(PheValuator)
connectionDetails <- createConnectionDetails(
dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
phenoTest <- createPhenoModel(
connectionDetails = connectionDetails,
xSpecCohort = 10934,
cdmDatabaseSchema = "my_cdm_data",
cohortDatabaseSchema = "my_results",
cohortDatabaseTable = "cohort",
outDatabaseSchema = "scratch.dbo", #should have write access
trainOutFile = "5XMI_train",
exclCohort = 1770120, #the xSens cohort
prevCohort = 1770119, #the cohort for prevalence determination
modelAnalysisId = "20181206V1",
excludedConcepts = c(312327, 314666),
addDescendantsToExclude = TRUE,
cdmShortName = "myCDM",
mainPopnCohort = 0, #use the entire person population
lowerAgeLimit = 18,
upperAgeLimit = 90,
gender = c(8507, 8532),
startDate = "20100101",
endDate = "20171231")이 예제에서, 코호트 테이블(“cohort”)과 그것이 존재하는 데이터베이스(“my_results”)를 지정하였고 (cohortDatabaseSchema, cohortDatabaseTable - “my_results.cohort”), CONDITION_OCCURRENCE, DRUG_EXPOSURE 등을 찾을 수 있는 CDM 데이터베이스의 위치를 설정했다 (cdmDatabaseSchema - “my_cdm_data”). CDM에서 2010년 1월 1일부터 2017년 12월 31일 사이에 첫 번째 의료기관 방문이 있는 환자만이 모델에 포함되었다. xSpec 코호트 생성 시 ‘312327’, ‘314666’ 및 그들의 하위 컨셉 등이 사용되지 않도록 하였다. 첫 번째 방문 시 환자의 나이는 18세에서 90 사이여야 한다.
네 번째 단계 : 평가용 코호트 생성
createEvalCohort 기능은 관심 outcome의 예측된 가능성을 가진 각각의 사람들의 대규모 코호트를 생성하기 위하여 PatientLevelPrediction 패키지 기능 applyModel를 사용한다. 이 기능은 xSpec 코호트를 지정해야 한다 (매개변수 xSpecCohort를 xSpec 코호트 ID로 지정함으로써). 우리는 앞 과정에서 진행했었던 바와 같이 평가 코호트에 포함된 사람들의 특징도 지정해야 할 수도 있다. 이것은 나이 제한의 상한과 하한선을 지정하는 것도 포함할 수 있다 (나이를 각각 lowerAgeLimit와 upperAgeLimit 전달 인자로 설정함으로써), 성별 (매개변수 gender를 concept ID의 남성 그리고/혹은 여성으로 지정함으로써), 시작과 종료 날짜 (날짜를 각각 startDate와 endDate 전달인자로 설정함으로써), 그리고 사용할 인구의 코호트 ID를 mainPopnCohort로 지정하여 사람을 선별할 대규모 인구를 설계한다.
예를 들면:
setwd("c:/temp")
connectionDetails <- createConnectionDetails(
dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
evalCohort <- createEvalCohort(
connectionDetails = connectionDetails,
xSpecCohort = 10934,
cdmDatabaseSchema = "my_cdm_data",
cohortDatabaseSchema = "my_results",
cohortDatabaseTable = "cohort",
outDatabaseSchema = "scratch.dbo",
testOutFile = "5XMI_eval",
trainOutFile = "5XMI_train",
modelAnalysisId = "20181206V1",
evalAnalysisId = "20181206V1",
cdmShortName = "myCDM",
mainPopnCohort = 0,
lowerAgeLimit = 18,
upperAgeLimit = 90,
gender = c(8507, 8532),
startDate = "20100101",
endDate = "20171231")이 예시에서, 매개변수는 평가 코호트 파일을 생성하기 위하여 (“…5XMI_eval…rds”) 이 모델 파일을 사용해야 한다고 명시한다 (“…5XMI_train…rds”). 이 과정에서 생성된 모델과 평가 코호트 파일은 다음 과정에서 제공될 코호트 정의의 평가에 사용될 것이다.
다섯 번째 단계 : 코호트 정의 생성 및 검증
다음 과정은 평가받을 수 있게 코호트 정의를 생성하고 테스트하는 것이다. 이상적인 성능 특성은 관심 연구 문제를 설명하기 위하여 코호트의 의도된 사용에 따라 달라질 수 있다. 몇몇 특정한 문제에는, 굉장히 민감한 알고리즘이 필요할 수 있다; 다른 것은 더욱 구체적인 알고리즘이 필요할 수 있다. PheValuator를 사용한 코호트 정의의 성능 특성을 결정하는 과정은 그림 16.4가 보여준다.
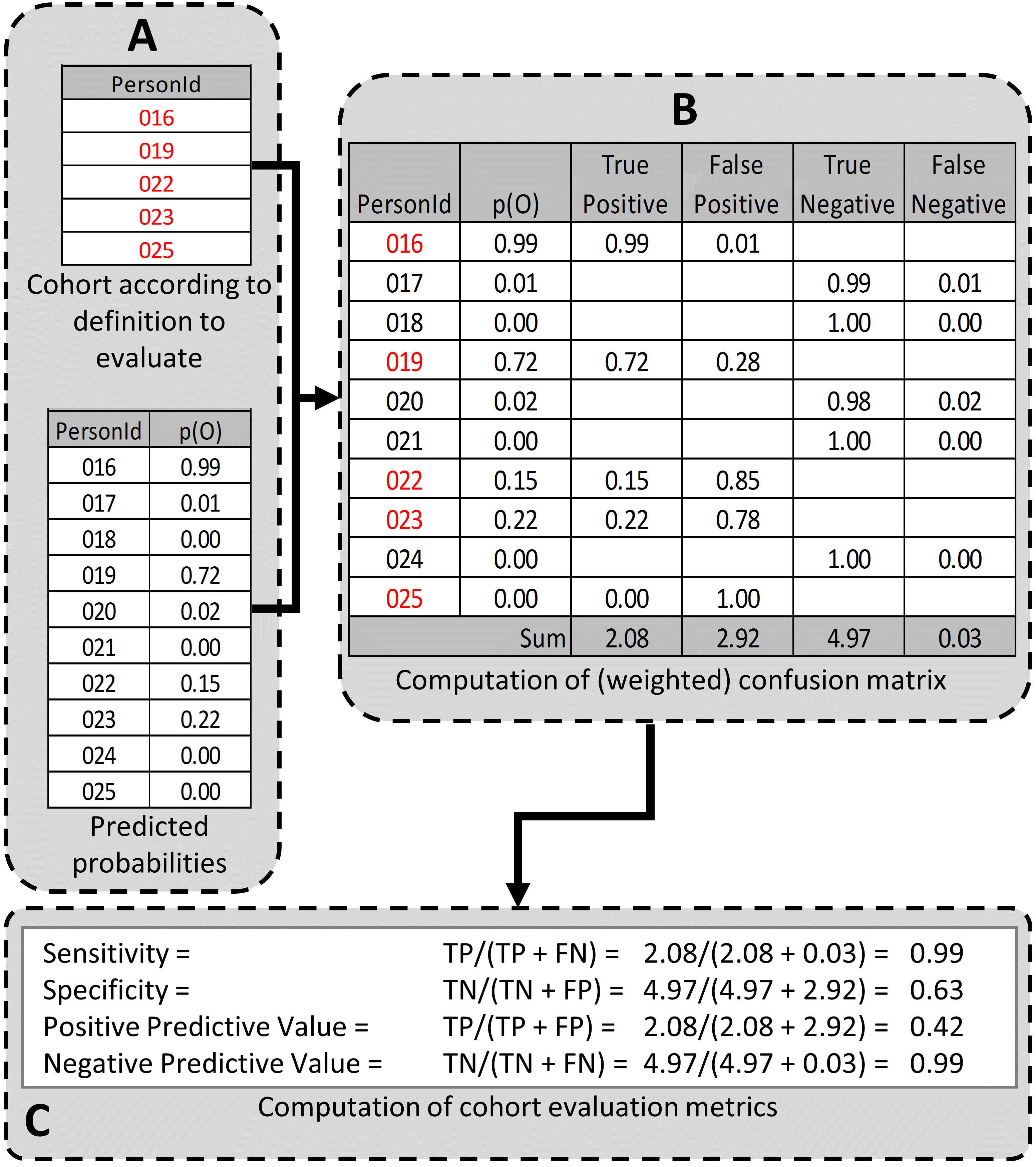
Figure 16.4: Determining the Performance Characteristics of a cohort definition using PheValuator. p(O) = Probability of outcome; TP = True Positive; FN = False Negative; TN = True Negative; FP = False Positive.
그림 16.4의 A 부분에서, 테스트가 진행될 코호트 정의에서 사람들을 조사하고 코호트 정의에 포함되었던 사람 (Person ID가 016, 019, 022, 023, 025), 포함되지 않았던 사람 (Person Id가 017, 018, 020, 021, 024)의 평가 코호트 (앞 과정에서 생성되었었다)로부터 사람들을 찾았다. 이전에 이미 만든 예측 모델 (p(O))을 사용하여 각각 포함된/포함되지 않은 사람들의 outcome 발생 가능성을 예측했었다.
다음과 같이 진양성, 진음성, 위양성, 위음성 결과를 추정하였다. (그림 16.4의 B 부분 참조):
사용자가 정의한 코호트 중 한 환자가 평가용 코호트에 포함된다면, 즉 사용자 정의 코호트가 어떤 사람을 ’양성’으로 간주하는 확률, 즉 양성에 대한 기대값이 진양성에 추가되며, 1에서 해당 확률을 뺀, 음성에 대한 확률이 기대값으로 진음성에 추가된다. 예를 들어 Person ID 016인 사람이 해당 임상 결과 outcome를 가질 (양성일) 확률이 99%라면, 기대값으로 0.99가 진양성에 더해지고, 기대값으로 0.01(1.00-0.99)이 위양성에 더해진다. 이러한 과정이 사용자가 정의한 코호트의 모든 사람 (예를 들어 Person ID가 019, 022, 023, 025인 사람)에 대해 적용된다.
마찬가지로, 평가 코호트에 존재하는 환자가 사용자 정의 코호트에 존재하지 않을 경우, 즉 사용자 정의 코호트가 음성으로 예측한 사람의 경우, 1에서 표현형에 대한 예측 확률에서 뺀 값이 음성에 대한 기대값으로 진음성에 추가되고, 표현형에 대한 예측 확률이 기대값으로 진양성에 추가된다. 예를 들어 Person ID가 017인 사람이 outcome을 가질 확률이 1%라고 예측했다면 (결과가 없을 확률이 99%라고 예측했다면), 1.00 – 0.01 = 0.99 가 진음성에 추가되고 0.01이 위음성에 추가된다. 이런 과정이 사용자 정의 코호트에 포함되지 않은 평가용 코호트의 환자 모두 (예를 들어 Person ID 018, 020, 021, 024인 사람)에게 반복된다.
평가용 코호트의 전체 집합에 대해 이러한 값을 추가한 후, 혼동 행렬의 4개 칸을 모두 추정값으로 채울 수 있다. 그리고 민감도, 특이도, 양성예측도와 같이 PA 성능의 추정치 point estimate를 만들 수 있다 (16.4 C). 추정치만을 예측할 수 있고, 추정치의 분산을 측정할 수는 없다는 것에 주의하자. 이 예제에서는 민감도, 특이도, 양성 예측도, 음성 예측도는 각각 0.99, 0.63, 0.42, 0.99로 나타났다.
testPhenotype 함수를 이용하여 사용자 정의 코호트의 성능을 측정할 수 있다. 이 함수는 우리가 모델과 평가용 코호트를 만들었던 이전 두 단계의 결과값을 사용한다. createPhenoModel 함수의 결과상 나오는 RDS file에 대한 modelFilName 을 입력한다. 사용자 정의 코호트의 성능을 테스트하기 위해, cohortPheno의 코호트 ID를 해당 코호트의 ID로 입력한다. phenText 인자에는 ’MI Occurrence, Hospital In-Patient Setting’과 같이 해당 코호트 정의에 대한 설명을 입력한다. testText에는 “5 X MI”와 같이 xSpec 정의에 대한 설명을 입력한다. 이 단계의 출력물은 코호트 정의의 성능의 추정치를 data frame 형태이다. cutPoints 인자에는 성능 추정치에 대한 참고치 값을 list 형태로 입력한다. 코호트 정의 성능은 @ref(fig:phevaluatorDiagram 그림에서 본 것과 같이 “기대값 expected value”으로 계산된다. 예측값 기반으로 성능을 추정하기 위하여, 우리는 cutPoints 인자에 “EV(expected value)”를 포함한 목록 list을 입력한다. 특정한 예측값을 기준으로 성능을 평가하고 싶을 수도 있을 것이다. 예를 들어, 예측 확률 0.5 이상에서 모든 성능이 결과에 대해 양성으로 간주하고 예측 확률 0.5 미만에서 모두 음성으로 간주하는 것을 확인하려면 “0.5”를 cutPoints 인자 목록에 추가한다. 예를 들어 다음과 같다.
setwd("c:/temp")
connectionDetails <- createConnectionDetails(
dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
phenoResult <- testPhenotype(
connectionDetails = connectionDetails,
cutPoints = c(0.1, 0.2, 0.3, 0.4, 0.5, "EV", 0.6, 0.7, 0.8, 0.9),
resultsFileName =
"c:/temp/lr_results_5XMI_eval_myCDM_ePPV0.75_20181206V1.rds",
modelFileName =
"c:/temp/lr_results_5XMI_train_myCDM_ePPV0.75_20181206V1.rds",
cohortPheno = 1769702,
phenText = "All MI by Phenotype 1 X In-patient, 1st Position",
order = 1,
testText = "MI xSpec Model - 5 X MI",
cohortDatabaseSchema = "my_results",
cohortTable = "cohort",
cdmShortName = "myCDM")이 예제에서 넓은 범위의 예측 임계값 prediction threshold이 예측값 expected value(“EV”)와 함께 cutPoints로 사용되었다. 주어진 설정에서, 이 단계에서 각각의 예측 임계값에 대한 민감도와 특이도와 같은 성능을 측정하여 제공한다. 평가는 이전 단계에서 만든 평가용 코호트과의 비교를 통해 이루어진다. 더욱 자세한 정보는 결과값으로 나온 데이터 프레임을 CSV로 저장하여 확인할 수 있다.
이러한 과정을 통해서 5개의 데이터 모음에서 4가지의 심근경색 코호트의 성능을 평가한 자료를 표 16.1에서 볼 수 있다. Cutrona 등이 평가한 것과 비슷한 코호트 (“>=1 X HOI, In-Patient”)의 경우 우리가 확인한 양성 예측도는 67% (59%-74%)이다.
| Phenotype Algorithm | Database | Sens | PPV | Spec | NPV |
|---|---|---|---|---|---|
| >=1 X HOI | CCAE | 0.761 | 0.598 | 0.997 | 0.999 |
| Optum1862 | 0.723 | 0.530 | 0.995 | 0.998 | |
| OptumGE66 | 0.643 | 0.534 | 0.973 | 0.982 | |
| MDCD | 0.676 | 0.468 | 0.990 | 0.996 | |
| MDCR | 0.665 | 0.553 | 0.977 | 0.985 | |
| >= 2 X HOI | CCAE | 0.585 | 0.769 | 0.999 | 0.998 |
| Optum1862 | 0.495 | 0.693 | 0.998 | 0.996 | |
| OptumGE66 | 0.382 | 0.644 | 0.990 | 0.971 | |
| MDCD | 0.454 | 0.628 | 0.996 | 0.993 | |
| MDCR | 0.418 | 0.674 | 0.991 | 0.975 | |
| >=1 X HOI, In-Patient | CCAE | 0.674 | 0.737 | 0.999 | 0.998 |
| Optum1862 | 0.623 | 0.693 | 0.998 | 0.997 | |
| OptumGE66 | 0.521 | 0.655 | 0.987 | 0.977 | |
| MDCD | 0.573 | 0.593 | 0.995 | 0.994 | |
| MDCR | 0.544 | 0.649 | 0.987 | 0.980 | |
| 1 X HOI, In-Patient, 1st Position | CCAE | 0.633 | 0.788 | 0.999 | 0.998 |
| Optum1862 | 0.581 | 0.754 | 0.999 | 0.997 | |
| OptumGE66 | 0.445 | 0.711 | 0.991 | 0.974 | |
| MDCD | 0.499 | 0.666 | 0.997 | 0.993 | |
| MDCR | 0.445 | 0.711 | 0.991 | 0.974 |
16.5 근거의 일반화
코호트는 특정 관찰 데이터베이스의 맥락 안에서 잘 정의되고 완전히 평가될 수 있지만, 임상 타당성은 그 결과가 관심 대상 모집단에 일반화될 수 있다고 간주하는 정도에 의해 제한된다. 동일한 주제에 대한 복수의 관찰형 연구는 서로 다른 결과를 산출할 수 있는데, 이는 설계와 분석 방법뿐만 아니라 데이터 출처의 선택에도 의해 발생할 수 있다. Madigan et al. (2013) 은 데이터베이스의 선택이 관찰 연구의 결과에 영향을 미친다는 것을 보여주었다. 그들은 10개의 관찰형 데이터베이스에 걸쳐 53개의 약물-결과 쌍과 2개의 연구 설계 (코호트 연구 설계 및 자기 대조군 환자 연구 설계)에 대한 결과에서 이질성을 체계적으로 조사했다. 연구 설계를 일정하게 유지했음에도 불구하고, 실제 추정치의 상당한 이질성이 관찰되었다.
OHDSI 네트워크의 관찰형 데이터베이스는 그들이 반영하는 인구 집단 (예를 들어 소아 대 노인, 사보험 가입자 대 공공보험 가입자), 데이터가 수집되는 치료 환경 (예를 들어 입원 환자 대 외래 환자, 1차 의료기관 대 2차, 특수 의료기관), 데이터 수집 프로세스 (예를 들어 행정 청구, 전자 건강 기록, 임상 레지스트리) 및 치료의 기반이 되는 국가 및 지역 보건 시스템 등에서 상당한 차이가 있다. 이러한 차이는 질병과 의료 개입의 영향을 연구할 때 관찰되는 결과의 이질성으로 나타날 수 있으며 네트워크 연구를 위한 각 데이터베이스의 품질에 대한 신뢰도에 영향을 미칠 수도 있다. OHDSI 네트워크 내의 모든 데이터베이스는 CDM으로 표준화되지만, 데이터 표준화는 모집단 전체에 존재하는 진정한 고유의 이질성을 없애기 위함이 아니라, 네트워크 전체의 이질성을 인식하고 더 잘 이해하기 위한 일관된 프레임워크 제공을 강화하기 위함이라는 것이 중요하다. OHDSI 연구 네트워크는 전 세계의 다양한 데이터베이스에 동일한 분석 프로세스를 적용할 수 있는 환경을 제공하기 때문에 연구자는 데이터 외 방법론적 측면을 일정하게 유지하면서 여러 데이터베이스에 걸쳐 결과를 해석할 수 있다. OHDSI의 커뮤니티의 오픈 사이언스에 대한 협력적 접근은 임상 영역의 전문가와 방법론적 분석 전문가가 함께 OHDSI 네트워크의 데이터에 걸쳐 임상적 타당성을 이해하는 집단 지성에 도달하기 위한 한 가지 방법이며, 이는 이 데이터를 이용하여 만들어진 근거의 신뢰를 구축하기 위한 근본이 되어야 한다.
16.6 요약
- 임상적 타당성은 원천 데이터의 특성을 이해하고, 분석 내의 코호트 정의에 대한 성능을 평가하고, 대상 모집단에 관한 연구의 일반성을 평가함으로써 확립할 수 있다.
- 코호트 정의 및 가용한 관찰 데이터에 근거하여 식별된 개인이 의도한 표현형에 진정으로 속하는지 여부를 판단하여 코호트 정의의 성능을 평가할 수 있다.
- 코호트 정의 타당성 검사를 위해서는 측정 오류를 완전히 요약하고 조정할 수 있도록 민감도, 특이도, 양성예측도를 포함한 여러 성능 지표를 추정해야 한다.
- 원천 기록 확인을 통한 임상적 판단 및 PheValuator는 코호트 정의 타당성 검사에 대한 두 가지 대안적 접근 방식이다.
- OHDSI 네트워크 연구는 데이터베이스의 이질성을 평가하고 연구 결과의 일반성을 확장하여 실세계 근거의 임상적 타당성을 향상하는 메커니즘을 제공한다.
References
Hripcsak, G., and D. J. Albers. 2017. “High-fidelity phenotyping: richness and freedom from bias.” J Am Med Inform Assoc, October.
Madigan, D., P. B. Ryan, M. Schuemie, P. E. Stang, J. M. Overhage, A. G. Hartzema, M. A. Suchard, W. DuMouchel, and J. A. Berlin. 2013. “Evaluating the impact of database heterogeneity on observational study results.” Am. J. Epidemiol. 178 (4): 645–51.
Rubbo, B., N. K. Fitzpatrick, S. Denaxas, M. Daskalopoulou, N. Yu, R. S. Patel, H. Hemingway, et al. 2015. “Use of electronic health records to ascertain, validate and phenotype acute myocardial infarction: A systematic review and recommendations.” Int. J. Cardiol. 187: 705–11.
Suchard, M. A., S. E. Simpson, Ivan Zorych, P. B. Ryan, and David Madigan. 2013. “Massive Parallelization of Serial Inference Algorithms for a Complex Generalized Linear Model.” ACM Trans. Model. Comput. Simul. 23 (1). New York, NY, USA: ACM: 10:1–10:17. doi:10.1145/2414416.2414791.
Swerdel, J. N., G. Hripcsak, and P. B. Ryan. 2019. “PheValuator: Development and Evaluation of a Phenotype Algorithm Evaluator.” J Biomed Inform, July, 103258.