Chapter 15 데이터의 질
Chapter leads: Martijn Schuemie, Vojtech Huser & Clair Blacketer
관찰 의료 연구에서 사용되는 대부분의 데이터는 연구를 목적으로 수집되지 않는다. 예를 들어, 전자 의무 기록 Electronic Health Records(EHR)은 환자의 진료를 지원하는데 필요한 정보를 수집하기 위해, 청구 데이터는 비용 지불자에게 비용을 청구하기 위한 근거를 제공하기 위해 수집된다. 많은 이가 이러한 데이터를 임상 연구에 사용하는 것이 적합한지 여부에 의문을 가지고 있으며 심지어, Lei (1991) 는 “데이터는 수집된 목적으로만 사용되어야 한다 (Data shall be used only for the purpose for which they were collected)”고 주장하였다. 문제는 데이터가 우리가 원하는 연구를 위해 수집되지 않았기 때문에, 충분한 품질을 보장할 수 없다는 것이다. 데이터의 품질이 낮으면 (garbage in), 그 데이터를 사용한 연구 결과의 품질도 낮을 수밖에 없다 (garbage out). 따라서 관찰 의료 연구에 있어서 데이터 품질을 평가하는 것은 중요하며, 다음의 질문에 답하는 것을 목표로 한다:
연구 목적에 적합한 데이터인가 (Are the data of sufficient quality for our research purposes) ?
우리는 데이터 품질 Data Quality(DQ)을 다음과 같이 정의할 수 있다 (Roebuck 2012):
데이터를 특정 목적에 적합하게 만드는 완전성 Completeness, 유효성 Validity, 일관성 Consistency, 적시성 Timeliness, 정확성 Accuracy의 상태 (The state of completeness, validity, consistency, timeliness and accuracy that makes data appropriate for a specific use).
주목할만한 것은 우리의 데이터가 완벽하지는 않지만, 목적에 충분히 적합할 수 있다는 것이다.
DQ를 직접적으로 관찰할 수는 없지만, 이를 평가하기 위한 방법론이 개발되어 왔다. DQ 평가는 2가지 유형으로 구분될 수 있다 (Weiskopf and Weng 2013): 보편적인 DQ를 확인하기 위한 평가, 특정 연구의 맥락에서 DQ를 확인하기 위한 평가.
본 장에서 우리는 먼저, DQ 문제가 발생할 수 있는 원인에 대해 검토하고, 보편적인 DQ와 연구 목적별 DQ 평가 이론에 대해 논의 후, OHDSI 도구를 사용하여 이러한 평가를 어떻게 수행하는지 단계별로 설명하고자 한다.
15.1 데이터 품질 문제에 대한 원인
14장에서 언급한 바와 같이, 의사들이 본인의 생각을 기록할 때 데이터 품질과 관련된 많은 위험요소가 발생한다. Dasu and Johnson (2003) 은 데이터의 수명 주기에 따른 단계를 명시하였고, 각 단계를 통합한 DQ 진행을 제시하였다. 그들은 이를 DQ 연속체 DQ continuum라 하였다:
- 데이터 수집 및 통합 Data gathering and integration. 자료의 수기 입력 시 오류와 비뚤림 등의 발생 가능한 문제 (예를 들어 upcoding in claims; 청구를 위하여 없는 진단명을 추가하는 등), EHR에서 잘못된 테이블 간의 결합, 결측값을 기본값으로 대체하는 것 등을 포함한다.
- 데이터 저장 및 지식 공유 Data storage and knowledge sharing. 데이터 모델에 대한 문서화 부족, 메타 데이터의 부족이 잠재적인 문제로 여겨진다.
- 데이터 분석 Data analysis. 잘못된 데이터 변환, 부정확한 데이터 해석, 그리고 부적절한 방법론 사용 등의 문제가 포함될 수 있다.
- 데이터 공유 Data publishing. 후속 사용을 위해 데이터를 게시하는 경우 (When publishing data for downstream use).
우리가 사용하는 데이터는 대부분 이미 수집되고 통합되어 있기 때문에, 1단계에서 개선할 수 있는 것은 거의 없다. 이 단계에서 생성된 DQ를 확인할 방법은 다음 절에서 논의될 것이다.
유사하게, 특정 형식으로 데이터를 받기 때문에 2단계에 대한 영향을 줄 수 있는 부분도 미미하다. 하지만 OHDSI에서는 관찰 데이터를 CDM으로 변환하기 때문에 이 변환 프로세스에 대한 주도권을 가지고 있다. 몇몇은 이러한 특정 단계가 DQ를 저하할 것이라 우려를 표한다. 하지만 우리는 이 변환 프로세스를 통제하기 때문에, 이후 15.2.2절에서 논의하는 것과 같이 DQ를 보존하기 위한 엄격한 안전장치를 구축할 수 있다. 여러 연구 (Defalco, Ryan, and Soledad Cepeda 2013; Makadia and Ryan 2014; Matcho et al. 2014; Voss, Ma, and Ryan 2015; Voss et al. 2015; Hripcsak et al. 2018)에 따르면 이 과정이 제대로 실행된다면 CDM으로 변환했을 때 오류가 거의 발생하지 않는 것으로 나타났다. 실제로, 대규모 공동체에 의해 공유되는 잘 문서화된 데이터 모델은 명백하고 명확한 방법으로 데이터 저장을 용이하게 한다.
3단계 (데이터 분석) 또한 우리의 통제 아래에 있다. OHDSI에서 우리는 이 단계의 품질 문제에 대해 DQ라는 용어 대신 각각 16장, 17장, 그리고 18장에서 다루는 임상적 타당성 clinical validity, 소프트웨어의 타당성 software validity 그리고 방법론적 타당성 method validity이라는 용어를 사용한다.
15.2 보편적인 데이터 품질
우리는 우리의 데이터가 관찰 연구의 보편적인 목적에 적합한지 여부에 대해 의문을 가질 수 있다. Kahn et al. (2016) 은 보편적인 DQ가 3가지 구성요소로 구성되어있다고 정의하였다:
- 적합성 Conformance: 데이터값이 지정된 표준과 형식을 준수하는가? 3가지 하위 유형으로 식별된다:
- Value: 기록된 데이터의 요소가 지정된 형식과 일치하는가? 예를 들어 모든 의료 제공자 Provider의 진료와 specialties는 유효한 전문 분야인가?
- Relational: 기록된 데이터가 지정된 관계적 제약 relational constraints 과 일치하는가? 예를 들어 DRUG_EXPOSURE 테이블의 PROVIDER_ID가 PROVIDER 테이블에도 상응하는 기록을 가지고 있는가?
- Computation: 데이터에 대한 계산이 의도한 결과를 산출하는가? 예를 들어 키와 몸무게에서 계산된 BMI와 데이터에 기록된 BMI가 일치하는가?
- 완전성 Completeness: 특정 변수가 존재하는지 여부 (예를 들어 진료실에서 측정된 체중이 기록되어 있는가?) 와 모든 변수의 값이 기록되어 있는지 (예를 들어 모든 사람이 성별에 관련된 데이터를 가지고 있는가?) 를 나타낸다.
- 타당성 Plausibility: 데이터의 값을 믿을 수 있는가? 3가지 하위 유형으로 정의된다:
- Uniqueness: 예를 들어 각각의 PERSON_ID는 PERSON 테이블에서 한 번만 발생하는가?
- Atemporal: 값, 분포 또는 밀도가 예상되는 값과 일치하는가? 예를 들어 데이터에 의해 계산된 당뇨병 유병률이 실제 알려진 유병률과 일치하는가?
- Temporal: 값의 변화가 예상 범위 내에서 일어나는가? 예를 들어 예방접종 순서는 권고사항과 일치하는가?
각각의 구성요소는 두 가지 방법으로 평가될 수 있다:
- 검증 Verification 외부 참조에 의존하지 않고 모델과 메타데이터의 데이터 제약, 시스템 추정, 그리고 기관 내 지식을 집중적으로 확인한다. 검증의 주요 특징은 기관 환경 내의 자원을 사용하여 예상되는 값과 분포를 설명하는 능력이다.
- 검토 Validation 관련된 외부 기준 benchmarks과 관련된 데이터 값과의 일치에 주력한다. 외부 기준으로 사용 가능한 원천으로는 다기관의 데이터를 결합한 결과가 될 수 있다.
15.2.1 데이터 품질 검사
Kahn은 데이터가 주어진 요구 조건을 준수하는지 확인하기 위해 데이터 품질 확인 data quality check (때로는 데이터 품질 규칙 data quality rule이라고도 함)이라는 용어를 도입하였다 (예를 들어 부정확한 출생 연도 또는 사망 사건의 누락으로 인해 141세라는 환자의 신뢰할 수 없는 연령 자료 삭제). 우리는 자동화된 DQ 도구를 만들어 소프트웨어 내에서 위와 같은 검사를 진행할 수 있다. 이러한 도구 중 하나가 ACHILLES Automated Characterization of Health Information at Large-scale Longitudinal Evidence Systems(ACHILLES)이다. (Huser et al. 2018) ACHILLES는 CDM에 부합하는 데이터베이스의 특성과 시각화를 제공하는 소프트웨어 도구이다. 따라서 데이터베이스 네트워크에서 DQ를 평가하는데 사용할 수 있다 (Huser et al. 2016). ACHILLES는 독립형 도구로써 사용 가능하며, “데이터 원천 Data Sources” 기능으로 ATLAS 안에도 통합되어 있다.
ACHILLES는 분석마다 분석 ID와 간단한 설명을 지닌 170개 이상의 데이터 특성 분석을 사전 계산한다. 이와 관련된 두 가지 예시는 다음과 같다. “715: DRUG_CONCEPT_ID에 의한 DAYS_SUPPLY의 분포” 그리고 “506: 성별에 따른 사망 연령 분포”. 이러한 분석 결과는 데이터베이스에 저장되며, 웹 뷰어 web viewer 또는 ATLAS에서 확인 할 수 있다.
공동체에서 DQ 평가를 위해 만든 또 다른 도구로 Data Quality Dashboard(DQD)가 있다. ACHILLES가 특성화 분석을 실행하여 CDM 인스턴스 instance에 대한 전반적인 시각적 이해를 제공한다면, DQD는 테이블별, 필드 별로 주어진 규격에 적합하지 않은 CDM의 기록 수를 제공한다. 전체적으로, 1,500건 이상의 확인이 수행되고, 각 확인은 Kahn의 프레임워크로 구성된다. 각 DQ의 결과는 임계값과 비교되며, FAIL은 임계값을 위반하는 행을 백분율로 계산한 결과로 결정된다. 표 15.1은 체크포인트 예시를 보여준다.
| 위반 행의 분율 | 확인 내용 설명 | 임계값 | 상태 |
|---|---|---|---|
| 0.34 | VISIT_OCCURRENCE의 provider_id가 CDM specification에 규정된 데이터 형식인가를 예 / 아니오로 나타낸 값. | 0.05 | FAIL |
| 0.99 | MEASUREMENT 테이블의 measurement_source_value 필드에서 0으로 매핑된 고유 원천 데이터의 수와 백분율. | 0.30 | FAIL |
| 0.09 | DRUG_ERA 테이블의 drug_concept_id 필드가 성분명 등급에 적합하지 않은 값을 가진 레코드 수와 백분율. | 0.10 | PASS |
| 0.02 | DRUG_EXPOSURE 테이블에서 verbatim_end_date 필드에 drug_exposure_start_date 이전에 발생한 값이 있는 레코드 수와 백분율. | 0.05 | PASS |
| 0.00 | PROCEDURE_OCCURRENCE 테이블의 procedure_occurrence_id 필드에 중복되는 값이 있는 레코드 수와 백분율. | 0.00 | PASS |
DQ 확인 도구는 여러 가지 방법으로 구성되며 테이블, 필드, concept 수준의 확인이 예시가 될 수 있다. 테이블 점검은 CDM 내에서 상위 수준에서 수행되는 점검으로 예를 들면 모든 필수 테이블이 존재하는지를 확인하는 것이다. 필드 수준의 확인은 모든 테이블의 모든 필드가 CDM 규격에 적합한지 평가하는 방법으로 수행된다. 이는 모든 기본 키가 실제로 고유한지 확인하는 것과 모든 표준 concept 필드가 수많은 CONCEPT_ID 중 적절한 도메인의 CONCEPT_ID 사용하는지 확인하는 것이 포함된다. Concept 수준의 검사는 개별적인 CONCEPT_ID를 확인하기 위해 조금 더 깊이 들어간다. 이 중 상당수가 Kahn의 프레임워크 중 타당성 Plausibility 항목에 해당하며 성별과 관련된 특정 개념이 부적절한 성별에 할당되지 않도록 보장하는 것이 예시로 해당된다 (예를 들어 여성 환자에서 전립선암).
ACHILLES와 DQD는 CDM 데이터를 대상으로 실행된다. 이렇게 식별된 DQ 문제는 CDM으로의 변환 과정이 원인일 수 있지만, 원천 데이터 상에서 이미 존재하는 DQ 문제를 반영할 수도 있다. 만일 변환 과정의 문제로 확인되는 경우 일반적으로 문제 해결을 연구자의 역량 내에서 진행할 수 있지만, 원천 데이터의 오류로 인한 문제의 유일한 조치는 오류 데이터 자체를 삭제하는 것이다.
15.2.2 ETL 단위 검정
상위 레벨의 데이터 품질 확인뿐만 아니라, 개별 수준의 데이터 품질 확인도 수행되어야 한다. 데이터가 CDM으로 변환되는 추출 변환 적재 Extract-Transform-Load(ETL) 과정은 종종 상당히 복잡하고, 이러한 복잡성으로 인해 실수를 눈치채지 못할 위험이 된다. 더욱이, 시간이 지남에 따라 원천 데이터 모델이 변경되거나, CDM 버전이 업데이트될 수 있으므로, ETL 과정의 수정이 필수적으로 진행되어야 한다. ETL과 같이 복잡한 과정의 변경은 의도하지 않은 결과를 초래할 수 있어, ETL의 모든 측면을 재고하고 검토해야 한다.
ETL의 향후 계획을 명확히 하고 지속적인 작업 진행을 위해 하나의 단위 검정 Unit test을 구성하는 것을 적극적으로 권장한다. 단위 검정이란 하나의 측면을 자동으로 확인하는 작은 코드 조각이다. 6장에서 설명한 Rabbit-in-a-Hat 도구로 이러한 단위검정을 더욱 쉽게 작성할 수 있는 단위 검정 프레임워크를 만들 수 있다. 이 프레임 워크는 원천 데이터베이스와 대상으로 하는 CDM 버전의 ETL을 위해 특별히 작성된 R 함수의 집합이다. 이러한 함수 중 일부는 원천 데이터 스키마를 준수하는 가짜 데이터 항목을 만들기 위한 것이며, 다른 일부는 CDM 형식으로 데이터에 대한 예상값을 정하는 데 사용될 수 있다. 단위 검정에 대한 예시는 다음과 같다:
source("Framework.R")
declareTest(101, "Person gender mappings")
add_enrollment(member_id = "M000000102", gender_of_member = "male")
add_enrollment(member_id = "M000000103", gender_of_member = "female")
expect_person(PERSON_ID = 102, GENDER_CONCEPT_ID = 8507
expect_person(PERSON_ID = 103, GENDER_CONCEPT_ID = 8532)예제에서, Rabbit-in-a-Hat에 의해 생성된 프레임워크는 나머지 코드에서 사용되는 함수를 불러오는 출처가 된다. 이후에 성별 매핑 Person gender mappings에 대한 테스트를 시작할 것이라 선언하였다. 원천 스키마는 ENROLLMENT 테이블을 가지고 있고, 우리는 Rabbit-in-a-Hat에서 생성된 add_enrollment 함수를 사용하여 MEMBER_ID와 GENDER_OF_MEMBER 필드에 대해 서로 다른 값을 지닌 두 개의 항목을 만들었다. 마지막으로, ETL 이후 PERSON 테이블에서 다양한 예상값을 지닌 두 개의 항목이 존재해야 한다는 것을 명시한다.
ENROLLMENT 테이블에는 다른 필드가 많이 존재하지만, 이 테스트의 맥락에서는 다른 필드가 어떤 값을 가지는지를 설명하지 않을 것이다. 하지만 이러한 값을 비워두면 (예를 들어 생년월일), 레코드를 삭제하거나 오류를 발생시키는 ETL의 원인이 될 수 있다. 테스트 코드를 읽기 쉽게 유지하면서 이러한 문제를 해결하기 위해서, add_enrollment 함수는 사용자가 명확하게 지정하지 않은 필드의 값에 기본값 (White Rabbit 스캔 보고서에서 관찰된 가장 일반적인 값)을 할당한다.
ETL의 모든 다른 논리에 대해 유사한 단위 검정이 만들어질 수 있으며, 일반적으로 수백 개의 시험을 진행할 수 있다. 테스트를 정의하는 것이 끝나면 프레임워크를 사용하여 두 개의 SQL 구문 모음을 만들 수 있다. 하나는 가짜 원천 데이터를 만드는 것이고, 다른 하나는 ETL된 데이터에 대한 테스트를 진행할 수 있는 구문이다.
insertSql <- generateInsertSql(databaseSchema = "source_schema")
testSql <- generateTestSql(databaseSchema = "cdm_test_schema")전반적인 과정은 그림 15.1에 묘사된 것과 같다.
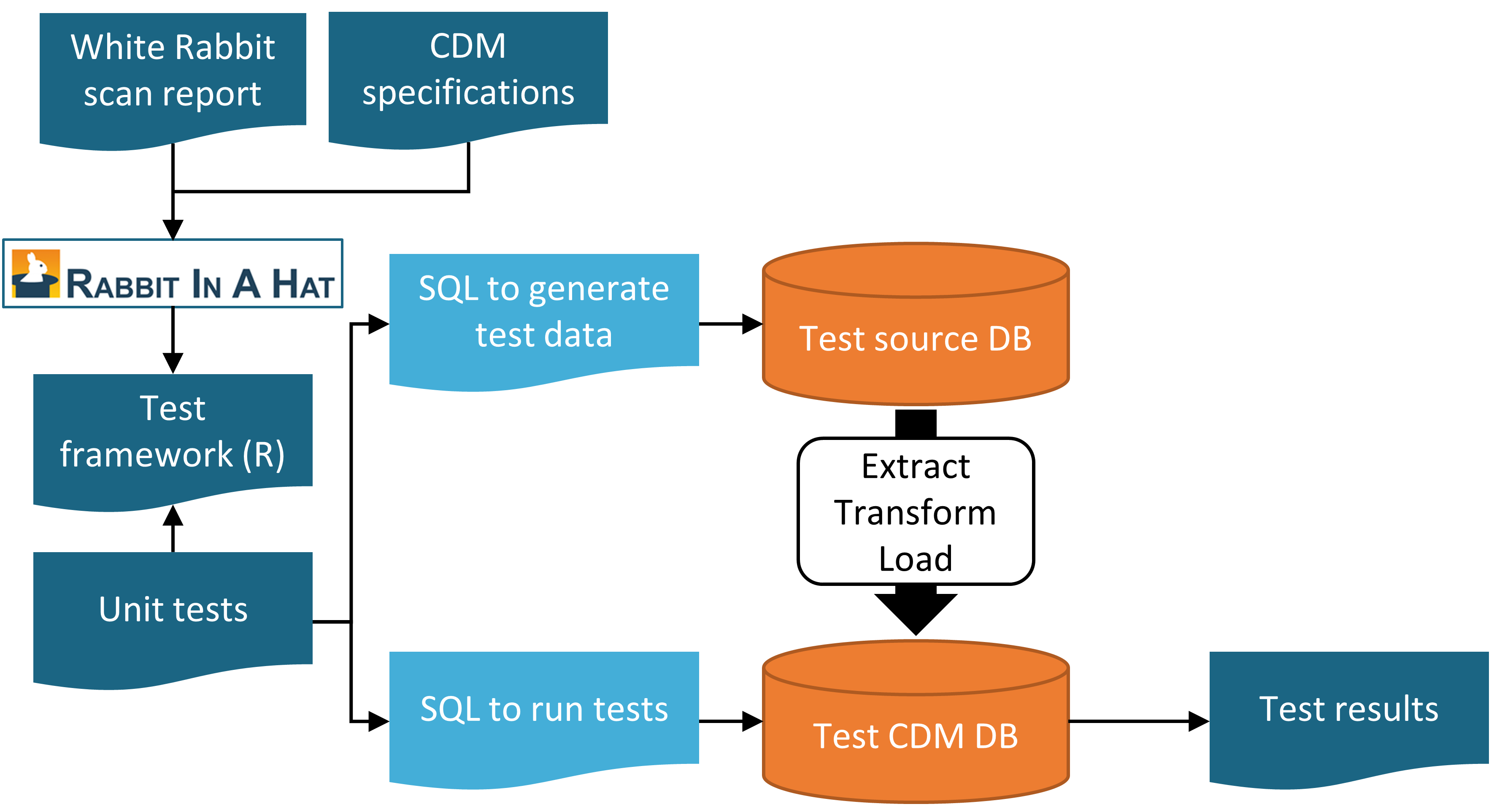
Figure 15.1: Rabbit-in-a-Hat 테스팅 프레임워크를 사용한 ETL 단위 검정 과정.
SQL을 통한 테스트는 표 15.2와 같은 테이블을 반환한다. 이 표에서는 우리가 앞서 정의한 두 가지 테스트를 통과하는 것을 알 수 있다.
| ID | 설 명 | 상 태 |
|---|---|---|
| 101 | 성별 매핑(Person gender mappings) | PASS |
| 101 | 성별 매핑(Person gender mappings) | PASS |
이 단위 검정의 강점은 ETL 프로세스가 변경될 때마다 쉽게 재실행할 수 있다는 것이다.
15.3 연구 별 검사
지금까지 보편적인 DQ 검사에 초점을 맞췄다. 이러한 검사는 데이터가 연구에 사용되기 이전에 실행되어야 한다. 이러한 검사는 연구 문제와 무관하게 수행되어야 하므로 이후에 연구 목적의 DQ 평가를 수행할 것을 권한다.
이러한 평가 중 일부는 특별히 연구와 관련된 DQ 규칙 rule의 형태를 취할 수 있다. 예를 들어, 관심 노출에 대한 레코드의 최소 90%가 노출 기간을 명시한다는 새로운 규칙 도입을 원할 수도 있다.
표준으로 시행하는 검사는 연구와 가장 관련된 concept, 예를 들어 코호트 정의에서 정의된 concept을 ACHILLES에서 검토하는 것이다. 전체기간에서 특정 코드의 사용 빈도가 급격히 변한다면 이것은 DQ 문제가 있다는 것을 알려주는 힌트가 될 수도 있다. 몇몇 예시는 이 장의 뒷부분에서 recommend를 설명하고 있다.
또 다른 평가는 연구를 위해 설정된 코호트 정의를 사용해 생성된 코호트 결과에 대한 유병률과 시간에 따른 유병률의 변화를 검토하고 이것이 외부 임상 지식에 기반한 예상값과 일치하는지 확인하는 것이다. 예를 들어, 신약의 노출은 시장에 소개되기 전에는 없어야 하고, 도입 이후에 시간이 지남에 따라 증가할 가능성이 있다. 유사하게 결과에 대한 유병률은 모집단에서 질환의 유병률에 대해 알려진 것과 일치해야 한다. 만약 연구가 데이터베이스의 네트워크에서 실행한다면, 우리는 데이터베이스 간의 코호트 유병률을 비교할 수 있다. 한 데이터베이스에서 높은 유병률을 보이지만, 다른 데이터베이스에서는 누락된 경우, DQ 문제가 있을 수 있다. 이러한 평가는 16장에서 논의한 바와 같이, 임상적 타당성 clinical validity의 개념과 중복된다는 것을 유의해야 한다. 몇몇 데이터베이스에서는 예상하지 못한 유병률 결과가 나올 수가 있는데, 이는 DQ 문제가 아니라 코호트 정의에서 연구 주제와 부합하는 건강 상태를 온전히 잡아내지 못했거나 데이터베이스마다 환자 모집단이 상이하여 발생할 수 있다.
15.3.1 매핑 검사하기
우리가 통제할 수 있는 오류의 원인 중 한 가지는 원천 용어 코드를 표준 concept에 매핑하는 것이다. 용어 매핑은 정교하게 제작되었으며, 매핑 상의 문제가 있다면 공동체 구성원에 의해 발견되어53에 보고 된후 다음 업데이트에 반영된다. 그런데도 모든 매핑을 직접 확인하는 것은 불가능하고 오류가 계속 존재할 수 있다. 그렇기 때문에, 연구를 수행할 때 연구와 관련 있는 concept의 매핑을 검토해보는 것을 권장한다. 다행히도, CDM에서 표준 용어 Concept뿐만 아니라 원천 코드도 같이 저장하기 때문에 이러한 작업은 쉽게 할 수 있다. 연구에 사용된 concept에 매핑된 원천 코드뿐만 아니라 그렇지 않은 원천 코드도 검토할 수 있다.
원천 코드를 검토하는 방법 중 하나는 MethodEvaluation R 패키지의 checkCohortSourceCodes 함수를 사용하는 것이다. 이 함수는 ATLAS에서 생성된 코호트 정의를 입력 input으로 사용하고 코호트 정의에서 사용된 각 concept 모음에 대해 concept과 매핑되는 원천 코드를 확인한다. 또한 전체 기간에 대한 코드의 빈도를 계산하여 특정 코드에서 발생하는 시간적인 문제를 확인하는 데 도움이 될 수 있다. 그림 15.2 예시 결과는 “우울증 Depression disorder”이라 불리는 concept 모음의 분석을 보여준다. 관심 분야의 데이터베이스에서 이 concept 모음의 가장 보편적인 concept은 440383 (우울증; Depressive disorder)이다. 데이터베이스 내의 ICD-9 코드의 3.11, ICD-10 코드의 F32.8과 F32.89 이 세 가지 코드가 해당 concept으로 매핑이 된 걸 볼 수 있다. 그림의 왼쪽부터 보면 전체로서의 concept은 시간이 지남에 따라 초반에는 증가하지만, 그 후에 급격히 감소하는 것을 볼 수 있다. 개별 코드를 살펴보면, 이러한 하락은 하락 시점에 ICD-9 코드의 사용이 중단되는 것으로 설명될 수 있다는 것을 알 수 있다. 이것이 ICD-10 코드가 사용되기 시작한 것과 같은 시간임에도 불구하고, 결합된 ICD-10 코드의 빈도가 ICD-9 코드의 빈도보다 훨씬 적다. 이 구체적인 예시는 ICD-10 코드 F32.9 (“주요 우울 장애, 단일 에피소드, 불특정”)도 이 concept으로 매핑돼야 했었기 때문이다. 이 문제는 vocabulary에서 해결되었다.
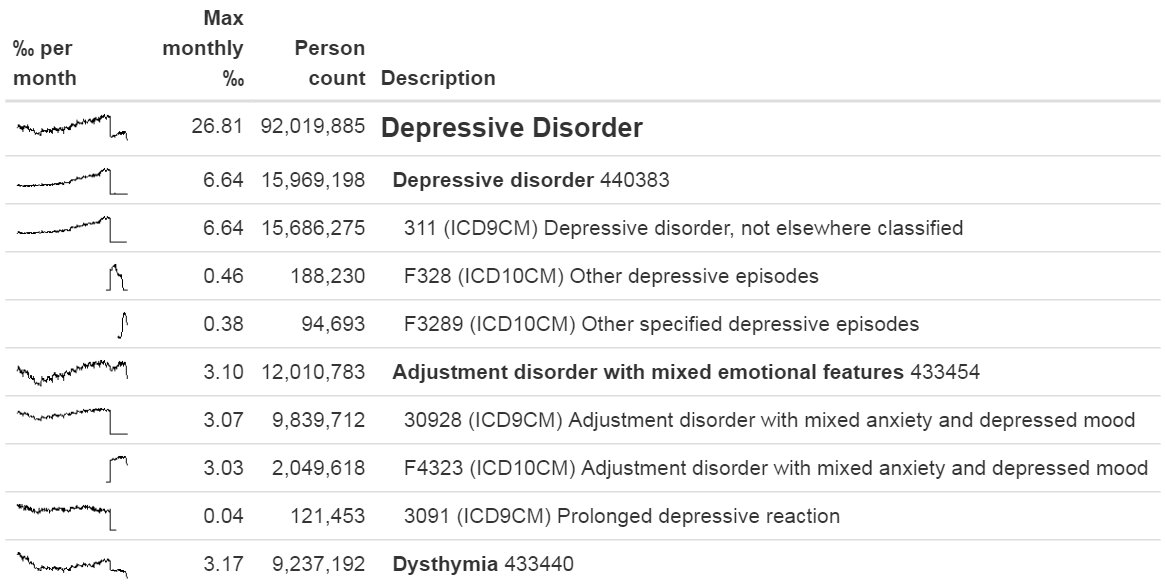
Figure 15.2: checkCohortSourceCodes 기능의 output 예시.
앞의 예시는 매핑되지 않은 원천 용어 코드를 발견하는 것을 묘사한 것으로, 일반적으로 누락된 매핑을 식별하는 것이 존재하는 매핑을 확인하는 것 보다 더 어렵다. 매핑 되어야 하는 원천 용어 코드 중 매핑 되지 않은 것을 알아야 한다. 이를 평가하는 반-자동화된 방법은 MethodEvaluation R 패키지의 findOrphanSourceCodes 함수를 사용하는 것이다. 이 함수는 간단한 텍스트 검색을 통해 원천 용어 코드에 대한 Vocabulary를 검색할 수 있게 하고, 이 원천 용어 코드가 특정 concept이나 그 concept의 하위 concept 중 하나와 매핑되는지 여부를 확인한다. 원천 코드의 결과는 현재 CDM 데이터베이스에 나타나는 코드로만 제한된다. 예를 들어, “괴저 장애 Gangrenous disorder” (439928) 와 모든 하위 concept은 모든 괴저 발생을 찾기 위해 사용되었다. 이것이 실제로 괴저를 나타내는 모든 원천 코드를 포함하는지 여부를 평가하기 위해, 원천 코드를 식별하기 위한 CONCEPT 테이블과 SOURCE_TO_CONCEPT_MAP 테이블의 설명을 검색하는데 몇 가지 용어 (예를 들어 “괴저 gangrene”)가 사용되었다. 자동 검색은 데이터에 나타나는 각 괴저 코드가 “괴저 장애”라는 concept에 직접 또는 간접적으로 매핑되었는지 여부를 평가하기 위해 사용된다. 이러한 평가의 결과는 그림 15.3과 같으며, ICD-10 코드 J85.0 (“폐의 괴저 및 괴사”; Gangrene and necrosis of lung)은 “괴저 장애의 하위 concept이 아닌 concept 4324261 (”폐 괴사“; Pulmonary necrosis)에만 매핑된 것을 알게 되었다.
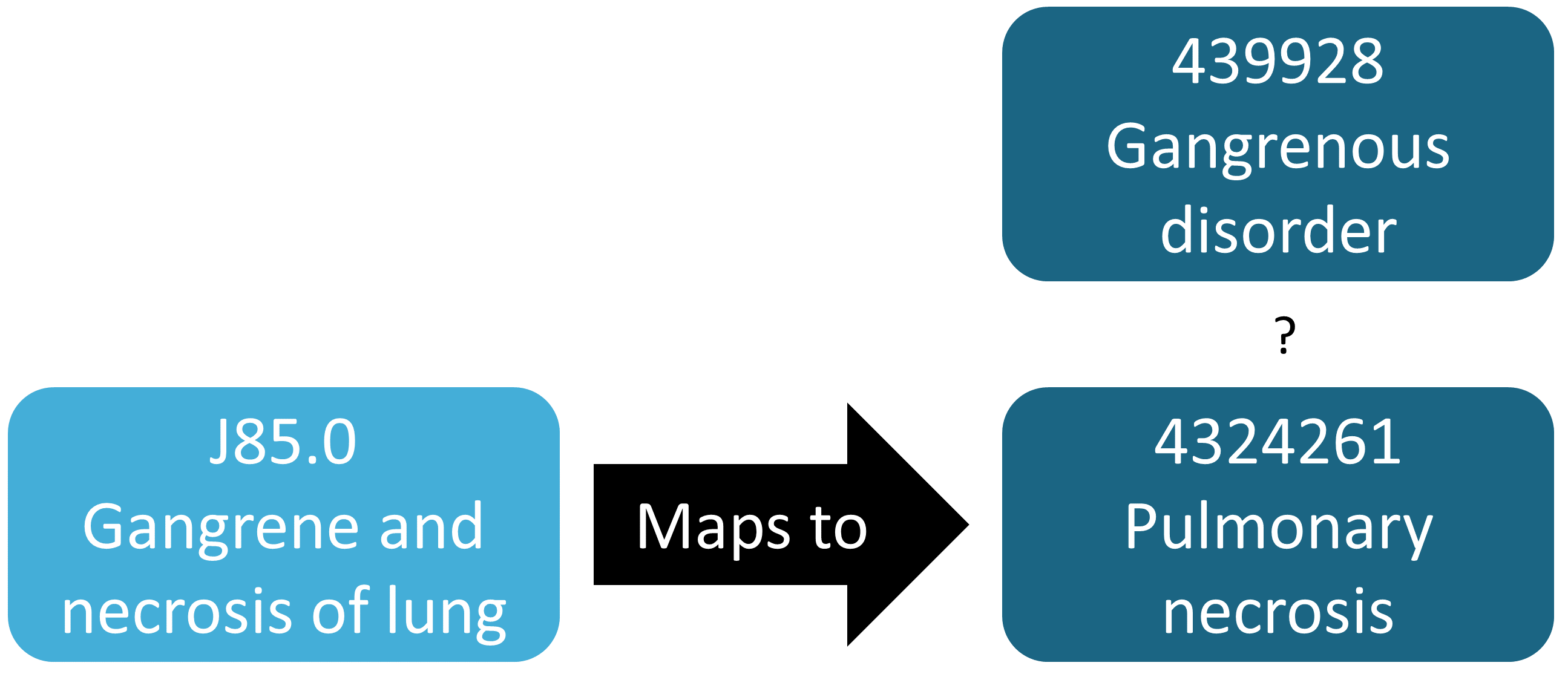
Figure 15.3: orphan 원천 코드 예시.
15.4 ACHILLES 실습
여기서는 CDM 형식의 데이터베이스에 대해 ACHILLES를 실행하는 방법을 보여준다.
먼저, R에서 서버를 연결하는 방법에 관해 설명할 필요가 있다. ACHILLES는 createConnectionDetails라는 함수를 제공하는 DatabaseConnector 패키지를 사용한다. 다양한 데이터베이스 관리 시스템 Database management systems(DBMS)에 필요한 특정 설정을 ?createConnectionDetails을 입력하여 확인할 수 있다. 예를 들어, 다음 코드를 이용하여 PostgreSQL과 연결할 수 있다:
library(Achilles)
connDetails <- createConnectionDetails(dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
cdmDbSchema <- "my_cdm_data"
cdmVersion <- "5.3.0"마지막 두 줄은 CDM 버전뿐만 아니라 cdmDbSchema 변수를 정의한다. 이를 사용하여 CDM 형식의 데이터가 어디에 있는지, 어떤 버전의 CDM이 사용되었는지 R에 입력한다. Microsoft SQL Server의 경우, cdmDbSchema <- "my_cdm_data.dbo"와 같이 데이터베이스와 스키마를 모두 지정해야 한다.
다음으로, ACHILLES를 실행한다:
result <- achilles(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
resultsDatabaseSchema = cdmDbSchema,
sourceName = "My database",
cdmVersion = cdmVersion)이 함수는 resultsDatabaseSchema에 여러 테이블을 생성하며, 여기에서는 CDM 데이터와 동일한 데이터베이스 스키마로 설정하였다.
ATLAS를 ACHILLES 결과 데이터베이스로 지정하거나 ACHILLES 결과를 JSON 파일로 내보내서 ACHILLES 데이터베이스의 특징을 볼 수 있다:
exportToJson(connectionDetails,
cdmDatabaseSchema = cdmDatabaseSchema,
resultsDatabaseSchema = cdmDatabaseSchema,
outputPath = "achillesOut")JSON 파일은 achillesOut 하위 폴더에 작성되고, 결과 확인을 위해 AchillesWeb 웹 애플리케이션과 함께 사용할 수 있다. 예를 들어, 그림 15.4 ACHILLES 데이터 밀도 도표를 보여준다. 이 도표는 2005년에 시작된 대량의 데이터를 보여준다. 하지만 1961년경에 몇 개의 레코드가 있는 것으로 나타나며, 이는 데이터에 오류가 있는 것일 수도 있다.
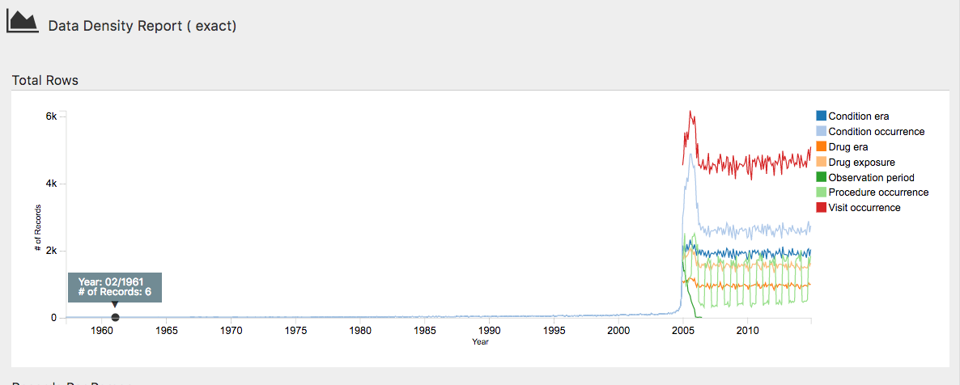
Figure 15.4: ACHILLES 웹 뷰어의 데이터 밀도 그림.
또 다른 예시로는 그림 15.5로, 당뇨병 진단 코드의 유병률에 급격한 변화를 보여주고 있다. 이러한 변화는 특정 국가에서 보험 청구 규정이 변경됨에 따라 진단 수가 증가한 것이지 실제로 유병률이 증가한 것은 아니다.
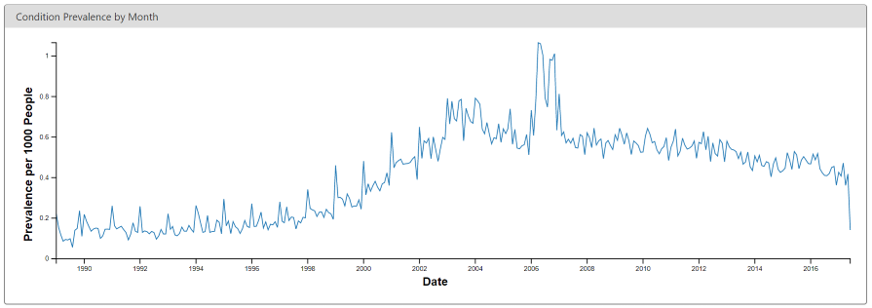
Figure 15.5: ACHILLES 웹 뷰어에서 코딩된 월별 당뇨병 발생률.
15.5 Data Quality Dashboard 실습
여기에서는 CDM 형식의 데이터베이스에서 Data Quality Dashboard를 실행하는 방법을 보여준다. 15.4절에서 설명한 CDM conncection에 대해 더 많은 checks를 수행한다. 현재 DQD는 CDM v5.3.1만 지원하기 때문에 실행 전 데이터베이스가 올바른 버전인지 확인이 필요하다. ACHILLES와 마찬가지로 cdmDbSchema를 작성하여 데이터를 찾을 위치를 R에 입력해야 한다.
cdmDbSchema <- "my_cdm_data.dbo"다음으로, Dashboard를 실행한다…
DataQualityDashboard::executeDqChecks(connectionDetails = connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
resultsDatabaseSchema = cdmDbSchema,
cdmSourceName = "My database",
outputFolder = "My output")위의 함수는 지정된 스키마에서 사용 가능한 모든 데이터 품질 체크포인트를 실행한다. 그런 다음 CDM과 동일한 스키마로 설정한 resultsDatabaseSchema에 테이블을 작성한다. 이 테이블은 CDM 테이블, CDM 필드, 검사명, 설명, Kahn의 카테고리와 하위 카테고리, 위반 행의 수, 임계값 레벨 그리고 검사의 통과 여부 등 각 체크포인트의 실행에 대한 모든 정보가 포함된다. 이 함수는 테이블뿐만 아니라 outputFolder로 JSON 파일을 작성할 위치를 지정한다. JSON 파일을 사용해서 웹 뷰어를 시작해 결과를 확인할 수 있다.
viewDqDashboard(jsonPath)jsonPath 변수는 위의 executeDqChecks 함수가 호출될 때, 지정된 outputFolder에 위치한 Dashboard의 결과가 포함된 JSON 파일의 경로여야 한다.
처음 Dashoboaard를 열면 그림 15.6과 같이 개요 테이블이 표시된다. 여기에는 내용별 Kahn의 카테고리에서 실행된 총 검사의 수, 각 검사의 PASS 수와 백분율 및 전체 통과 비율이 표시된다.
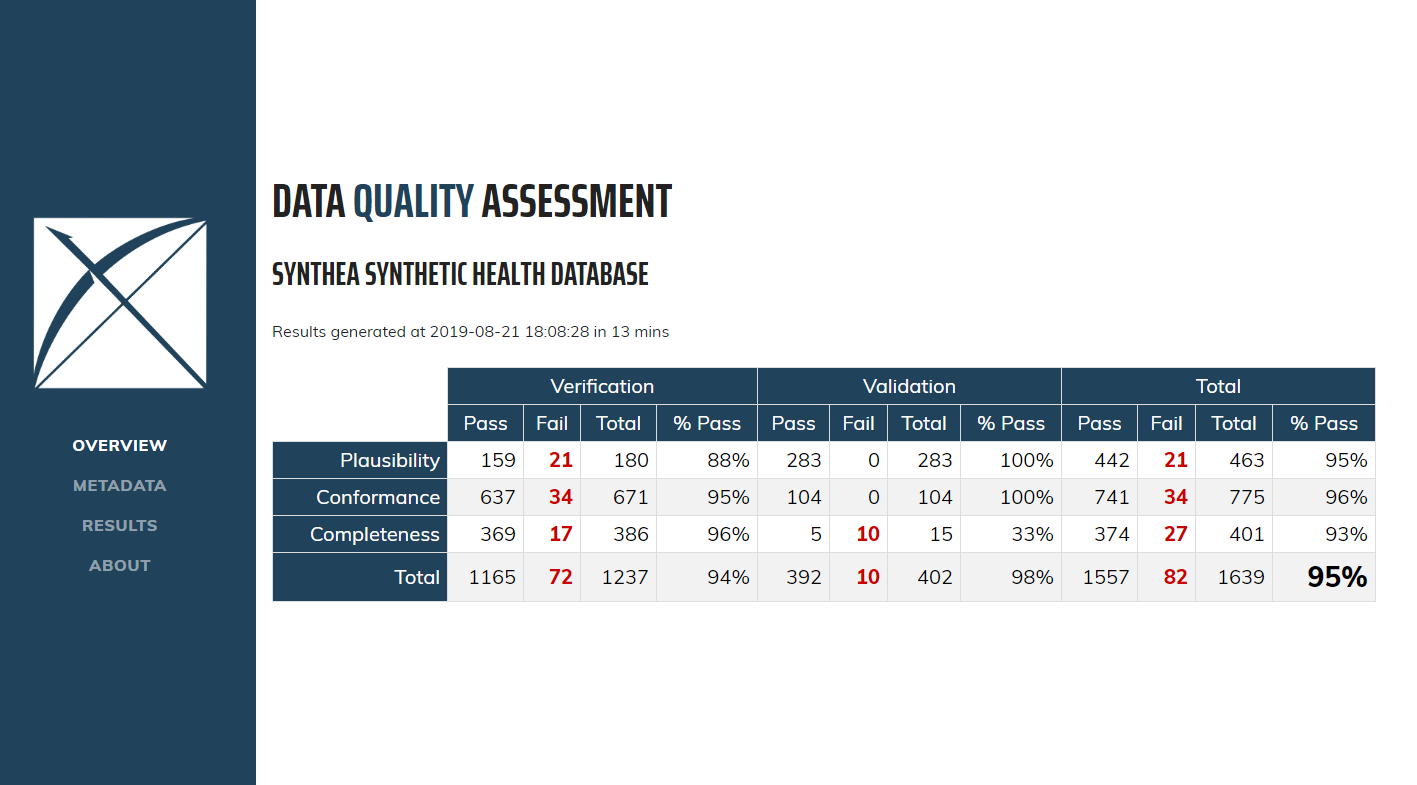
Figure 15.6: 데이터 품질 Dashboard에서의 데이터 품질 점검 개요.
왼쪽 메뉴에서 Results를 클릭하면 실행된 각 검사에 대한 상세 결과 페이지로 이동한다 (그림 15.7 참조). 예시의 테이블은 개별적인 CDM 테이블의 완전성을 확인하거나 CDM에서 특정 테이블에 최소 1개 이상의 레코드를 가진 인원수 및 백분율을 확인하기 위한 검사에 대한 것이다. 이 경우 나열된 5개의 테이블이 Dashboard에 Fail로 나타났으며 모두 비어있다. ![]() 아이콘을 클릭하면 나열된 결과를 생성하기 위해 데이터에서 실행된 정확한 쿼리를 보여주는 창이 열린다. 이를 통해 Dashboard에서 Fail로 간주한 행을 쉽게 식별할 수 있다.
아이콘을 클릭하면 나열된 결과를 생성하기 위해 데이터에서 실행된 정확한 쿼리를 보여주는 창이 열린다. 이를 통해 Dashboard에서 Fail로 간주한 행을 쉽게 식별할 수 있다.
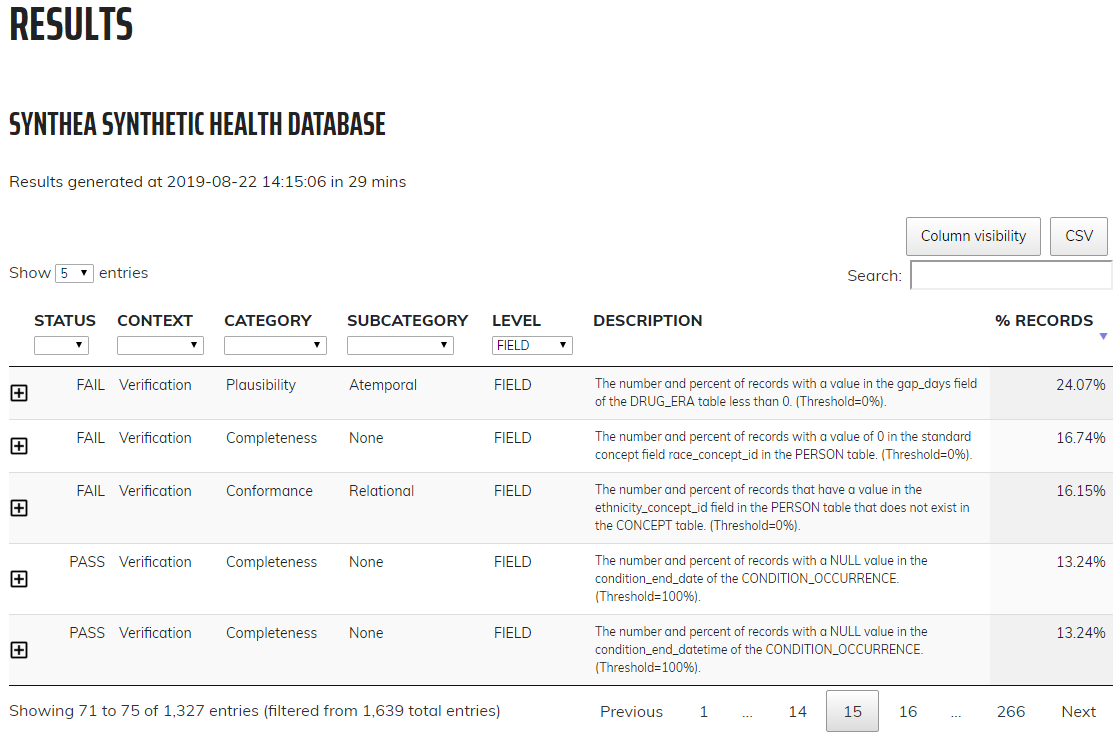
Figure 15.7: 데이터 품질 Dashboard에서의 데이터 품질 평가 구체적 관찰.
15.6 연구별 검사 실습
다음으로, 부록 B.4에 제공된 혈관 부종 코호트 정의에 대한 몇 가지 검사를 수행할 것이다. 15.4절에 설명된 것처럼 연결 세부사항이 설정되어 있고, 코호트 정의 JSON과 코호트 정의에 대한 SQL이 각각 “cohort.json”과 “cohort.sql” 파일에 저장되어있다고 가정한다. JSON 파일과 SQL 파일은 ATLAS 코호트 정의 기능의 내보내기 탭에서 얻을 수 있다.
library(MethodEvaluation)
json <- readChar("cohort.json", file.info("cohort.json")$size)
sql <- readChar("cohort.sql", file.info("cohort.sql")$size)
checkCohortSourceCodes(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
cohortJson = json,
cohortSql = sql,
outputFile = "output.html")그림 15.8과 같이 웹 브라우저에서 출력 output 파일을 열 수 있다. 여기서 혈관 부종 코호트 정의에 “Inpatient or ER visit”과 “Angioedema” 두 가지 concept이 있는 것을 확인할 수 있다. 이 예제 데이터베이스에서, 방문은 ETL 중에 표준 concept과 매핑되었지만, Vocabulary에는 없는, “ER”과 “IP”라는 데이터베이스 특정 원천 코드를 통해 발견되었다. 혈관 부종은 하나의 ICD-9 코드와 두 개의 ICD-10 코드를 통해 발견되었다. 개별 코드에 대한 피크 라인을 봤을 때, 두 가지 코딩 시스템 간의 교대 시점을 명확하게 알 수 있지만, 전체적인 concept에서는 불연속성이 없다.
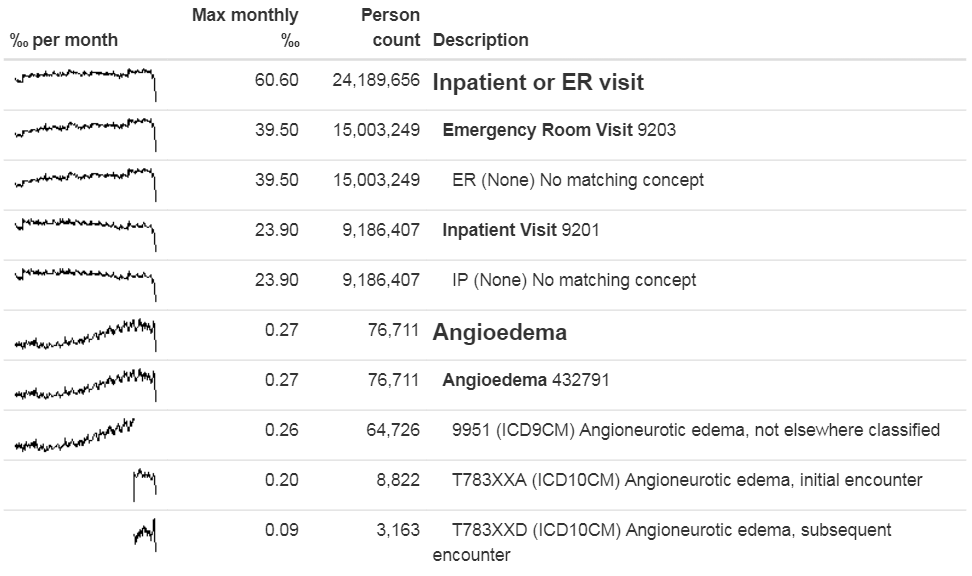
Figure 15.8: Angioedema 코호트 정의에서 사용된 원천 코드.
다음으로, 표준 concept 코드에 매핑되지 않은 원천 코드인 orphan 원천 용어 코드를 검색할 수 있다. 표준 concept인 “혈관 부종 Angioedema”을 찾은 다음 “혈관 부종” 또는 그 이름이 일부 포함되어 있거나 동의어가 있는 concept과 코드를 찾는다:
orphans <- findOrphanSourceCodes(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
conceptName = "Angioedema",
conceptSynonyms = c("Angioneurotic edema",
"Giant hives",
"Giant urticaria",
"Periodic edema"))
View(orphans)| code | 설명 | vocabularyId | overallCount |
|---|---|---|---|
| T78.3XXS | Angioneurotic edema, sequela | ICD10CM | 508 |
| 10002425 | Angioedemas | MedDRA | 0 |
| 148774 | Angioneurotic Edema of Larynx | CIEL | 0 |
| 402383003 | Idiopathic urticaria and/or angioedema | SNOMED | 0 |
| 232437009 | Angioneurotic edema of larynx | SNOMED | 0 |
| 10002472 | Angioneurotic edema, not elsewhere classified | MedDRA | 0 |
데이터에서 실제로 사용된 유일한 잠재적 orphan 코드는 “혈관 신경성 부종 Angioneurotic edema, 후유증 sequela”이며, 이는 혈관 부종과 매핑되어서는 안 된다. 따라서 이 분석에서는 누락된 코드가 발견되지 않았다.
15.7 요약
대부분의 관찰형 의료 데이터는 연구를 위해 수집되지 않는다.
데이터 품질은 데이터가 연구 목적에 적합한지를 확인하기 위해 평가되어야 한다.
보편적인 연구 목적을 위해, 특정 연구의 맥락에서 비판적으로 데이터 품질을 평가해야 한다.
데이터 품질의 일부 측면은 Data Quality Dashboard의 예시와 같이 사전 정의된 많은 규칙을 통해 자동적으로 평가될 수 있다.
- 특정 연구와 관련된 코드 매핑을 평가하기 위한 다른 도구가 있다.
15.8 예제
전제조건
예제 실습을 위해 8.4.5절에서 설명한 것과 같이 R, R-studio 및 Java가 설치되어 있다고 가정한다. 또한 SqlRender, DatabaseConnector, ACHILLES 및 Eunomia 패키지가 필요하다. 아래의 코드를 사용하여 설치할 수 있다:
install.packages(c("SqlRender", "DatabaseConnector", "devtools"))
devtools::install_github("ohdsi/Achilles")
devtools:install_github("ohdsi/DataQualityDashboard")
devtools::install_github("ohdsi/Eunomia", ref = "v1.0.0")Eunomia 패키지는 로컬 R 세션 내에서 실행되는 CDM에서 시뮬레이션 된 데이터 모음을 제공한다. 자세한 접속정보는 아래를 활용하여 얻을 수 있다:
connectionDetails <- Eunomia::getEunomiaConnectionDetails()CDM 데이터베이스 스키마는 “main”이다.
Exercise 15.1 Eunomia 데이터베이스에 대해 ACHILLES를 실행하라.
Exercise 15.2 Eunomia 데이터베이스에 대해 DataQaulityDashbiard를 실행하라.
Exercise 15.3 DQD 검사 목록을 추출하라.
제안된 답변은 부록 E.10에서 찾을 수 있다.
References
Dasu, Tamraparni, and Theodore Johnson. 2003. Exploratory Data Mining and Data Cleaning. Vol. 479. John Wiley & Sons.
Defalco, F. J., P. B. Ryan, and M. Soledad Cepeda. 2013. “Applying standardized drug terminologies to observational healthcare databases: a case study on opioid exposure.” Health Serv Outcomes Res Methodol 13 (1): 58–67.
Hripcsak, G., M. E. Levine, N. Shang, and P. B. Ryan. 2018. “Effect of vocabulary mapping for conditions on phenotype cohorts.” J Am Med Inform Assoc 25 (12): 1618–25.
Huser, Vojtech, Frank J. DeFalco, Martijn Schuemie, Patrick B. Ryan, Ning Shang, Mark Velez, Rae Woong Park, et al. 2016. “Multisite Evaluation of a Data Quality Tool for Patient-Level Clinical Data Sets.” EGEMS (Washington, DC) 4 (1): 1239. doi:10.13063/2327-9214.1239.
Huser, Vojtech, Michael G. Kahn, Jeffrey S. Brown, and Ramkiran Gouripeddi. 2018. “Methods for Examining Data Quality in Healthcare Integrated Data Repositories.” Pacific Symposium on Biocomputing. Pacific Symposium on Biocomputing 23: 628–33.
Kahn, Michael G., Tiffany J. Callahan, Juliana Barnard, Alan E. Bauck, Jeff Brown, Bruce N. Davidson, Hossein Estiri, et al. 2016. “A Harmonized Data Quality Assessment Terminology and Framework for the Secondary Use of Electronic Health Record Data.” EGEMS (Washington, DC) 4 (1): 1244. doi:10.13063/2327-9214.1244.
Lei, Johan van der. 1991. “Use and Abuse of Computer-Stored Medical Records.” Methods of Information in Medicine 30 (02). Schattauer GmbH: 79–80.
Makadia, R., and P. B. Ryan. 2014. “Transforming the Premier Perspective Hospital Database into the Observational Medical Outcomes Partnership (OMOP) Common Data Model.” EGEMS (Wash DC) 2 (1): 1110.
Matcho, A., P. Ryan, D. Fife, and C. Reich. 2014. “Fidelity assessment of a clinical practice research datalink conversion to the OMOP common data model.” Drug Saf 37 (11): 945–59.
Roebuck, Kevin. 2012. Data Quality: High-Impact Strategies-What You Need to Know: Definitions, Adoptions, Impact, Benefits, Maturity, Vendors. Emereo Publishing.
Voss, E. A., Q. Ma, and P. B. Ryan. 2015. “The impact of standardizing the definition of visits on the consistency of multi-database observational health research.” BMC Med Res Methodol 15 (March): 13.
Voss, E. A., R. Makadia, A. Matcho, Q. Ma, C. Knoll, M. Schuemie, F. J. DeFalco, A. Londhe, V. Zhu, and P. B. Ryan. 2015. “Feasibility and utility of applications of the common data model to multiple, disparate observational health databases.” J Am Med Inform Assoc 22 (3): 553–64.
Weiskopf, Nicole Gray, and Chunhua Weng. 2013. “Methods and Dimensions of Electronic Health Record Data Quality Assessment: Enabling Reuse for Clinical Research.” Journal of the American Medical Informatics Association: JAMIA 20 (1): 144–51. doi:10.1136/amiajnl-2011-000681.