Chapter 8 OHDSI 분석 도구
Chapter leads: Martijn Schuemie & Frank DeFalco
OHDSI는 관찰 환자 수준 데이터에 대한 다양한 데이터 분석 사용 사례를 지원하는 광범위한 오픈 소스 도구를 제공한다. 이러한 도구의 공통점은 공통 데이터 모델(CDM)을 사용하여 하나 이상의 데이터베이스와 상호 작용할 수 있다는 것이다. 또한, 이러한 도구는 다양한 사용 사례 use case에 대한 분석을 표준화한다. 처음부터 시작하는 것이 아니라 표준 템플릿을 작성함으로써 분석을 구현할 수 있다. 이렇게 하면 분석을 더 쉽게 수행할 수 있고, 재현성과 투명성을 향상할 수 있다. 예를 들어, 발생률을 계산하는 방법은 무한에 가까운 수가 있는 것처럼 보이지만, 이러한 방법은 몇 가지 선택사항으로 OHDSI 도구에 지정할 수 있으며, 동일한 선택을 하는 사람은 동일한 방법으로 발병률을 계산할 것이다.
이 장에서는 먼저 분석을 실행하기 위해 선택할 수 있는 다양한 방법과 분석에서 어떤 전략을 사용할 수 있는지 설명한다. 그런 다음 다양한 OHDSI 도구와 다양한 사용 사례에 적합한 방법을 검토한다.
8.1 분석 구현
그림 8.1은 CDM을 사용하여 데이터베이스에 대한 연구를 구현하도록 선택할 수 있는 다양한 방법을 보여준다.
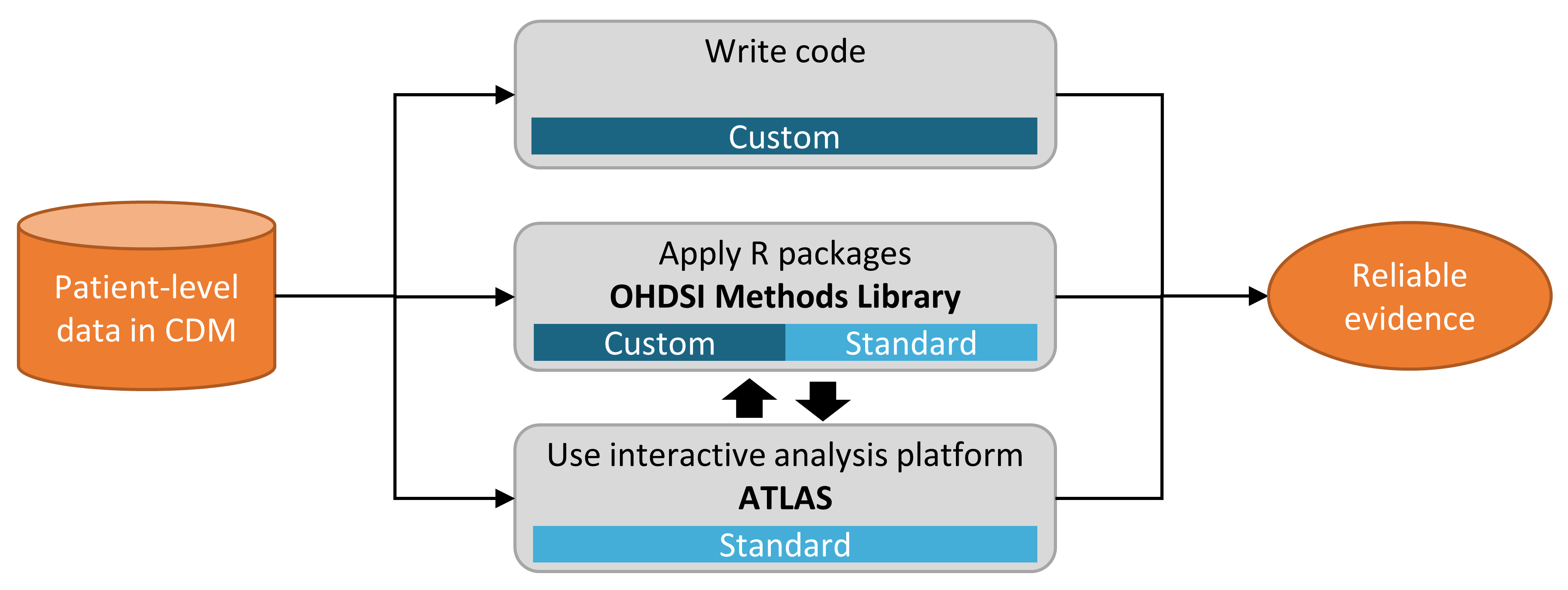
Figure 8.1: CDM의 데이터에 대한 분석을 구현하는 다양한 방법
연구를 이행하는 데는 세 가지 주요 접근법이 있다. 첫 번째는 OHDSI가 제공하는 어떤 도구도 사용하지 않고 사용자가 직접 코드를 작성하는 것이다. R, SAS 또는 다른 언어로 새로운 분석 코드를 작성할 수 있다. 이는 최대의 유연성을 제공하며, 특정 분석이 우리의 도구에 의해 뒷받침되지 않는 경우 사실상 유일한 선택사항이 될 수 있다. 그러나 이러한 경로에는 많은 전문적 기술과 시간, 노력이 필요하며, 분석의 복잡성이 증가함에 따라 코드의 오류를 피하기 어려워진다.
두 번째 접근방식은 R을 이용하고, 연구방법론 라이브러리 OHDSI Methods Library의 패키지를 이용하는 것이다. 최소한 9장에 설명된 SqlRender 및 DatabaseConnector 패키지를 사용하여 PostgreSQL, SQL Server, 그리고 Oracle과 같은 다양한 데이터베이스 플랫폼에서 동일한 코드를 실행할 수 있다. CohortMethod와 PatientLevelPrediction과 같은 다른 패키지는 자신의 코드로 호출할 수 있는 CDM에 대한 고급 분석을 위한 R 기능을 제공한다. 이것은 여전히 많은 기술적 전문지식이 필요하지만, 연구방법론 라이브러리의 검증된 구성요소를 다시 사용함으로써 사용자가 모든 코드를 다 짜는 것보다 더 효율적이고 오류가 덜 발생할 수 있다.
세 번째 접근법은 프로그래머가 아닌 사람이 다양한 분석을 효율적으로 수행할 수 있도록 해주는 웹 기반 도구인 대화형 분석 플랫폼 ATLAS에 의존한다. ATLAS는 Method Libraries를 사용하지만, 분석을 설계하기 위한 간단한 그래픽 인터페이스를 제공하며 많은 경우 분석을 실행하는 데 필요한 R 코드를 생성한다. 그러나 ATLAS는 연구방법론 라이브러리에서 사용할 수 있는 모든 옵션을 다 지원하지는 않는다. 대부분의 연구가 ATLAS를 통해 수행될 수 있을 것으로 예상되지만, 일부 연구는 두 번째 접근방식이 제공하는 유연성을 필요로 할 수 있다.
ATLAS와 연구방법론 라이브러리는 독립적이지 않다. ATLAS에서 호출할 수 있는 더 복잡한 분석 중 일부는 연구방법론 라이브러리의 패키지에 대한 호출을 통해 실행된다. 마찬가지로 연구방법론 라이브러리에 사용되는 코호트는 ATLAS에서 설계되는 경우가 많다.
8.2 분석 전략
사용자 정의 코드를 사용하거나 연구방법론 라이브러리의 표준 분석 코드를 사용하여 CDM에 대한 분석을 구현하는 것 외에도, 그러한 분석 기법을 사용하여 근거를 생성하는 데에는 여러 가지 전략이 있다. 그림 8.2은 OHDSI에 채택된 세 가지 전략을 보여준다.
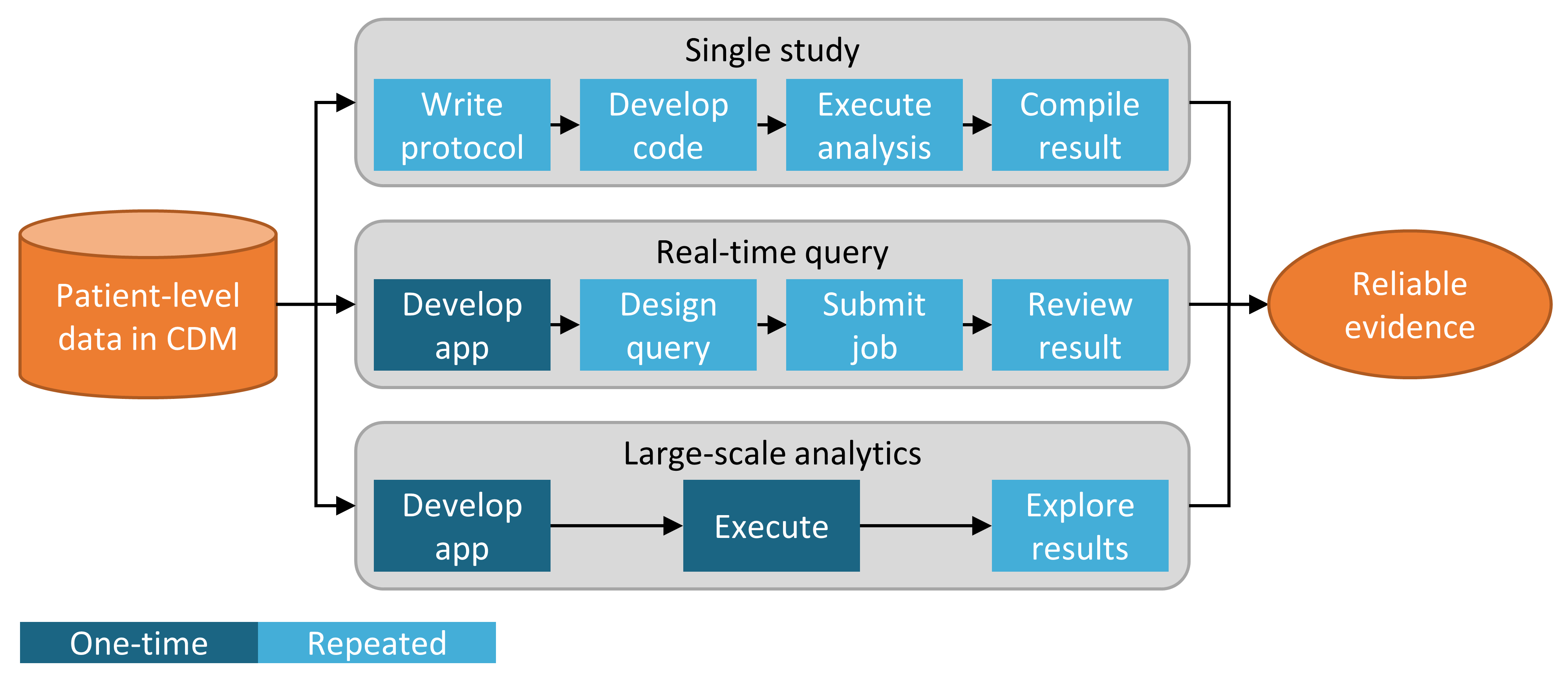
Figure 8.2: (임상적) 질문에 대한 근거를 생성하기 위한 전략
첫 번째 전략은 모든 분석을 하나의 개별적인 연구로 본다. 분석은 프로토콜에 미리 지정되어야 하고, 코드로 구현되어야 하며, 데이터에 대해 실행되어야 하며, 그 후에 결과를 컴파일하고 해석할 수 있어야 한다. 모든 질문에 대해 모든 단계를 반복해야 한다. 그러한 분석의 예로는 phenytoin과 비교하여 levetiracetam과 관련된 혈관부종angioedema의 위험에 대한 OHDSI 연구가 있다. (Duke et al. 2017) 이 연구에서, 프로토콜이 처음으로 작성되었고, OHDSI 연구방법론 라이브러리를 이용한 분석 코드가 OHDSI 네트워크를 통해 개발되어 실행되었으며, 결과를 편집하여 저널 간행물에 배포하였다.
두 번째 전략은 사용자가 특정 종류의 질문에 실시간으로 또는 거의 실시간으로 답할 수 있는 애플리케이션을 개발한다. 애플리케이션이 개발되면 사용자는 애플리케이션과 상호 작용하여 쿼리를 정의하고 제출하고 결과를 볼 수 있다. 이 전략의 예로는 ATLAS의 코호트 정의 및 생성 도구가 있다. 이 도구는 사용자가 다양한 수준의 복잡한 코호트 정의를 내리고 원하는 데이터베이스에 대해 실행함으로써 얼마나 많은 환자가 다양한 포함 기준 및 제외 기준을 충족하는지 알 수 있게 한다.
세 번째 전략은 비슷하게 한 클래스의 질문 a class of questions에 초점을 맞추지만, 일단 작동하기 시작하면 그 클래스에 속한 모든 의문점에 대해 광범위하고 철저하게 모든 근거를 남김없이 생성하려고 시도한다. 사용자는 다양한 인터페이스를 통해 필요에 따라 (미리 생성된) 근거를 탐색할 수 있다. 한 예로 우울증 치료의 영향에 대한 OHDSI 연구가 있다. (Schuemie, Ryan, et al. 2018) 이 연구에서 모든 우울증 치료는 4개의 큰 관찰 데이터베이스에서 큰 규모의 임상결과 집합에 대해 비교된다. 광범위한 연구 진단과 함께 경험적으로 보정된 17,718개의 위험비hazard ratio를 포함한 전체 결과는 대화형 웹 앱에서 이용할 수 있다.37
8.3 ATLAS
ATLAS는 CDM 형식으로 표준화된 환자 수준 관찰 데이터에 대한 분석 설계와 실행을 도와주는 OHDSI 커뮤니티에서 개발한 무료 웹 기반 도구이다. ATLAS는 OHDSI WebAPI와 함께 웹 애플리케이션으로 배포되며 일반적으로 Apache Tomcat에서 호스팅 된다. 실시간 분석을 수행하려면 CDM에 있는 환자 수준 데이터에 액세스해야 하므로 일반적으로 조직의 방화벽 내부에 설치된다. 그러나 공용 ATLAS38도 있으며, 이 ATLAS 인스턴스는 몇 개의 소규모 시뮬레이션 데이터 세트에만 액세스할 수 있지만, 여전히 테스트와 훈련을 포함한 여러 용도로 사용할 수 있다. ATLAS의 공개 인스턴스를 사용하여 효과 추정 또는 예측 연구를 완전히 정의하고, 연구를 실행하기 위한 R 코드를 자동으로 생성할 수도 있다. 이 코드는 ATLAS와 WebAPI를 설치할 필요 없이 사용 가능한 CDM이 있는 모든 환경에서 실행될 수 있다.
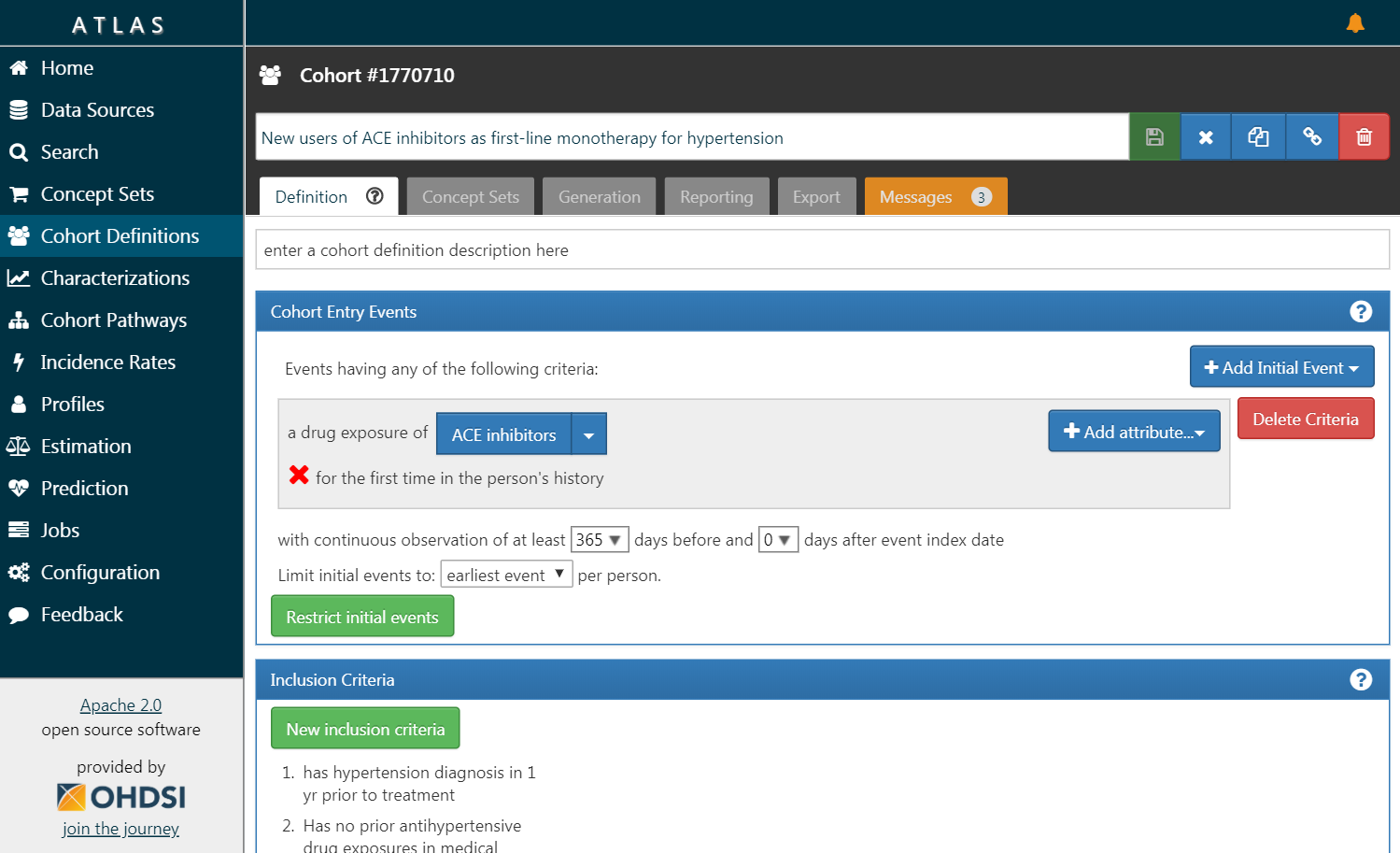
Figure 8.3: ATLAS 사용자 인터페이스
ATLAS 스크린샷은 그림 8.3에 제공된다. 왼쪽에는 ATLAS에서 제공하는 다양한 기능을 보여주는 내비게이션 바가 있다:
- Data Sources
- 데이터 원천Data sources은 ATLAS 플랫폼 내에서 구성한 각 데이터 원본에 대해 기술적이고 표준화된 보고 기능을 제공한다. 이 기능은 대규모 분석 전략을 사용한다. 모든 기술 통계량은 사전에 계산된 것이다. 데이터 출처는 11장에서 논한다.
- Vocabulary Search
- ATLAS는 OMOP 표준용어집을 검색하고 탐색하여 그 용어 안에 존재하는 개념 concept과 데이터 원천에 대한 표준 분석에서 해당 concept을 적용하는 방법을 이해할 수 있는 기능을 제공한다. 이 특성은 5장에서 논한다.
- Concept Sets
- 개념 집합 concept set은 표준화된 분석에서 사용할 concept set을 식별하는 데 사용할 수 있는 논리 표현식의 집합을 만들 수 있게 한다. concept set은 단순한 코드나 값 리스트보다 더 정교하게 만들어진다. concept set은 사용자가 용어 계층에 관련 concept을 포함하거나 배제할 수 있도록 하는 논리적 지표와 함께 표준화된 용어에서 나온 여러 concept으로 구성되어 있다. 용어를 검색하고, concept set을 식별하며, concept set을 해결하기 위해 사용할 논리를 명시하는 것은 분석 계획에서 흔히 접하는 모호한 의학 언어를 명확히 정의할 수 있게 하는 강력한 메커니즘을 제공한다. 이러한 concept set은 ATLAS 내에 저장한 다음 코호트 정의 또는 분석 규격의 일부로 분석 내내 사용할 수 있다.
- Cohort Definitions
- 코호트 정의 Cohort Definition는 일정 기간 하나 이상의 기준을 충족하는 일련의 사람을 구성할 수 있게 하며, 이러한 코호트는 이후 모든 분석 시 입력의 기초basis of input가 된다. 이 특성은 10장에서 논한다.
- Characterizations
- 특성은 당신이 정의한 하나 이상의 코호트를 보고 그 환자군에 대한 특성을 요약할 수 있는 분석 기능이다. 이 기능은 실시간 쿼리 전략을 사용하며, 11장에서 논한다.
- Cohort Pathways
- 코호트 경로(Cohort pathways)는 하나 이상의 인구집단 내에서 발생하는 임상 사건의 순서를 살펴볼 수 있는 분석 도구이다. 이 기능은 실시간 쿼리 전략을 사용하며, 11장에서 논한다.
- Incidence Rates
- 발생률은 관심 인구 (코호트) 내에서 임상 결과의 발생률을 추정할 수 있는 도구이다. 이 기능은 실시간 쿼리 전략을 사용하며, 11장에서 논한다.
- Profiles
- 프로파일은 개별 환자에 대해 종적 관찰 데이터를 탐색하여 특정 개인 내에서 일어나는 일을 요약할 수 있는 도구이다. 이 기능은 실시간 쿼리 전략을 사용한다.
- Population Level Estimation
- 추정은 비교 코호트 설계를 사용하여 인구 수준 효과 추정 연구를 정의할 수 있는 기능이며, 여기서 하나 이상의 대상 코호트와 비교 코호트 간의 비교를 통해 일련의 결과에 대해 탐색할 수 있다. 이 기능은 코딩이 필요하지 않으므로 실시간 쿼리 전략을 구현한다고 말할 수 있으며, 12장에서 논의한다.
- Patient Level Prediction
- 예측은 주어진 대상 노출 군 내에서 임상 결과를 예측할 수 있는 환자 수준 예측 분석을 수행하기 위해 기계 학습 알고리즘을 적용할 수 있는 기능이다. 이 기능은 코딩이 필요하지 않으므로 실시간 쿼리 전략을 구현한다고 할 수 있으며, 13장에서 논한다.
- Jobs
- WebAPI를 통해 실행 중인 프로세스의 상태를 탐색하려면 이 기능을 선택하라. 각각의 작업 job은 종종 코호트를 생성하거나코호트 특성 보고서를 생성하는 것과 같은 장기 실행 과정일 수 있다.
- Configuration
- 원천 구성 섹션에 구성된 데이터 원천을 검토하려면 구성 메뉴 항목을 선택하라.
- Feedback
- 피드백 링크는 ATLAS의 이슈 로그로 이동 시켜 새로운 이슈를 기록하거나 기존 이슈를 검색할 수 있도록 해준다. 새로운 기능이나 개선사항에 대한 아이디어가 있다면, 이것은 개발 커뮤니티에 대한 참고 사항이기도 하다.
8.3.1 보안
ATLAS와 WebAPI는 전체 플랫폼 내의 기능 또는 데이터 원천에 대한 액세스를 제어하기 위한 세분화된 보안 모델을 제공한다. 이 보안 시스템은 Apache Shiro 라이브러리를 활용하여 구축된다. 보안 시스템에 대한 추가 정보는 온라인 WebAPI 보안 위키에서 찾을 수 있다.39
8.3.2 설명서
ATLAS에 대한 설명서는 ATLAS GitHub repository wiki.40 에 있다. 이 위키에는 온라인 비디오 튜토리얼에 대한 링크뿐만 아니라 다양한 애플리케이션 기능에 대한 정보가 포함되어 있다.
8.4 연구방법론 라이브러리 Methods Library
OHDSI Methods Library는 그림 8.4에 표시된 오픈 소스 R 패키지의 모음이다.

Figure 8.4: OHDSI 연구방법론 라이브러리의 패키지
패키지는 완전한 관찰 연구를 수행하기 위해 함께 사용할 수 있는 R 기능을 제공하며, CDM의 데이터에서 시작하여 결과 추정치와 이를 뒷받침하는 통계, 수치 및 표를 제공한다. 패키지는 CDM의 관찰 데이터와 직접 상호작용하며, 단순히 9장에서 설명한 대로 완전한 사용자 정의 분석에 대한 플랫폼 간 호환성을 제공하는 데 사용하거나, 인구 특성화를 위한 고급 표준화 분석 (11장 참조), 인구 수준 효과 추정 (12장 참조) 및 환자 수준 예측 (13장 참조)을 제공할 수 있다. 연구방법론 라이브러리는 (이전 또는 진행 중인 연구에서 학습한) 투명성, 재현성, 그뿐만 아니라 “특정 맥락에서 방법론의 작동 특성 operating characteristics 측정” 및 이어지는 “methods로부터 생성된 측정치의 경험적 교정 empirical calibration”과 같은 관찰 데이터 및 관찰 연구 설계의 사용을 위한 모범 사례를 지원한다.
연구방법론 라이브러리는 이미 발표된 많은 임상 연구 (Boland et al. 2017; Duke et al. 2017; Ramcharran et al. 2017; Weinstein et al. 2017; Wang et al. 2017; Ryan et al. 2017; Ryan et al. 2018; Vashisht et al. 2018; Yuan et al. 2018; Johnston et al. 2019)와 방법론 연구에 사용되어 왔다. (Schuemie et al. 2014; Schuemie et al. 2016; Reps et al. 2018; Tian, Schuemie, and Suchard 2018; Schuemie, Hripcsak, et al. 2018; Schuemie, Ryan, et al. 2018; Reps, Rijnbeek, and Ryan 2019) 연구방법론 라이브러리에서 방법론 구현의 타당성은 17장에 설명되어 있다.
8.4.1 대규모 분석 지원
모든 패키지에 통합된 한 가지 주요 특징은 많은 분석을 효율적으로 실행할 수 있는 능력이다. 예를 들어 인구 수준 추정을 수행할 때 CohortMethod 패키지는 다양한 분석 설정을 사용하여 많은 노출 exposure 및 결과 outcome에 대한 효과 크기 추정치 effect-size estimates를 계산할 수 있도록 하며, 패키지는 필요한 모든 중간 및 최종 데이터 세트를 계산하는 최적의 방법을 자동으로 선택한다. “공변량 추출 extraction of covariates”이나 하나의 대상군-비교군 쌍 target-comparator pair과 복수의 결과에 사용되는 “성향 모델 맞춤 fitting a propensity model”과 같이 재사용할 수 있는 단계는 한 번만 실행된다. 가능한 경우 계산 자원의 사용을 극대화하기 위해 병렬처리 연산이 가능하다.
이러한 효율적 계산은 대규모 분석을 가능하게 하여 한꺼번에 많은 질문에 답할 수 있으며, 또한 제어 가설 (예를 들어, 음성대조군 negative controls)을 포함해 방법론의 작동 특성을 측정하고 18장에 기술된 경험적 교정을 수행하는 데 필수적이다.
8.4.2 빅데이터 지원
연구방법론 라이브러리는 또한 매우 큰 데이터베이스에 대해 실행하고 대량의 데이터를 포함하는 계산을 수행할 수 있도록 설계되었다. 이는 다음과 같은 세 가지 방법으로 달성되었다:
- 대부분의 데이터 조작은 데이터베이스 서버에서 수행된다. 분석은 일반적으로 데이터베이스에 있는 전체 데이터의 극히 일부만을 필요로 하며 연구방법론 라이브러리는 SqlRender 및 DatabaseConnector 패키지를 통해 서버에서 고급 작업을 수행하여 관련 데이터를 사전 처리하고 추출할 수 있도록 한다.
- 대용량 로컬 데이터 객체는 메모리 효율적인 방식으로 저장된다. 로컬 시스템으로 다운로드되는 데이터의 경우 연구방법론 라이브러리는 ff 패키지를 사용하여 대용량 데이터 객체를 저장하고 작업한다. 이것은 우리가 메모리를 직접적으로 사용하는 것보다 훨씬 더 큰 데이터로 작업할 수 있게 해준다.
- 필요한 곳에 고성능 컴퓨팅을 적용한다. 예를 들어, Cyclops 패키지는 대량의 변수와 관측치로 인해 다른 방법으로는 할 수 없는 대규모 회귀 분석을 수행할 수 있는 매우 효율적인 회귀 엔진을 구현했으며 연구방법론 라이브러리 전체에서 이 엔진을 사용할 수 있다.
8.4.3 문서화
R은 패키지를 문서화하는 표준화된 방법을 제공한다. 각 패키지에는 패키지에 포함된 모든 기능과 데이터 세트를 문서화하는 패키지 설명서가 있다. 모든 패키지 매뉴얼은 연구방법론 라이브러리 웹 사이트43를 통해 패키지 GitHub 온라인 저장소를 통해 사용할 수 있으며 CRAN을 통해 사용할 수 있는 패키지의 경우는 CRAN에서 찾을 수 있다. 또한, R 내에서 물음표를 사용하여 패키지 설명서를 참조할 수 있다. 예를 들어 DatabaseConnector 패키지를 로드한 후 ?connect 명령을 입력하면 “연결 connect” 기능에 대한 문서가 나타난다.
패키지 설명서 외에도 많은 패키지가 vignette를 제공한다. Vignettes는 특정 작업을 수행하기 위해 어떻게 패키지를 사용할 수 있는지 설명하는 긴 형식의 문서다. 예를 들어, 하나의 vignette44은 CohortMethod 패키지를 사용하여 여러 가지 분석을 효율적으로 수행하는 방법을 설명한다. 또한 Vignettes는 연구방법론 라이브러리 웹 사이트, 패키지 GitHub 저장소를 통해 찾을 수 있으며, CRAN을 통해 이용할 수 있는 패키지의 경우 CRAN에서 찾을 수 있다. Vignettes는 연구방법론 라이브러리 웹 사이트를 통해 패키지 GitHub 온라인 저장소를 통해 사용할 수 있으며 CRAN을 통해 사용할 수 있는 패키지의 경우 CRAN에서 찾을 수 있다.
8.4.4 시스템 요구 사항
시스템 요구 사항을 논의할 때 두 가지 컴퓨팅 환경을 고려해야 한다: 데이터베이스 서버 및 분석 워크스테이션
데이터베이스 서버는 의료 데이터를 CDM 형식으로 보관해야 한다. 연구방법론 라이브러리는 전통적인 데이터베이스 시스템 (PostgreSQL, Microsoft SQL Server, 그리고 Oracle), 병렬 데이터 웨어하우스 (Microsoft APS, IBM Netezza, 그리고 Amazon RedShift) 및 빅데이터 플랫폼 (Impala를 통한 Hadoop, 그리고 Google BigQuery)을 포함한 광범위한 데이터베이스 관리 시스템을 지원한다.
분석 워크스테이션은 연구방법론 라이브러리가 설치되어 실행되는 곳이다. 이것은 누군가의 랩탑과 같은 로컬 시스템이나 RStudio Server를 실행하는 원격 서버일 수 있다. 모든 경우에, R은 RStudio와 함께 설치되어야 한다. 연구방법론 라이브러리는 또한 Java가 설치되어야 한다. 분석 워크스테이션은 데이터베이스 서버에 연결할 수 있어야 하며, 특히 이 사이의 방화벽은 데이터베이스 서버 접근 포트를 워크스테이션에 개방해야 한다. 일부 분석은 계산 집약적일 수 있으므로 여러 개의 처리 코어와 충분한 메모리를 갖는 것이 분석 속도를 높이는 데 도움이 될 수 있다. 적어도 4개의 코어와 16GB의 메모리를 가질 것을 추천한다.
8.4.5 설치 방법
다음은 OHDSI R 패키지를 실행하는 데 필요한 환경을 설치하는 단계다. 다음 네 가지를 설치해야 한다:
- R은 통계 컴퓨팅 환경이다. 그것은 주로 명령어 인터페이스인 기본 사용자 인터페이스와 함께 제공된다.
- RTools는 Windows에서 원천으로부터 R 패키지를 만드는 데 필요한 프로그램의 모음이다.
- RStudio는 R을 사용하기 쉽게 하는 통합 개발 환경 Integrated Development Environment(IDE)이다. 여기에는 코드 편집기, 디버깅 및 시각화 도구가 포함되어 있다. 사용하기 편한 유저인터페이스를 원한다면 사용하기를 권한다.
- Java는 OHDSI R 패키지의 일부 구성 요소 (예를 들어, 데이터베이스에 연결하는 데 필요한 구성 요소)를 실행하는 데 필요한 컴퓨팅 환경이다.
아래에서는 Windows 환경에 이러한 각 항목을 설치하는 방법에 관해 설명한다.
R 설치하기
- https://cran.r-project.org/으로 이동하여, “Download R for Windows”를 클릭 후 “base”를 클릭한 다음 그림 8.5에 표시된 다운로드 링크를 클릭하라.
Figure 8.5: CRAN으로부터 R 다운로드
- 다운로드가 완료된 후 설치 프로그램을 실행하라. 다음 두 가지 예외를 제외하고 모든 곳에서 기본 옵션을 사용하라. 첫째, 프로그램 파일 폴더에 설치하지 않는 것이 좋다. 대신 R을 그림 8.6과 같이 C 드라이브의 하위 폴더로 만들라. 둘째, R과 Java 간의 아키텍처 차이로 인한 문제를 방지하려면 그림 8.7과 같이 32-bit 아키텍처를 비활성화하라.
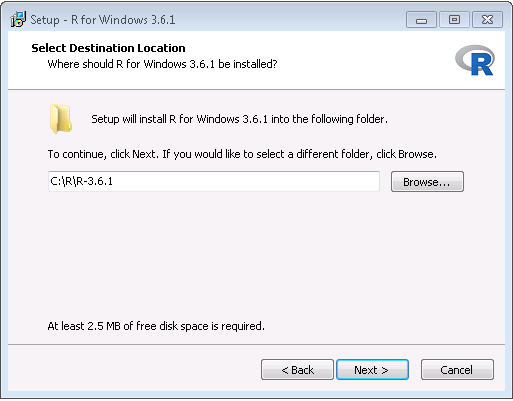
Figure 8.6: R의 대상 폴더 설정하기.
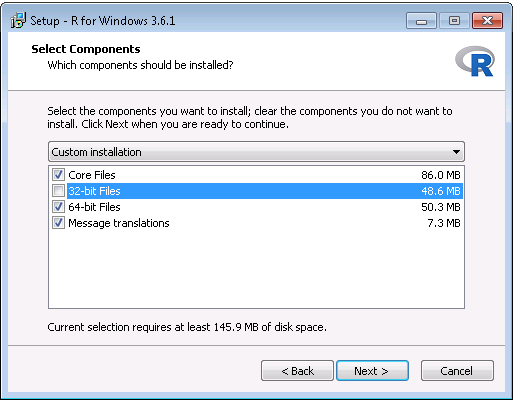
Figure 8.7: 32-bit 버전의 R을 사용하지 않도록 설정하기.
완료되면 시작 메뉴에서 R을 선택할 수 있어야 한다.
RTools 설치하기
https://cran.r-project.org/으로 이동하여 “Windows용 R 다운로드”를 클릭한 다음 “Rtools”를 클릭하고 다운로드할 최신 버전의 RTools를 선택하라.
다운로드가 완료된 후 설치 프로그램을 실행하라. 어디에서나 기본 옵션을 선택하라.
RStudio 설치하기
- https://www.rstudio.com/으로 이동하여, “Download RStudio”을 선택 (또는 “RStudio”에서 “Download” 버튼을 선택)하고, 무료 버전을 선택한 후, 그림 8.8과 같이 Windows용 설치 프로그램을 다운로드하라.
Figure 8.8: RStudio 다운로드
- 다운로드한 후, 설치 관리자를 시작하고, 모든 곳에서 기본 옵션을 선택하라.
Java 설치하기
- https://java.com/en/download/manual.jsp으로 이동하여, 그림 8.9와 같이 Windows 64-bit installer를 선택하라. 32-bit 버전의 R을 설치한 경우 반드시 다른 32-bit 버전의 Java도 설치해야 한다.

Figure 8.9: Java 다운로드
- 다운로드한 후 설치 프로그램을 실행하라.
설치 검수하기
이제 시작할 준비를 해야 하지만, 그 전에 확실히 해야 한다. RStudio를 시작하고 및 아래의 내용을 입력하자.
install.packages("SqlRender")
library(SqlRender)
translate("SELECT TOP 10 * FROM person;", "postgresql")## [1] "SELECT * FROM person LIMIT 10;"이 기능은 Java를 사용하기 때문에, 만약 모든 것이 잘 된다면, R과 Java가 모두 올바르게 설치되었다는 것을 알 수 있다!
또 다른 테스트는 원천 패키지를 제대로 구축할 수 있는지 확인하는 것이다. 다음 R 코드를 실행하여 OHDSI GitHub 저장소에서 CohortMethod 패키지를 설치하라:
install.packages("drat")
drat::addRepo("OHDSI")
install.packages("CohortMethod")8.5 설치 전략
ATLAS 및 연구방법론 라이브러리를 포함한 전체 OHDSI 도구 스택을 기관에 모두 설치하는 것은 어려운 작업이다. 의존성 높은 구성 요소를 많이 고려해야 하고, 설정해야 할 환경이 많다. 이 때문에 두 이니셔티브 (Broadsea와 Amazon Web Services(AWS))는 일부 가상화 형태를 이용해 전체 스택을 하나의 패키지로 설치할 수 있는 통합 설치 전략을 개발했다.
8.5.1 Broadsea
Broadsea45는 Docker 컨테이너 기술을 사용한다.46 OHDSI 도구는 라이브러리 간 의존성과 함께 Docker Image라는 단일 휴대용 이진 파일로 패키징 된다. 그러면 이 이미지는 Docker 엔진 서비스에서 실행되고, 모든 소프트웨어가 설치되어 실행 준비가 된 가상 시스템 virtual machine을 생성할 수 있다. Docker 엔진은 Microsoft Windows, MacOS, Linux를 포함한 대부분의 운영 체제에 사용할 수 있다. Broadsea Docker 이미지에는 연구방법론 라이브러리와 ATLAS를 포함한 주요 OHDSI 도구가 포함되어 있다.
8.5.2 Amazon AWS
Amazon은 버튼 클릭 한 번으로 OHDSI 환경을 AWS 클라우드 컴퓨팅 환경에서 바로 인스턴스화할 수 있는 두 가지 환경, 즉 OHDSI-in-a-Box47와 OHDSIonAWS.48을 준비했다.
OHDSI-in-a-Box는 특별히 학습 환경으로 만들어졌으며, OHDSI 커뮤니티에서 제공하는 대부분의 튜토리얼에 사용된다. 그것은 많은 OHDSI 도구, 샘플 데이터 세트, RStudio 및 기타 지원 소프트웨어를 저렴한 단일 Windows 가상 머신에 포함했다. PostgreSQL 데이터베이스는 CDM을 저장하고 ATLAS의 중간 결과를 저장하는 데 사용된다. OMOP CDM 데이터 매핑과 ETL 도구도 OHDSI-in-a-Box에 포함되어 있다. OHDSI-in-a-Box 아키텍처는 그림 8.10에 나타나 있다.
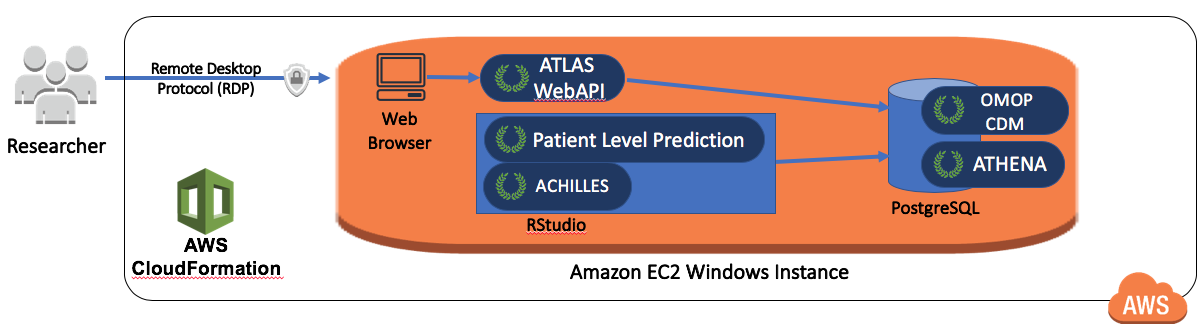
Figure 8.10: OHDSI-in-a-Box용 Amazon Web Services 아키텍처
OHDSIonAWS는 기관이 그들의 데이터 분석을 수행하는 데 사용할 수 있는 엔터프라이즈급, 다중 사용자, 확장할 수 있는 내결함성 OHDSI 환경을 위한 참조 아키텍처이다. 여기에는 몇 가지 샘플 데이터 세트가 포함되어 있으며 기관의 실제 의료 데이터를 자동으로 적재할 수도 있다. 데이터는 OHDSI 도구에 의해 지원되는 Amazon Redshift 데이터베이스 플랫폼에 배치된다. ATLAS의 중간 결과는 PostgreSQL 데이터베이스에 저장된다. 프론트 엔드에서 사용자는 웹 인터페이스(leveraging RStudio Server)를 통해 ATLAS와 RStudio에 접근할 수 있다. RStudio에는 OHDSI 연구방법론 라이브러리가 이미 설치되어 있으며, 데이터베이스에 연결하는 데 사용할 수 있다. OHDSIonAWS를 배포하는 자동화 도구는 오픈 소스로서 기관의 관리 도구와 모범 사례를 포함하도록 사용자 정의할 수 있다. OHDSIonAWS에 대한 아키텍처는 그림 8.11에 설명되어 있다.
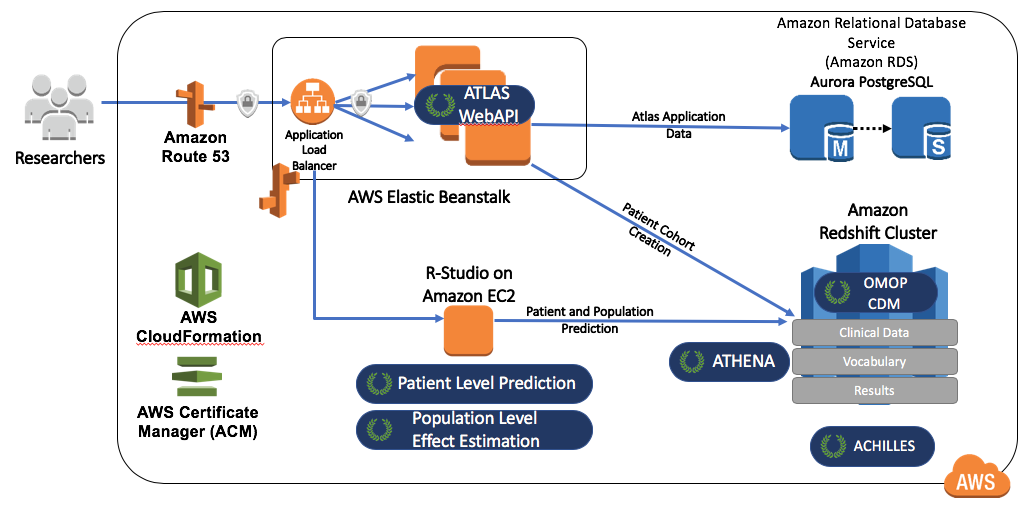
Figure 8.11: OHDSIonAWS를 위한 Amazon Web Services 아카이브
8.6 요약
- 다음과 같은 방식으로 CDM 데이터에 대한 분석을 수행할 수 있다.
- 사용자가 직접 분석코드 작성
- OHDSI 연구방법론 라이브러리에서 R 패키지를 사용하여 코드 작성
- 분석 코드 작성 없이 대화형 분석 플랫폼 ATLAS를 사용
- OHDSI 도구는 다양한 분석 전략을 사용한다.
- 단일 연구
- 실시간 쿼리
- 대규모 분석
- 대부분의 OHDSI 분석 도구가 다음에 내장되어 있다.
- 대화형 분석 플랫폼 ATLAS
- OHDSI 연구방법론 라이브러리 R 패키지
- OHDSI 도구의 구축을 촉진하는 몇 가지 전략이 존재한다.
References
Boland, M. R., P. Parhi, L. Li, R. Miotto, R. Carroll, U. Iqbal, P. A. Nguyen, et al. 2017. “Uncovering exposures responsible for birth season - disease effects: a global study.” J Am Med Inform Assoc, September.
Duke, J. D., P. B. Ryan, M. A. Suchard, G. Hripcsak, P. Jin, C. Reich, M. S. Schwalm, et al. 2017. “Risk of angioedema associated with levetiracetam compared with phenytoin: Findings of the observational health data sciences and informatics research network.” Epilepsia 58 (8): e101–e106.
Johnston, S. S., J. M. Morton, I. Kalsekar, E. M. Ammann, C. W. Hsiao, and J. Reps. 2019. “Using Machine Learning Applied to Real-World Healthcare Data for Predictive Analytics: An Applied Example in Bariatric Surgery.” Value Health 22 (5): 580–86.
Ramcharran, D., H. Qiu, M. J. Schuemie, and P. B. Ryan. 2017. “Atypical Antipsychotics and the Risk of Falls and Fractures Among Older Adults: An Emulation Analysis and an Evaluation of Additional Confounding Control Strategies.” J Clin Psychopharmacol 37 (2): 162–68.
Reps, J. M., P. R. Rijnbeek, and P. B. Ryan. 2019. “Identifying the DEAD: Development and Validation of a Patient-Level Model to Predict Death Status in Population-Level Claims Data.” Drug Saf, May.
Reps, J. M., M. J. Schuemie, M. A. Suchard, P. B. Ryan, and P. R. Rijnbeek. 2018. “Design and implementation of a standardized framework to generate and evaluate patient-level prediction models using observational healthcare data.” Journal of the American Medical Informatics Association 25 (8): 969–75. doi:10.1093/jamia/ocy032.
Ryan, P. B., J. B. Buse, M. J. Schuemie, F. DeFalco, Z. Yuan, P. E. Stang, J. A. Berlin, and N. Rosenthal. 2018. “Comparative effectiveness of canagliflozin, SGLT2 inhibitors and non-SGLT2 inhibitors on the risk of hospitalization for heart failure and amputation in patients with type 2 diabetes mellitus: A real-world meta-analysis of 4 observational databases (OBSERVE-4D).” Diabetes Obes Metab 20 (11): 2585–97.
Ryan, P. B., M. J. Schuemie, D. Ramcharran, and P. E. Stang. 2017. “Atypical Antipsychotics and the Risks of Acute Kidney Injury and Related Outcomes Among Older Adults: A Replication Analysis and an Evaluation of Adapted Confounding Control Strategies.” Drugs Aging 34 (3): 211–19.
Schuemie, M. J., G. Hripcsak, P. B. Ryan, D. Madigan, and M. A. Suchard. 2016. “Robust empirical calibration of p-values using observational data.” Stat Med 35 (22): 3883–8.
Schuemie, M. 2018. “Empirical confidence interval calibration for population-level effect estimation studies in observational healthcare data.” Proc. Natl. Acad. Sci. U.S.A. 115 (11): 2571–7.
Schuemie, M. J., P. B. Ryan, W. DuMouchel, M. A. Suchard, and D. Madigan. 2014. “Interpreting observational studies: why empirical calibration is needed to correct p-values.” Stat Med 33 (2): 209–18.
Schuemie, M. J., P. B. Ryan, G. Hripcsak, D. Madigan, and M. A. Suchard. 2018. “Improving reproducibility by using high-throughput observational studies with empirical calibration.” Philos Trans A Math Phys Eng Sci 376 (2128).
Tian, Y., M. J. Schuemie, and M. A. Suchard. 2018. “Evaluating large-scale propensity score performance through real-world and synthetic data experiments.” Int J Epidemiol 47 (6): 2005–14.
Vashisht, R., K. Jung, A. Schuler, J. M. Banda, R. W. Park, S. Jin, L. Li, et al. 2018. “Association of Hemoglobin A1c Levels With Use of Sulfonylureas, Dipeptidyl Peptidase 4 Inhibitors, and Thiazolidinediones in Patients With Type 2 Diabetes Treated With Metformin: Analysis From the Observational Health Data Sciences and Informatics Initiative.” JAMA Netw Open 1 (4): e181755.
Wang, Y., M. Desai, P. B. Ryan, F. J. DeFalco, M. J. Schuemie, P. E. Stang, J. A. Berlin, and Z. Yuan. 2017. “Incidence of diabetic ketoacidosis among patients with type 2 diabetes mellitus treated with SGLT2 inhibitors and other antihyperglycemic agents.” Diabetes Res. Clin. Pract. 128 (June): 83–90.
Weinstein, R. B., P. Ryan, J. A. Berlin, A. Matcho, M. Schuemie, J. Swerdel, K. Patel, and D. Fife. 2017. “Channeling in the Use of Nonprescription Paracetamol and Ibuprofen in an Electronic Medical Records Database: Evidence and Implications.” Drug Saf 40 (12): 1279–92.
Yuan, Z., F. J. DeFalco, P. B. Ryan, M. J. Schuemie, P. E. Stang, J. A. Berlin, M. Desai, and N. Rosenthal. 2018. “Risk of lower extremity amputations in people with type 2 diabetes mellitus treated with sodium-glucose co-transporter-2 inhibitors in the USA: A retrospective cohort study.” Diabetes Obes Metab 20 (3): 582–89.