Chapter 13 환자 수준 예측
Chapter leads: Peter Rijnbeek & Jenna Reps
임상 의사 결정 clinical decision making이란 임상 의사가 알 수 있는 환자의 병력에 대한 정보와 현재 임상지침에 따라 진단 또는 치료 경로를 추론해야 하는 복잡한 일이다. 임상 예측 모델은 이러한 의사 결정 과정을 지원하기 위해 개발되었으며 광범위한 전문 분야에서 임상 실무에 사용된다. 이러한 모델은 인구 통계학적 정보, 질병력 및 치료력과 같은 환자 특성을 조합하여 이를 기반으로 진단 또는 예후 결과를 예측한다.
임상 예측 모델을 설명하는 출판물의 수가 지난 10년 동안 많이 증가했다. 현재 사용되는 대부분의 모델은 소규모 데이터를 이용하여 학습하며, 적은 수의 환자 특성만 고려한다. 이처럼 소표본이고 그래서 낮아지는 통계적 검정력으로 인해 데이터 분석가는 엄격한 가정 하에 모델링을 수행하게 된다. 환자 특성 또는 사용되는 변수 선택은 현재의 전문적 지식에 강하게 의존한다. 이는 환자가 매우 다양한 자료, 디지털 트레일 digital trail을 생성하는 현대 의학의 현실과 크게 대조되며, 이는 다양한 의료 전문가가 협력할 수 있는 방향이 아니다. 현재의 의학 관리 체계는 전자 의무 기록 Electronic Health Records(EHR)에 엄청난 양의 환자의 개인별 정보를 생성 및 저장하고 있다. 여기에는 진단, 약물치료, 실험실 검사 결과와 같은 정형화된 데이터와 임상적 기술 clinical narratives에 포함된 비정형화된 데이터가 포함되어 있다. 방대한 EHR으로부터 대량의 환자 데이터를 얻고 이것을 활용하여도 예측 정확도를 얼마나 얻을 수 있는지는 알 수 없다.
대규모 데이터 모음 분석을 위한 머신 러닝의 발전으로 이러한 유형의 데이터에 환자-수준 예측을 적용하는 데 관심이 높아졌다. 그러나 환자-수준 예측을 위한 많은 수의 출판물은 모델 개발 지침을 따르지 않아 광범위한 외적 타당도 external validation 수행에 실패하거나 또는 독립적인 연구자가 그 모델을 재현하고 외적 타당도를 검증하기 위한 세부사항을 제공하지 않는다. 이로 인해, 모델의 예측 성능을 공정하게 평가하기 어렵고 임상 실무에서 모델이 적절하게 사용될 가능성이 낮아진다. 표준화를 개선하기 위해 예측 모델을 개발하고 보고하는 모범 사례에 대한 지침을 자세히 설명하는 여러 논문이 작성되었다. 예를 들어, 개별 예측 또는 진단(TRIPOD)52 선언문에 다변량 예측 모델의 투명한 보고 Transparent Reporting는 예측 모델 개발 및 타당도 보고에 대한 명확한 권장 사항을 제공하고 투명성과 관련된 일부 우려를 해결한다.
OHDSI CDM을 통해 전례 없는 규모의 데이터를 균일하고 투명하게 분석할 수 있게 되어, 대규모의 환자별 예측 모델링이 현실이 되었다. CDM으로 표준화된 데이터베이스 네트워크가 증가하면서 전 세계의 다양한 의료 환경에서 모델의 외적 타당성 검증이 가능하게 되었다. 우리는 이런 환경이 치료의 질적 개선을 가장 필요로 하는 많은 수의 환자 공동체를 돌볼 수 있는 즉각적인 기회를 제공한다고 믿는다. 그러한 모델은 진정한 개인 맞춤형 의료를 제공할 수 있기 때문에, 환자의 예후를 크게 개선할 수 있게 될 것으로 믿는다.
이 장에서는 환자-수준 예측을 위한 OHDSI의 표준화된 프레임워크 (Reps et al. 2018) 를 설명하고 개발 및 타당도 검증을 위해 확립된 모범 사례를 구현하는 PatientLevelPrediction R 패키지에 관해 설명한다. 우리는 환자-수준 예측의 개발과 평가에 필요한 이론을 제공하는 것으로 시작하여 구현된 기계 학습 알고리즘에 대한 큰 그림 수준의 개요를 제공할 것이다. 그런 다음 예측 문제에 대한 예제에 대하여 논의하고 ATLAS 또는 사용자 정의 R 코드를 사용하여 예측 문제를 정의하고 실행하는 단계별 지침을 제공할 것이다. 마지막으로 연구 결과를 널리 알리기 위한 Shiny 앱 사용법에 대해서 논의한다. 이는 최근 많이 각광 받는 인공 지능, 머신 러닝 모델을 코딩 전문성이 없는 일반 연구자가 구현하고 평가할 수 있음을 의미한다!
13.1 예측 문제
그림 13.1은 우리가 다루는 예측 문제를 보여준다. 연구 대상군 중 어떤 환자가 위험 노출 시간 동안에 어떤 결과를 경험할 것인지 특정 시점 (t = 0) 에서 예측하는 것을 목표로 한다. 예측은 해당 시점 (t = 0) 이전의 관찰 기간 observation window에서 관찰된 환자 정보만 사용하여 수행된다.
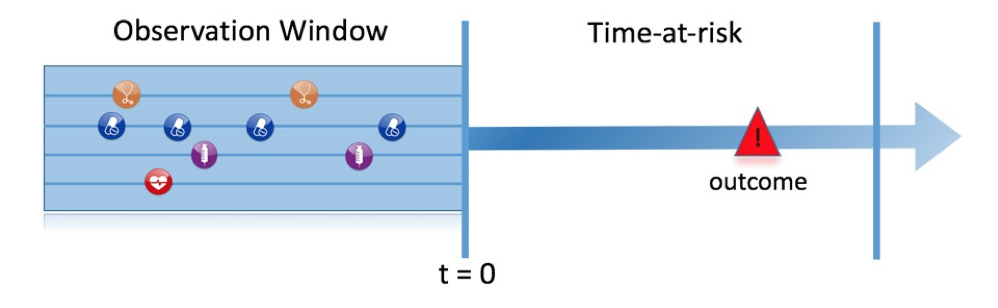
Figure 13.1: 예측 문제
표 13.1에서 볼 수 있듯이 예측 문제를 밝히려면 대상 코호트 target cohort의 t = 0, 결과 코호트 outcome cohort에 의해 예측하고자 하는 결과, 그리고 위험 노출 시간을 정의해야 한다. 표준 예측 질문을 다음과 같이 정의한다:
[대상 코호트, T]에서, 누가 [위험에 노출된 시간, t] 내에 [결과 코호트, O]가 발생하는가?
또한 개발하고자 하는 모델에 대해 디자인을 선택하고 내적 및 외적 타당도 검증을 수행할 관찰 데이터 모음을 결정해야 한다.
| 선택 | 설명 |
|---|---|
| 대상 코호트 Target cohort | 우리가 예측을 원하는 대상이 되는 환자군 |
| 결과 코호트 Outcome cohort | 예측을 원하는 결과 outcome 발생군 |
| 위험노출기간 Time-at-risk | t=0와 비교하여 결과 발생 여부를 살펴볼 기간 time window |
| 모델 Model | 머신 러닝 알고리즘 및 예측 모델 개발시 사용할 변수 |
이 개념적 프레임워크는 다음과 같은 모든 유형의 예측 문제에 적용된다, 예를 들면:
- 질병 발병 및 진행
- 구조: [질병 A]로 새로 진단된 환자 중, [진단 시점 t] 내에 [또 다른 질병이나 합병증, B] 이 생길 사람은 누구인가?
- 예제: 새로 진단된 심방세동 환자 중 향후 3년 이내에 허혈성 뇌졸중이 발생할 사람은 누구인가?
- 치료 선택
- 구조: [치료 1] 또는 [치료 2]로 치료한 [대상 질병, D]에 걸린 환자 중 [치료 1]로 치료받은 환자는 누구인가?
- 예제: 와파린 또는 리바록사반을 복용한 심방세동 환자 중 어떤 환자가 와파린을 복용했는가? (예를 들어 성향 모델의 경우)
- 치료 반응
- 구조: [치료 1]을 처음 사용하는 사람 중, 누가 [시간대 t]에서 [어떤 효과, E]를 경험했는가?
- 예제: 메트포민으로 치료받기 시작한 당뇨병 환자 중 어떤 환자가 3년 동안 메트포민을 유지하는가?
- 치료 안전
- 구조: [치료 1]을 처음 사용하는 사람 중 누가 [시간대 t]에서 [이상 반응 E]를 경험하게 되는가?
- 예제: 와파린을 처음 사용하는 사람 중 누가 1년 안에 위장관 출혈이 발생하는가?
- 치료 준수
- 구조: [치료 1]을 처음 사용하는 사람 중 누가 [시간대, t]에서 [준수 지표 수치]를 달성하는가?
- 예제: 메트포민으로 치료를 시작한 당뇨병 환자 중 어떤 환자가 1년 중 80% 이상의 복용 순응도를 보이는가?
13.2 데이터 추출
예측 모델을 만들 때 상태에 따라 분류된 예제 기반으로 공변량과 결과 상태 간의 관계를 유추하기 위하여 기계학습과 같은 지도 학습이라는 프로세스를 사용한다. 따라서, 대상 코호트에 있는 사람의 CDM에서 공변량을 추출하는 방법이 필요하며 그들의 결과 레이블을 얻을 필요가 있다.
공변량 (“예측변수”, “특징” 또는 “독립 변수”라고도 함)은 환자의 특성을 묘사한다. 공변량은 연령, 성별, 특정 질병 존재, 그리고 환자 기록에 있는 노출 코드 그리고 그 외 여러 가지가 될 수 있다. 공변량은 FeatureExtraction 패키지를 사용하여 구성되었고, 11장에 자세히 설명돼 있다. 예측을 위해 우리는 오직 대상 코호트에 들어오는 날짜 기준으로 환자의 이전 또는 그때의 데이터만 사용할 수 있다. 이 날짜를 인덱스 날짜라고 한다.
또한, 위험 노출 기간 time-at-risk(TAR) 동안 모든 환자의 결과 상태 (“라벨” 또는 “분류”라고도 함) 를 생성할 필요가 있다. 만약 결과가 위험에 노출된 시간 안에 발생하거나 그 결과 상태는 “양성”으로 정의된다.
13.2.1 데이터 추출 예제
표 13.2는 두 개의 코호트가 있는 COHORT 테이블에 대한 예를 보여준다. 코호트 정의 ID 1인 코호트는 대상 코호트 (예를 들어 “최근 심방세동 진단을 받은 사람”)이고 코호트 정의 ID 2는 결과 코호트 (예를 들어 “뇌졸중”)이다.
| COHORT_DEFINITION_ID | SUBJECT_ID | COHORT_START_DATE |
|---|---|---|
| 1 | 1 | 2000-06-01 |
| 1 | 2 | 2001-06-01 |
| 2 | 2 | 2001-07-01 |
표 13.3은 CONDITION_OCCURRENCE에 대한 예제이다. 개념(concept) ID 320128은 “본태성 고혈압”을 나타낸다.
| PERSON_ID | CONDITION_CONCEPT_ID | CONDITION_START_DATE |
|---|---|---|
| 1 | 320128 | 2000-10-01 |
| 2 | 320128 | 2001-05-01 |
이 예제 데이터를 기반으로, 위험에 노출된 시간이 인덱스 날짜 (대상 코호트 시작 날짜)의 다음 연도라고 가정하고 다음과 같이 공변량 및 결과 상태를 구성할 수 있다. Person ID가 1인 환자 (인덱스 날짜 이후에 발생한 질병)의 경우 “이전 해의 본태성 고혈압”을 나타내는 공변량은 값 0 (현재 아님)으로 하고, person ID가 2인 환자는 값 1 (현재 진행)을 갖는다. 유사하게, 결과 상태는 person ID가 1인 환자 (이 사람은 결과 코호트에 못 들어감)의 경우는 값 0을 갖고 person ID가 2인 환자 (인덱스 날짜 다음 1년 이내에 결과가 발생하였음)의 경우에는 값 1을 갖는다.
13.2.2 결측
관찰 의료 데이터는 데이터 누락 여부를 거의 반영하지 않는다. 이전의 예제에서, 우리는 person ID가 1인 환자가 인덱스 날짜 이전에 본태성 고혈압이 없었음을 관찰했다. 사실은 10년 전에 고혈압 진단을 받았으나, 해당 데이터베이스에 기록이 없었을 수도 있다.머신 러닝 알고리즘은 두 시나리오를 구분할 수 없고 사용 가능한 데이터 안에서 예측값을 대략 평가한다는 것을 인지해야 한다. (역자 주: 개인적으로는 결측 자체도 의미를 가진다고 생각한다.)
13.3 모델 적합
예측 모델을 적합할 때 상태에 따라 분류된 예제로부터 공변량과 관찰된 결과 상태 간의 관계를 알려고 노력한다. 만약 수축기 혈압과 이완기 혈압의 두 가지 공변량이 있다고 가정하면 그림 13.2와 같이 2차원 공간에서 그림으로 각 환자를 나타낼 수 있다. 이 그림에서 데이터를 나타내는 점의 모양은 환자의 결과 상태 (예를 들어, 뇌졸중) 를 나타낸다.
지도 학습 모델은 두 결과 분류 class를 최적으로 분리하는 결정 경계 decision boundary를 찾아내려고 노력할 것이다. 다른 지도 학습 기법은 다른 분류 결정 경계로 이어지고 분류 결정 경계의 복잡성에 영향을 줄 수 있는 하이퍼-파라미터 hyper-parameters가 종종 있다.
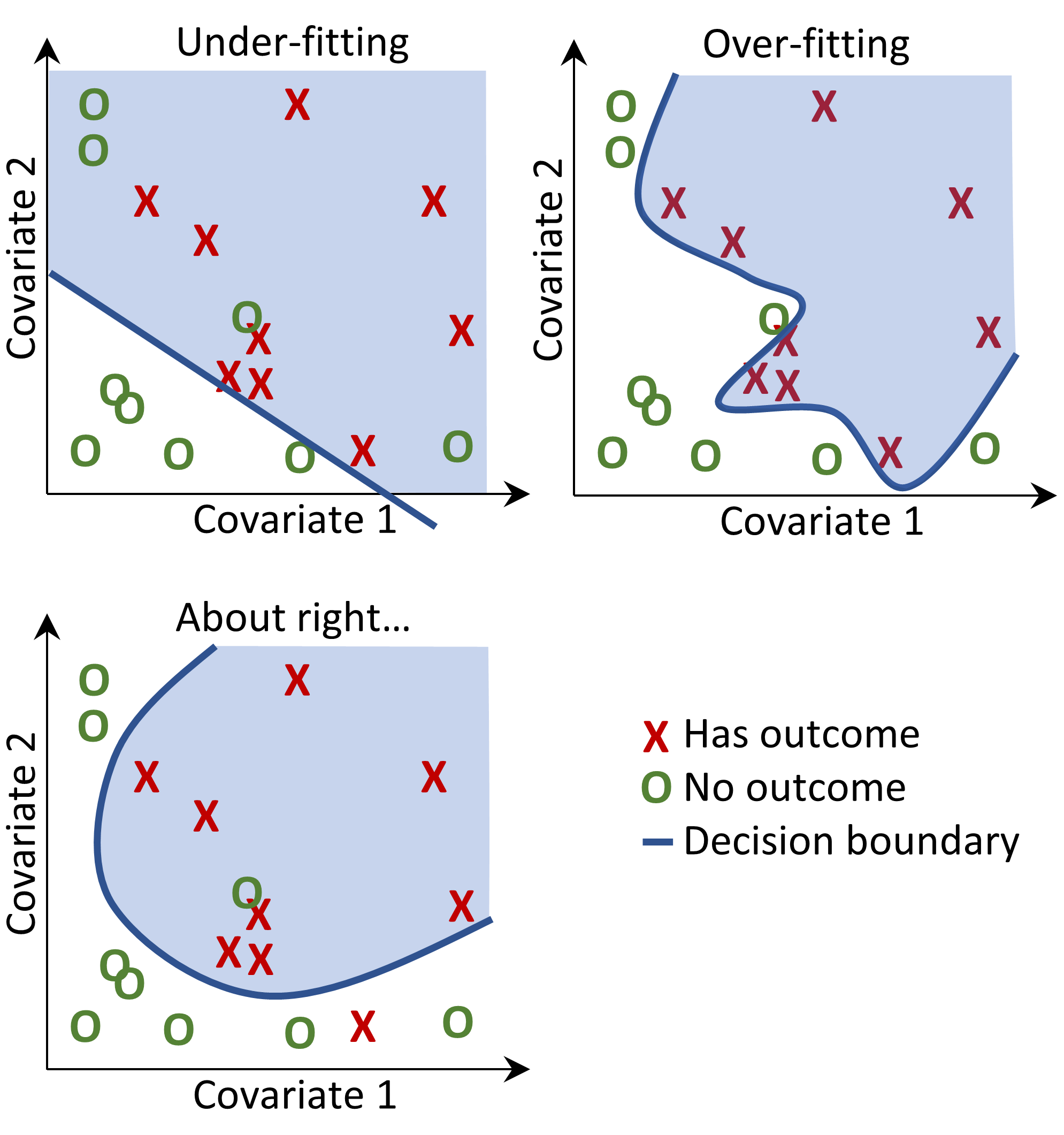
Figure 13.2: 결정 경계 Decision boundary
그림 13.2에서 세 가지 다른 결정 경계를 볼 수 있다. 그 경계는 새로운 데이터 포인트의 결과 상태를 추론하기 위해 사용되기도 한다. 새 데이터 포인트가 음영이 있는 영역에 포함되면 모델은 “결과가 있음”을 예측하고, 그렇지 않으면 “결과가 없음”으로 예측한다. 이상적으로는 결정 경계가 두 분류를 완벽하게 구분해야 한다. 그러나 너무 복잡한 모델은 데이터에 “과적합 overfit”할 위험이 있다. 이는 보이지 않는 데이터에 대한 모델의 일반화에 부정적인 영향을 줄 수 있다. 예를 들어, 데이터에 레이블이 없거나 잘못 지정된 데이터 포인트를 갖는 잡음 noise이 포함된 경우 해당 잡음에 예측 모델을 적합하고 싶지 않을 것이다. 그러므로 우리는 학습 데이터 training data를 갖고 완벽하게 판별하지는 않지만 “실제의” 복잡성을 반영하는 결정 경계를 정의하는 것을 선호 할 수 있다. 정규화 regularization와 같은 기술은 복잡성을 최소화하면서 모델 성능을 최대화하는 것을 목표로 한다.
각각의 지도 학습 알고리즘마다 의사 결정 경계를 학습하는 방법이 다르므로 어떤 알고리즘이 데이터에 가장 적합한지 간단하지 않다. No Free Lunch 정리에 따르면 모든 예측 문제에서 하나의 알고리즘이 언제나 다른 알고리즘보다 성능이 우수하지는 않다는 것을 알 수 있다. 따라서 환자-수준 예측 모델을 개발할 때 다양한 하이퍼-파라미터 설정으로 여러 개의 지도 학습 알고리즘을 사용하는 것이 좋다.
다음의 알고리즘은 PatientLevelPrediction 패키지에서 사용할 수 있다:
13.3.1 정규화된 로지스틱 회귀
Least Absolute Shrinkage and Selection Operator(LASSO) 로지스틱 회귀는 변수의 선형결합을 알 수 있는 일반화 선형 모델 generalized linear models에 속하고 로지스틱 함수는 결국 0과 1 사잇값으로 배치구조를 나타낸다. LASSO 정규화는 모델 학습 시 모델 복잡도에 따른 비용을 목적 함수 objective function에 추가한다. 이 비용은 계수의 선형 결합의 절댓값의 합이다. 모델은 이 비용을 최소화하면서 특징 선택 feature selection을 자동으로 수행한다. 우리는 대규모 정규화 로지스틱 회귀 분석을 수행하기 위하여 Cyclops(Cyclic coordinate descent for logistic, Poisson and survival analysis) 패키지를 사용한다.
| 파라미터 | 설명 | 주로 사용되는 설정 |
|---|---|---|
| 시작 분산Starting variance | 사전 분포에 대한 시작 분산. | 0.1 |
교차 검증에서 표본 외 out-of-sample 우도 likelihood를 최대화하여 분산이 최적화되므로 시작 분산은 결과 모델의 성능에 거의 영향을 미치지 않는다. 그러나 시작 분산이 최적값에서 너무 차이가 나면 모델 적합 시간이 길어질 수 있다.
13.3.2 Gradient Boosting Machines
Gradient boosting machines은 부스팅 앙상블 기법 boosting ensemble technique이며 프레임워크 안에서 다중 의사 결정 나무 multiple decision tree를 연결한다. 부스팅은 의사 결정 나무 tree를 반복적으로 추가하는 것이지만, 다음에 생성될 의사 결정 나무를 학습할 때 비용 함수 cost function에서 이전 의사 결정 나무에 의해 잘못 분류된 데이터 포인트에 더 많은 가중치를 추가한다. CRAN에서 제공하는 xgboost R 패키지로 수행된 gradient boosting framework를 효율적으로 구현하는 Extreme Gradient Boosting을 사용한다.
| 파라미터 | 설명 | 주로 사용되는 설정 |
|---|---|---|
| earlyStopRound | Stopping after rounds without improvement | 25 |
| learningRate | The boosting learn rate | 0.005,0.01,0.1 |
| maxDepth | Max levels in a tree | 4,6,17 |
| minRows | Min data points in a node | 2 |
| ntrees | Number of trees | 100,1000 |
13.3.3 랜덤 포레스트
랜덤 포레스트 Random forest는 다중 의사 결정 나무 multiple decision tree를 연결하는 배깅 앙상블 기법 bagging ensemble technique이다. 배깅의 기본 개념은 유사도가 낮은 분류기를 사용하여 유사도가 높은 분류기로 결합하여 과적합 가능성을 줄이는 것이다. 랜덤 포레스트 Random forest는 다중 의사 결정 나무를 학습하여 저장하는 것으로 하지만 각 나무에서 변수의 하위 집합만 사용하며 변수 하위 집합은 의사 결정 나무마다 다르다. 우리 패키지는 Python의 Sklearn 내 Random forest를 사용한다.
| 파라미터 | 설명 | 주로 사용되는 설정 |
|---|---|---|
| maxDepth | Max levels in a tree | 4,10,17 |
| mtries | Number of features in each tree | -1 = square root of total features,5,20 |
| ntrees | Number of trees | 500 |
13.3.4 K-최근접 이웃
K-최근접 이웃 K-nearest neighbors(KNN)은 몇 개의 거리 척도 distance metric를 사용하여 레이블이 지정되지 않은 새로운 데이터 포인트에 가장 가까운 K개의 레이블이 있는 데이터 포인트를 찾는 알고리즘이다. 새로운 데이터 포인트의 예측은 K-최근접의 레이블이 된 데이터 포인트의 가장 보편적인 분류이다. 모델에 새 데이터에 대한 예측을 수행하기 위해 레이블이 지정된 데이터가 필요하므로 KNN의 공유 제한이 있으며 데이터 사이트 간에 이 데이터를 공유할 수 없는 경우가 종종 있다. 여기에서는 대규모 KNN classifier인 OHDSI에서 개발된 BigKnn 패키지를 이용한다.
| 파라미터 | 설명 | Typical values |
|---|---|---|
| k | Number of neighbors | 1000 |
13.3.5 나이브 베이즈
나이브 베이즈 Naive Bayes 알고리즘은 클래스 변수의 값이 주어지는 모든 특징 사이의 조건부 독립성의 나이브 추정을 가진 베이즈 Bayes 이론을 적용한다. 클래스의 사전 배포와 데이터가 클래스에 속할 가능성에 기초하여, 사후 배포가 얻어진다. 나이브 베이즈는 하이퍼-파라미터를 갖지 않는다.
13.3.6 AdaBoost
AdaBoost는 부스팅 앙상블 기법이다. 부스팅은 분류기 classifier를 반복적으로 추가하여 수행되지만, 다음 분류기가 학습될 때 비용 함수에서 이전 분류기에 의해 잘못 분류된 데이터 포인트에 더 많은 가중치를 준다. Python에 있는 sklearn AdaboostClassifier 을 사용한다.
| 파라미터 | 설명 | Typical values |
|---|---|---|
| nEstimators | The maximum number of estimators at which boosting is terminated | 4 |
| learningRate | Learning rate shrinks the contribution of each classifier by learning_rate. There is a trade-off between learningRate and nEstimators | 1 |
13.3.7 의사 결정 나무
의사 결정 나무 decision tree는 탐욕 접근 greedy approach방식을 사용하여 선택한 개별 테스트를 사용하여 가변 공간을 분할하는 분류기이다. 이것은 클래스를 분리하는 데 가장 많은 정보를 얻는 파티션을 찾는 것을 목표로 한다. 의사 결정 나무는 많은 수의 파티션 tree depth을 사용하게 되면 쉽게 과적합 되기 때문에 종종 일부 정규화 (예를 들어, 모델의 복잡성을 제한하는 하이퍼-파라미터의 정리 또는 지정) 가 필요하다. 우리는 Python에 있는 sklearn DecisionTreeClassifier 구현을 사용한다.
| Parameter | Description | Typical values |
|---|---|---|
| classWeight | “Balance” or “None” | None |
| maxDepth | The maximum depth of the tree | 10 |
| minImpuritySplit | Threshold for early stopping in tree growth. A node will split if its impurity is above the threshold, otherwise it is a leaf | 10^-7 |
| minSamplesLeaf | The minimum number of samples per leaf | 10 |
| minSamplesSplit | The minimum samples per split | 2 |
13.3.8 Multilayer Perceptron
Multilayer perceptrons은 비선형 함수를 사용하여 입력에 가중치를 부여하는 여러 계층의 노드를 포함하는 신경망이다. 첫 번째 레이어는 입력 레이어 input layer이고 마지막 레이어는 출력 레이어 output layer이며 그리고 그 사이에는 히든 레이어 hidden layers가 있다. 신경망은 일반적으로 역 전파 back-propagation를 사용하여 학습된다. 즉, 학습 입력 training input이 네트워크를 통해 앞으로 전달되어 출력을 생성하고, 출력과 결과 상태 사이의 오류가 계산되며, 이 오류는 네트워크를 통해 뒤로 전달되어 선형 함수 가중치를 업데이트한다.
| Parameter | Description | Typical values |
|---|---|---|
| alpha | The l2 regularization | 0.00001 |
| size | The number of hidden nodes | 4 |
13.3.9 딥 러닝
Deep net, convolutional neural networks 또는 recurrent neural networks와 같은 딥 러닝 deep learning은 Multilayer perceptrons와 유사하지만, 예측에 유용한 잠재 표현을 학습하는 것을 목표로 하는 숨겨진 레이어를 여러 개 가지고 있다. PatientLevelPrediction 패키지의 별도의 vignette 에 이러한 모델과 하이퍼-파라미터에 대해 자세히 기술되어 있다.
13.3.10 다른 알고리즘
다른 알고리즘도 환자-수준 예측 프레임워크에 추가될 수 있다. 이것은 이 장의 범위를 벗어난다. 자세한 내용은 PatientLevelPrediction 패키지의 “Adding Custom Patient-Level Prediction Algorithms” vignette에 있다.
13.4 예측 모델 평가
13.4.1 평가 유형
예측값과 관측값의 일치 여부를 평가함으로써 예측 모델을 평가할 수 있는데, 그렇게 하려면 결과 상태가 있는 데이터가 필요하다.
평가는 다음 두 가지로 나뉜다:
- 내적 타당도 internal validation: 같은 데이터베이스에서 추출된 다른 데이터 모음을 사용하여 모델을 개발하고 평가.
- 외적 타당도 external validation: 한 데이터베이스에서 모델을 개발하고 다른 데이터베이스에서 평가.
내적 타당도를 수행하는 데는 두 가지 방법이 있다:
- 홀드 아웃 모음 holdout set 접근법은 레이블이 지정된 데이터를 두 개의 독립된 훈련 모음 training set과 테스트 모음 test set으로 나누는 것이다. 훈련 모음은 모델 학습에 사용되고, 테스트 모음은 모델 평가에 사용된다. 환자군을 무작위로 훈련 모음과 테스트 모음으로 나누거나 다음과 같이 선택할 수 있다:
- 날짜를 기반으로 데이터를 분할한다 (시점 타당도 temporal validation). 예를 들어, 특정 날짜 이전의 데이터로 학습시키고, 그 특정 날짜 이후 데이터로 평가하는 것이다. 이것은 모델이 서로 다른 기간에서도 일반화가 가능한지 여부를 알 수 있게 해준다.
- 지리적 위치를 기반으로 데이터를 분할한다. 이는 훈련 모음과 테스트 모음에 outcome 발생이 동일한 비율로 나타나도록 환자들을 나눈다는 뜻이다.(공간 타당도 spatial validation).
- 교차 검증은 데이터가 제한적일 때 유용하다. 데이터를 \(n\)개의 동일 크기의 모음으로 분할한다. 여기서 \(n\)은 미리 정해져야 한다 (예를 들어, \(n=10\)). 이러한 각 모음에 대해 한 모음의 데이터를 제외한 모든 데이터를 이용해 모델을 학습하며, 감추어 둔 한 모음 (홀드 아웃 모음)은 예측 (평가)에 사용된다. 이를 \(n\)번 반복하여 모든 데이터가 한 번씩 모델을 구축하는 알고리즘을 평가하는데 사용된다. PLP 프레임 워크에서는 최적의 하이퍼-파라미터를 선택하는 데 교차 검증을 이용한다.
외적 타당도는 다른 데이터베이스, 즉 개발에 사용된 데이터베이스가 아닌 다른 데이터베이스로부터 데이터를 얻어 모델의 성능을 평가하는 것을 목표로 한다. 훈련에 사용한 그 데이터베이스뿐만 아니라 다른 데이터베이스에도 우리가 개발한 모델을 적용하기를 원하기 때문에 모델의 타 기관 적용 방법론은 중요하다. 다른 데이터베이스란 다른 환자 집단, 다른 의료 시스템 및 다른 데이터 획득 프로세스를 대변한다. 대규모 데이터베이스 모음에 대한 예측 모델의 외적 타당도 검증은 임상 실무에서 예측 모델을 수용하고 구현하기 위해 결정적이라고 생각한다.
13.4.2 성능 지표
임계값 지표
예측 모델은 위험에 노출된 시간 동안 관심 결과가 발생할 위험이 있는 각 환자에 대해 0과 1 사이의 값을 할당한다. 0값은 0% 위험을 의미하고 0.5값은 50% 위험을 의미하고 1값은 100% 위험을 의미한다. 위험에 처한 시간 동안 환자가 관심 결과를 갖는지 여부를 결정하는 임계치(기준치)를 정하면 정확도, 민감도, 특이도, 양성 예측도와 같은 일반적인 측정 지표를 계산해 낼 수 있다. 예를 들어, 표 13.11에서 임계값을 0.5로 설정하면 환자 1, 3, 7 및 10은 예상 위험이 임계값 0.5보다 크거나 같으므로 관심 결과가 발생할 것으로 예측된다. 다른 모든 환자는 0.5 미만의 예측 위험을 가지고 있으므로 관심 결과는 없을 것으로 예측된다.
| Patient ID | Predicted risk | Predicted class at 0.5 threshold | Has outcome during time-at-risk | Type |
|---|---|---|---|---|
| 1 | 0.8 | 1 | 1 | TP |
| 2 | 0.1 | 0 | 0 | TN |
| 3 | 0.7 | 1 | 0 | FP |
| 4 | 0 | 0 | 0 | TN |
| 5 | 0.05 | 0 | 0 | TN |
| 6 | 0.1 | 0 | 0 | TN |
| 7 | 0.9 | 1 | 1 | TP |
| 8 | 0.2 | 0 | 1 | FN |
| 9 | 0.3 | 0 | 0 | TN |
| 10 | 0.5 | 1 | 0 | FP |
환자에게 관심 결과 outcome가 예측되고 (위험 노출 시간 동안) 관심 결과가 발생했다면 이를 진양성(TP)이라고 한다. 환자에게 관심 결과가 있을 것으로 예상되지만 그 결과가 없는 경우라면 이를 거짓 양성(FP)이라고 한다. 환자에게 관심 결과가 없을 것으로 예상되고 실제 결과가 없는 경우 이를 진음성(TN)이라고 한다. 마지막으로, 환자에게 관심 결과가 없을 것으로 예상되지만 결과가 있는 경우 이를 거짓 음성(FN)이라고 한다.
다음과 같이 임계값-기반 지표를 계산할 수 있다:
- 정확도 accuracy: \((TP+TN)/(TP+TN+FP+FN)\)
- 민감도 sensitivity: \(TP/(TP+FN)\)
- 특이도 specificity: \(TN/(TN+FP)\)
- 양성예측도 positive predictive value: \(TP/(TP+FP)\)
임계값을 낮추면 이러한 값이 감소하거나 증가 할 수 있다. 분류기의 임계값을 낮추면 반영되는 결과 수가 증가하여 분모가 증가할 수 있다. 이전에 임계값을 너무 높게 설정한 경우, 새로운 결과는 모두 진양성이 될 수 있으며, 이는 양성 예측도를 증가시킨다. 이전 임계값이 적절하거나 또는 너무 낮다면 임계값을 더 낮추게 되면 거짓 양성이 발생하여 양성 예측도가 감소한다. 민감도의 분모는 분류기의 임계값 (\(TP+FN\)은 상수)에 의존하지 않는다. 이는 분류기의 임계값을 낮추면 진양성 결과 수를 증가 시켜 민감도를 높일 수 있음을 의미한다. 또한, 임계값을 낮추면 민감도가 바뀌지 않고 양성 예측도가 변할 수 있다.
판별력
판별력 discrimination은 위험에 노출된 시간 동안 관심 결과를 경험할 환자에게 더 높은 위험을 할당하는 능력이다. Receiver Operating Characteristics(ROC) 곡선은 가능한 모든 임계값에 대하여 x축은 1-특이도를 그리고 y축에는 민감도를 그린 것이다. ROC 곡선은 이 장의 뒷부분에 있는 그림 13.17에 나와 있다. area under the receiver operating characteristic curve(AUC)가 0.5이면 위험에 무작위로 할당되는 것을 의미하고 값이 1이면 완벽하게 판별한다는 의미이다. 대부분 출판된 예측 모델의 AUC는 0.6-0.8 사이의 값을 갖고 있다.
AUC는 위험에 노출된 시간 동안 관심 결과를 경험한 환자와 그렇지 않은 환자 간에 예측된 위험 분포가 얼마나 다른지 결정하는 방법을 제공한다. AUC가 높으면 위험 분포가 대부분 분리되지만, 겹치는 부분이 많을 때는 그림 13.3과 같이 AUC가 0.5에 가까워진다.
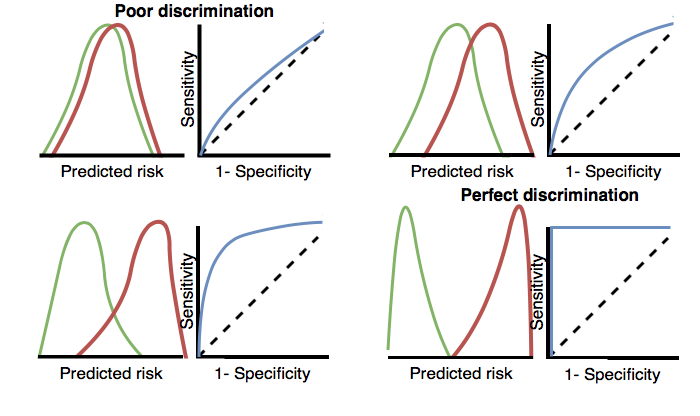
Figure 13.3: ROC 플롯이 어떻게 판별력과 연관되어 있는지를 설명한다. 두 등급의 예측 위험 분포가 유사한 경우, ROC는 대각선에 가까우며 AUC는 0.5에 가깝다 (좌측 상단).
결과가 드물게 발생할 경우, AUC가 높은 값을 갖는 모델이라도 주어진 임계값을 초과하는 모든 양성에 대해 음성이 많을 수 있으므로 (즉, 양성 예측도가 낮을 수 있기 때문에) 실용적이지 않을 수 있다. 결과의 심각성과 일부 중재에 대한 비용 (건강 위험/금전적)에 따라, 거짓 양성비가 커질 수 있다. 즉, outcome 이 매우 드물다면, AUROC가 높더라도 실제 예측력은 낮을 수 있다는 뜻이다. 이런 경우 area under the precision-recall curve(AUPRC)라고 알려진 다른 측정법이 권장된다. AUPRC는 x축은 민감도 (재현율 recall이라고도 함)와 y축은 양성예측도 (정밀도 precision라고도 함)을 나타내는 곡선하 면적이다.
적합도
적합도 calibration는 모델이 정확한 위험을 할당하는 능력이다. 예를 들어, 모델이 100명의 환자에게 10%의 위험이 발생 가능성이 있다고 할 경우 10명의 환자는 위험 노출 시간 중에 결과를 경험해야 한다. 모델이 100명의 환자에게 80%의 위험 발생 가능성이 있다고 하면 80명의 환자는 위험 노출 시간 동안 결과를 경험해야 한다. 적합도는 일반적으로 예측된 위험에 따라 환자를 십분위(열 등분)로 분할하고 각 그룹에서 평균 예측 위험과 실제 결과를 경험한 환자의 비율을 이용하여 계산한다. 그런 다음 10개의 점 (y축에 예측 위험과 x축에 관찰된 위험)을 그린 후 그 점들이 x = y인 선에 가까이 있는지, 즉 모델이 잘 보정되었는지를 확인한다. 적합도 그래프는 이 장의 뒷부분에 있는 그림 13.18에 나와 있다. 또한 점을 사용하여 절편 (0에 가까워 야 함)과 기울기 (1에 가까워야 함)를 계산하여 선형 모델을 만든다. 기울기가 1보다 크면 모델이 실제 위험보다 높은 위험을 할당하고 있고 기울기가 1보다 작으면 모델이 실제 위험보다 낮은 위험을 나타내는 것이다. 예측된 위험과 관찰된 위험 사이의 비선형 관계를 더욱 잘 포착하기 위해서, PLP R 패키지에 Smooth Calibration Curves를 구현했다.
13.5 환자-수준 예측 연구 설계
이 장에서는 예측 연구를 설계하는 방법을 보여준다. 첫 번째 단계는 예측 문제를 명확하게 정의하는 것이다. 흥미롭게도, 많은 출판된 논문에서 예측 문제가 잘 정의되어 있지 않다. 예를 들어 기준 날짜 (대상 코호트의 시작)가 어떻게 정의되어 있는지 명확하지 않다. 잘못 정의된 예측 문제는 임상 실무에서의 구현은 물론 다른 사람에 의한 외적 타당도 검정이 불가능하다. 환자-수준 예측 프레임워크에서 표 13.1에 정의된 주요 선택 사항을 명시적으로 정의하게 함으로써 예측 문제에 대한 적절한 명세서를 만들도록 한다. 여기서는 “치료 안전성”을 보는 유형의 예측 문제를 예로 들어 이 프로세스를 살펴보자.
13.5.1 문제 정의
혈관 부종 Angioedema은 ACE 억제제의 잘 알려진 부작용이며, ACE 억제제에 대한 레이블에 보고된 혈관 부종의 발생률은 0.1 % - 0.7% 범위이다. (Byrd, Adam, and Brown 2006) 혈관 부종은 드물지만, 생명을 위협하여 호흡 정지 및 사망으로 이어질 수 있기 때문에 이러한 부작용에 대한 환자 모니터링은 중요하다. (Norman et al. 2013) 또한, 혈관 부종이 조기에 인식되지 않으면, 식별할 원인의 범위가 넓어지고 값비싼 정밀검사를 해야 할 수 있다. (Norman et al. 2013; Thompson and Frable 1993) 아프리카계 미국인 인종이 ACE 억제제으로 인한 혈관 부종 발생의 위험 인자라는 것을 제외하면 알려진 소인은 없다. (Byrd, Adam, and Brown 2006) 대부분의 혈관부종은 ACE 억제제 시작 후 첫 주 또는 한 달 안에, 그리고 종종 초기 복용 후 몇 시간 내에 발생한다 (Cicardi et al. 2004). 그러나 치료가 시작된 후 몇 년 후에 발생할 수도 있다. (O’Mara and O’Mara 1996) 어떤 환자가 고위험군인지 구체적으로 식별하는 진단 테스트는 없다. 만약 고위험군을 식별할 수만 있다면, 의사는 ACE 억제제 처방을 중단하고 다른 고혈압 약물을 처방할 것이다.
관찰 의료 데이터에 환자-수준 예측 프레임워크를 적용하여 다음의 환자-수준 예측 질문에 응용해 보자:
처음에 ACE 억제제로 치료를 시작한 환자 중, 어떤 환자에서 1년 안에 혈액 부종이 발생할 것인가?
13.5.2 연구 모집단 정의
예측 모델을 개발하기 위한 최종 연구 모집단은 종종 대상 코호트의 하위 집단인데, 왜냐하면 관심 결과에 의존하는 기준을 적용하거나 또는 대상 코호트의 부분 모집단 sub-population의 민감도 분석을 수행하고자 하기 때문이다. 이것을 위하여 우리는 다음의 질문을 설명해야만 한다:
대상 코호트의 기준 날짜 이전의 최소 필요 관찰 기간은 얼마인가? 이 선택은 학습 데이터에서 사용 가능한 환자 시간에 따라 달라질 수 있지만, 장차 모델을 적용하려는 데이터 소스에서 사용 가능할 것으로 예상되는 시간에 따라 달라질 수도 있다. 최소 관측 시간이 길어질수록 특징 추출에 사용할 수 있는 기저력 시간 baseline history time이 길어지지만, 대신 분석에 사용할 수 있는 환자 수는 줄어든다. 또한, 단기 또는 장기의 과거력 관찰 시간을 선택해야 하는 임상적 이유가 있을 수 있다. 예제에서는 기준 날짜로부터 365일 이전까지의 기록을 기저 관찰 기간 (휴약기 washout period)으로 사용한다.
환자가 대상 코호트에 여러 번 포함될 수 있는가? 대상 코호트 정의에서, 사람은 서로 다른 시간 간격 동안, 예를 들어 서로 다른 에피소드의 질병이 있거나 의료 제품에 대한 또 다른 노출 기간이 있는 경우 그 코호트에 여러 번 포함될 수 있다. 코호트 정의는 환자가 한 번만 들어갈 수 있도록 제한을 적용할 필요는 없지만, 특정 환자-수준 예측 문제와 관련하여 코호트를 첫 번째 관찰로 제한 할 수 있다. 이 예제에서는 기준이 ACE 억제제의 첫 번째 사용을 기반으로 했기 때문에 대상 코호트에 한 번만 들어갈 수 있다.
이전에 관심 결과를 이미 경험 한 사람이 코호트에 들어가도록 허용하는가? 대상 코호트에 포함되기 전에 관심 결과를 이미 경험한 사람이 대상 코호트에 다시 들어가도록 허용하는가? 특정 환자-수준 예측 문제에 따라, 결과가 처음 발생하게 되는 것을 예측하고자 할 수 있으며, 이 경우 이전에 결과를 경험한 환자는 그 결과가 처음 발생한 것이 아니므로 대상 코호트에서 제외돼야 한다. 다른 상황에서, 유행하는 에피소드를 예측하고자 하는 경우가 있을 수 있는데, 이로 인해 사전에 관심 결과를 가진 환자가 분석에 포함될 수 있고 사전 결과 자체가 미래 결과를 예측하기 위한 변수가 될 수 있다. 예제에서는, 이전에 혈관 부종을 앓는 사람을 포함하지 않도록 선택할 것이다.
대상 코호트 시작 날짜를 기준으로 결과 발생을 예측할 기간을 어떻게 정의하는가? 이 질문에 답하기 위해 두 가지 결정을 내려야 한다. 첫째, 결과가 발생할 수 있는 위험 기간이 대상 코호트가 시작된 그 날짜 (기준 날짜 index date)에 시작되는가 혹은 그 후에 시작되는가? 결과 발생 예측 기간을 나중으로 하자는 주장은, 결과 발생이 실제로는 대상 코호트가 시작되기 전에 이미 발생했지만, 기록이 늦게 되어 마치 기준 날짜와 비슷한 날짜에 나타난 것처럼 보일 수 있는 가능성을 배제하기 원하거나, 혹은 결과를 막기 위한 개입이 일어날 수 있는 여유시간을 남겨 두고 싶을 수 있기 때문이다. 둘째, 대상 코호트 시작 또는 종료 날짜를 기준으로 얼마동안의 기간 time window 동안 결과 발생을 관찰할 지 여부이다. 예제에서는 대상 코호트의 시작일 하루 뒤부터 365일까지를 위험 발생 가능 기간으로 정하여 예측할 것이다.
결과 발생 최소 위험 시간 minimum amount of time-at-risk이 필요한가? 관심 결과가 생기지는 않았지만, 정의한 위험 발생 기간이 끝나기 전에 데이터베이스에 남지 않는 (자료가 없어서 관찰이 종료된) 환자를 포함할 것인지 결정해야 한다. 그러한 환자는 우리가 그들을 더 이상 관찰하지 않는 동안에 결과가 생길 수도 있다. 예제에서는 위험 노출 최소 시간이 필요한 것으로 했는데, 그 이유는 더 이상 관찰하지 않는 동안에 결과가 생길 수도 있기 때문이다. 또한 이 제약 조건이 결과를 경험한 사람에게도 적용되게 할지 결정해야 한다. 그렇지 않으면 전체 위험 발생 시간과 관계없이 결과가 발생한 모든 사람을 연구에 포함하게 될 것이다. 예를 들어, 결과가 사망인 경우 전체 위험 발생 기간이 완료되기 전에 사망한 사람이 중도 절단될 수 있다.
13.5.3 모델 개발 설정
예측 모델을 개발하기 위해 학습하고자 하는 알고리즘을 결정해야 한다. 특정 예측 문제에 대해서 최상의 알고리즘을 선택하는 방법은 경험적 질문이라고 본다. 즉, 데이터를 이용해서 그 자체가 여러 개의 접근법을 시도하게 하고 그중에서 최상의 것을 선택하도록 하는 것을 선호한다. PLP 프레임워크에서는 13.3절에 설명된 대로 많은 알고리즘을 구현했을 뿐더러, 연구자가 다른 알고리즘을 추가 할 수도 있다. 이 예제에서는 작업을 단순하게 하기 위해 Gradient Boosting Machines 알고리즘을 선택한다.
또한, 모델을 학습하기 위하여 사용할 공변량을 결정해야 한다. 이 예제에서는 성별, 연령, 모든 질병, 의약품과 의약품 그룹 및 방문 횟수를 추가한다. 우리는 기준 날짜 이전부터 1년 전까지의 이러한 여러 임상적 사건을 사용할 것이다.
13.5.4 모델 평가
마지막으로 모델 평가 방법을 정의해야 한다. 편의상 여기서는 내적 타당도를 선택했다. 학습과 테스트를 위한 데이터 모음에서 이것을 나누는 방법과 환자를 이 두 데이터 모음으로 할당하는 방법을 결정해야 한다. 여기에서는 일반적으로 하는 75%-25% 분할을 사용한다. 매우 큰 데이터 모음의 경우 학습데이터 모음에 더 많은 데이터를 포함해서 사용할 수 있다.
13.5.5 연구 요약
우리 연구를 위하여 완벽하게 정의한 내용을 표 13.12에 나타내었다.
| 선택 | 설명 |
|---|---|
| 대상 코호트 Target cohort | 생애 처음으로 ACE 억제제를 사용하기 시작한 환자군. 기저 관찰 기간이 1년 미만이거나 이전에 혈관부종이 발생한 환자는 제외한다. |
| 결과 코호트 Outcome cohort | 혈관부종 발생군 |
| 위험노출기간 Time-at-risk | 코호트 시작일로부터 1일 이후부터 365일까지. 즉 위험노출기간이 최소한 364일 필요하다. |
| 모델 Model | Gradient boosting machine에 다음의 하이퍼파라미터를 사용한다: ntree=5000, max depth=4 or 7 or 10, learning rate=0.001 or 0.01 or 0.1 or 0.9; 예측 변수로는 성별, 나이, 상태 condition, 약물력 drug, 약물 그룹 drug group 과 방문 횟수 visit count를 포함한다. 데이터 분할: 훈련 모음 75%, 테스트 모음 25%를 사람별로 무작위 배정한다. |
13.6 ATLAS에서의 연구 구현하기
예측 연구를 설계하기 위한 인터페이스는 ATLAS 메뉴  버튼을 누르면 열 수 있다. 새로운 예측 연구를 만든다. 새로운 예측 연구를 생성하라. 만든 연구에 알기 쉬운 이름을 부여하라. 연구 디자인은
버튼을 누르면 열 수 있다. 새로운 예측 연구를 만든다. 새로운 예측 연구를 생성하라. 만든 연구에 알기 쉬운 이름을 부여하라. 연구 디자인은  버튼을 클릭하면 언제든지 저장이 가능하다.
버튼을 클릭하면 언제든지 저장이 가능하다.
예측 디자인 기능에는 4개의 세션이 있다: 예측 문제 설정 Prediction Problem Setting, 분석 설정 Analysis Setting, 실행 설정 Execution Setting, 학습 설정 Training Setting. 다음은 각 세션에 대한 설명이다.
13.6.1 예측 문제 설정 Prediction Problem Setting
여기서는 대상 코호트와 결과 코호트를 선택할 수 있다. 대상 코호트와 결과 코호트의 모든 조합에 대해 예측 모델이 개발될 것이다. 예를 들어, 만약 두 개의 대상 모집단과 두 개의 대상 결과를 지정한다면 네 개의 예측 문제가 지정된다.
대상 코호트를 선택하기 위해서는 사전에 ATLAS에서 정의하여야 한다. 예시 코호트가 10장에 있다. 부록은 이 예시에 사용된 대상 (부록 B.1)과 결과 (부록 B.4) 코호트의 전체 정의를 제공한다. 대상 모집단을 코호트에 추가하기 위해서는 “대상 코호트 추가 [Add Target Cohort]” 버튼을 클릭해라. 결과 코호트를 추가하는 것은 “결과 코호트 추가 [Add Outcome Cohort]” 버튼을 클릭하면 마찬가지로 작동된다. 완료되면, Dialog는 그림 13.4처럼 보일 것이다.
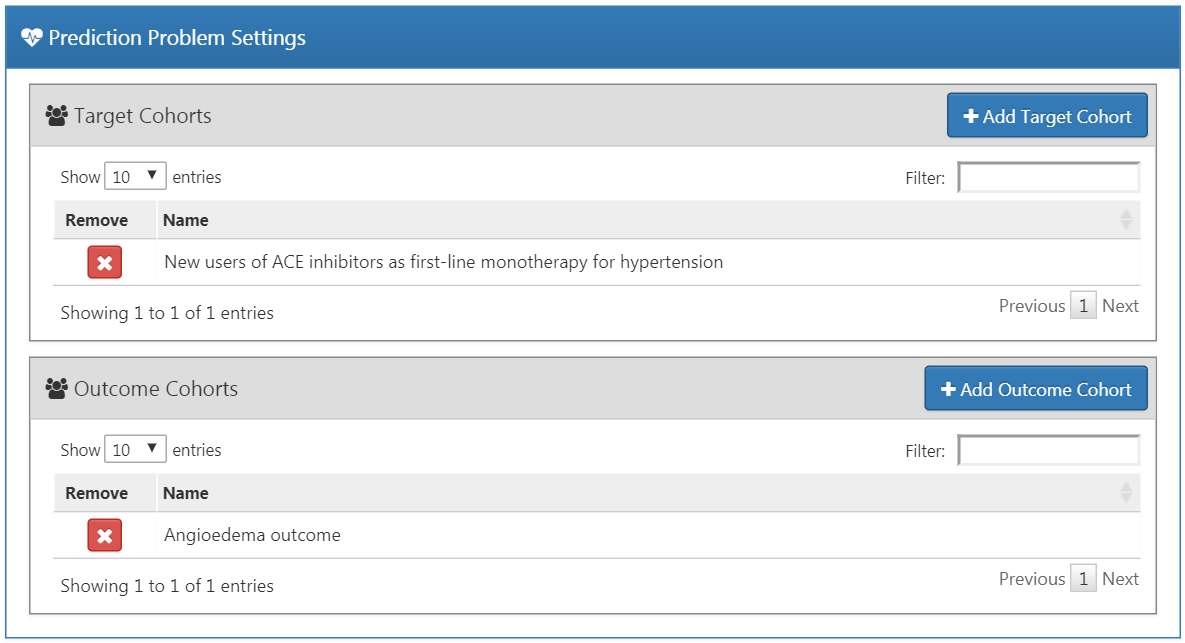
Figure 13.4: 예측 문제 설정.
13.6.2 분석 설정 Analysis Setting
분석 설정에서 지도 학습 알고리즘, 공변량 및 모집단 설정을 할 수 있다.
모델 설정 Model Setting
모델 개발을 위해 하나 혹은 더 많은 지도 학습 알고리즘을 선택할 수 있다. 지도 학습 알고리즘을 추가하기 위해서는 “모델 설정 추가 [Add Model Settings]”버튼을 눌러라. 현재 ATLAS에서 지원되는 모든 모델이 포함된 드롭다운이 나타난다. 드롭다운 메뉴에 있는 이름을 클릭하여 원하는 연구가 포함된 지도 학습 모델을 선택 할 수 있다. 그러고 나면 지정된 모델의 창을 볼 수 있고, 하이퍼 파라미터값을 선택 할 수 있다. 여러 값이 제공되는 경우 가능한 모든 값의 조합으로 교차 검증을 사용하여 최적의 조합을 선택하기 위해 그리드서치 Grid Search를 시행한다.
여기의 예에서는 Gradient Boosting Machine(GBM)을 선택하고 그림 13.5에 지정된 것처럼 하이퍼 파라미터를 설정한다.
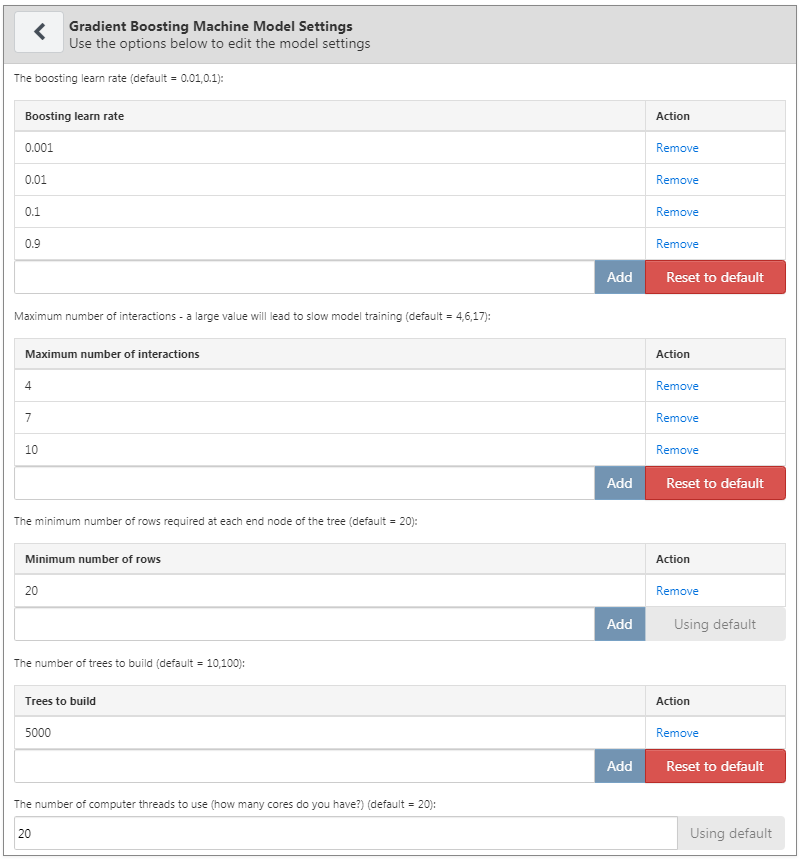
Figure 13.5: Gradient Boosting Machine 설정.
공변량 설정
우리는 이미 CDM 포맷의 관찰 데이터에서 추출할 수 있는 표준 공변량 covariate을 정의했다. 공변량 설정 창에서 포함할 표준 공변량을 선택할 수 있다. 다양한 유형의 공변량을 정의 할 수 있으며 각 모형은 각각 지정된 공변량 설정과 함께 별도로 생성될 것이다.
연구에서 공변량 설정을 추가하려면, “공변량 설정 추가 [Add Covariate Settings]”을 클릭하라. 그러면 공변량 설정 창이 열린다.
공변량 설정 창의 첫 번째 부분은 제외/포함 옵션이다. 공변량은 일반적으로 모든 개념에 맞게 구성된다. 그러나, 어떤 개념이 대상 코호트 정의와 연결된 경우와 같이 특정한 개념을 추가/제외하기 원할 수도 있다. 특정 개념만 포함하려면 ATLAS에서 개념 모음을 설정한 다음에 “환자-수준 예측 모형의 기저 공변량에 어떤 개념을 포함하시겠습니까? (모든 것을 포함하려면 비워 두십시오) What concepts do you want to include in baseline covariates in the patient-level prediction model?” 아래  를 클릭하여 개념 모음을 선택한다. “포함한 개념 목록에 그 하위 개념을 추가합니까? Should descendant concepts be added to the list of included concepts?” 라는 질문에 “예”라고 답함으로써 모든 하위 개념을 자동으로 개념 모음에 추가할 수 있다. 같은 절차는 “환자 수준 예측 모형의 기저 공변량에 어떤 개념을 제외하시겠습니까? (모든 것을 포함하려면 공란으로 남겨두자) What concepts do you want to exclude in baseline covariates in the patient-level prediction model” 질문에서 동일한 과정을 반복할 수 있고 선택된 개념에 해당하는 공변량을 제거할 수 있다. 마지막 옵션인 “쉼표로 구분되는 공변량 ID의 리스트의 범위를 정한다. A comma delimited list of covariate IDs that should be restricted to”를 사용하면 공변량의 ID 모음 (개념 ID가 아닌) 을 쉼표로 구분하여 추가할 수 있다. 이 옵션은 고급 사용자에게만 필요하다. 완료되면 포함 및 제외 옵션이 그림 13.6과 같아야 한다.
를 클릭하여 개념 모음을 선택한다. “포함한 개념 목록에 그 하위 개념을 추가합니까? Should descendant concepts be added to the list of included concepts?” 라는 질문에 “예”라고 답함으로써 모든 하위 개념을 자동으로 개념 모음에 추가할 수 있다. 같은 절차는 “환자 수준 예측 모형의 기저 공변량에 어떤 개념을 제외하시겠습니까? (모든 것을 포함하려면 공란으로 남겨두자) What concepts do you want to exclude in baseline covariates in the patient-level prediction model” 질문에서 동일한 과정을 반복할 수 있고 선택된 개념에 해당하는 공변량을 제거할 수 있다. 마지막 옵션인 “쉼표로 구분되는 공변량 ID의 리스트의 범위를 정한다. A comma delimited list of covariate IDs that should be restricted to”를 사용하면 공변량의 ID 모음 (개념 ID가 아닌) 을 쉼표로 구분하여 추가할 수 있다. 이 옵션은 고급 사용자에게만 필요하다. 완료되면 포함 및 제외 옵션이 그림 13.6과 같아야 한다.
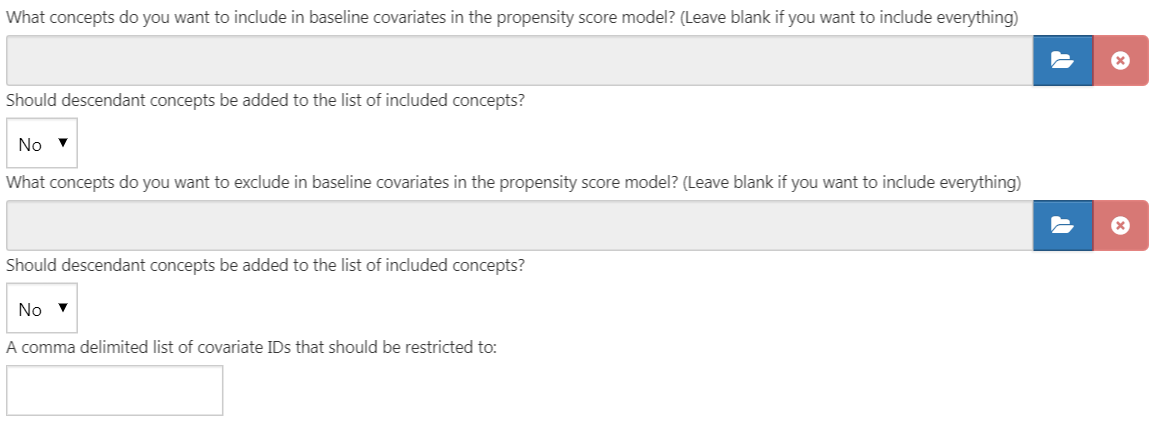
Figure 13.6: 공변량 포함 및 제외 설정.
다음에서는 시간과 독립적인 변수를 선택할 수 있다.
- 성 [Sex]: 남자 또는 여자 성별을 나타내는 이항 변수
- 나이 [Age]: 연령에 해당하는 연속 변수
- 나이 그룹 [Age group]: 5년 단위의 이항 변수 (0-4, 5-9, 10-14, …, 95+)
- 인종 [Race]: 각 인종의 이항 변수, 1은 환자의 인종 기록이 있음을 의미하며, 그렇지 않으면 0이다.
- 민족 [Ethnicity]: 민족에 대한 이항 변수, 1은 환자의 민족 기록이 있음을 의미하며, 그렇지 않으면 0이다.
- 기준 연도 [Index year]: 각 코호트 연도 시작 날짜에 대한 이항 변수, 예를 들어 2012라는 기준 연도 값에 1이라는 값이 있다면 해당 환자의 코호트 시작 연도가 2012년이라는 것을 뜻하고, 코호트 시작 연도가 2013이라면 해당 값은 0일 것이다. 환자의 코호트 시작 연도가 2013이라면 2013이라는 기준 연도 값에 1이 표기 될 것이다.미래에 발생하는 일에 대해서도 이 모형을 적용하길 원할 수 있기 때문에, 색인 연도를 포함하는 것은 보통 합리적이지 않다.
- 기준인 월 [Index month]: 각 코호트 달 시작 날짜에 대한 이항 변수, 값에 1이 있다면 해당 월이 환자 코호트 시작 월임을 의미하고, 그렇지 않으면 0이다.
- 사전 관찰 시간 [Prior observation time]: [예측에 사용을 권장하지 않음] 코호트 시작일 이전에 환자가 데이터베이스에 있었던 기간(일)에 해당하는 연속 변수
- 사후 관찰 시간 [Post observation time]: [예측에 사용을 권장하지 않음] 코호트 시작일 이후에 환자가 데이터베이스에 있었던 기간(일)에 해당하는 연속 변수
- 코호트 내 시간 [Time in cohort]: 환자가 코호트에 있었던 기간에 해당하는 연속 변수 (코호트 종료일에서 코호트 시작일을 뺀)
- 기준 연도 및 월 [Index year and month]: [예측에 사용을 권장하지 않음] 각 코호트 시작 연월 날짜에 대한 이항변수, 1은 환자 코호트 시작 연월 날짜이고, 그렇지 않으면 0이다.
완료된다면, 그림 13.7과 같아야 한다.

Figure 13.7: 공변량 선택.
표준 공변량은 공변량에 대해 세 개의 유동적인 시간 간격을 가능하게 한다:
- 종료일: 코호트 시작 날짜를 기준으로 종료 시기와의 간격 [기본값 0]
- 장기간 [기본 값: 코호트 시작일 이전 365일부터 코호트 시작일 바로 하루 전까지]
- 중기간 [기본 값: 코호트 시작일 이전 180일부터 코호트 시작일 바로 하루 전까지]
- 단기간 [기본 값: 코호트 시작일 이전 30일부터 코호트 시작일 바로 하루 전까지]
완료가 된다면, 그림 13.8과 같을 것이다.
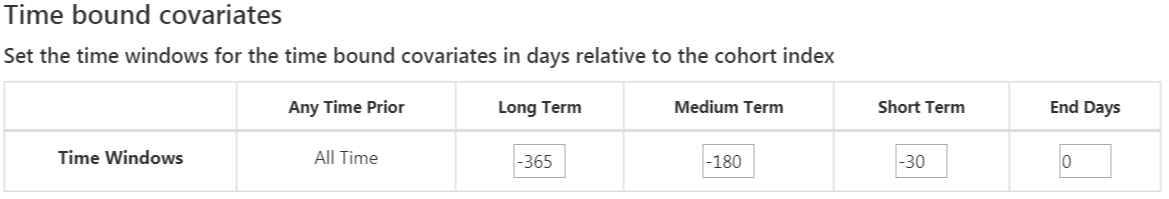
Figure 13.8: Time bound 공변량.
다음 옵션은 ERA 테이블을 이용해 추출한 공변량이다:
- Condition: 각 상태(질병) 개념의 연속된 기간(era)과 선택한 시간 간격을 이용해서 공변량 구축. 연구자가 정한 사전(기저) 관찰 기간 내에 era의 시작과 끝이 포함되거나, 혹은 era의 시작과 끝이 지정한 사전 관찰 기간을 내포하는 경우 공변량 값은 1이고, 그렇지 않으면 0이다. 연구자가 정한 사전 관찰기간은 코호트 시작일보다 이전이어야 한다. (역자 주: 질병 개념의 연속된 기간(era)은 CDM 데이터베이스를 만들 때 모두 계산해서 era 테이블에 저장해 둔다.)
- Condition group: 각 질병 개념 및 그 하위(자식) 개념을 포함하는 연속된 기간 (era)과 선택한 시간 간격을 이용해서 공변량 구축. 환자가 질병 era 테이블에서 코호트 시작일 이전에 지정된 시간 간격 동안, era가 있는 개념 ID 또는 하위 개념 ID(any descendant concept ID) 를 갖는 경우, 공변량 값은 1이고, 그렇지 않으면 0이다. (역자 주: PLP package는 대부분의 예측인자에 대해 이항 변수화 one-hot encoding을 취하기 때문에 condition을 추가할 경우 환자들이 가지고 있는 진단 코드 하나하나가 모두 예측인자로 추가된다. 우리는 ‘당뇨망막증을 동반한 당뇨병’ 보다는 상위개념인 ’당뇨병’만을 예측인자로 사용하고 싶을 수 있다. grouping이라는 것은 상위 개념을 사용하여 granularity를 감소시킨다는 것을 의미한다.)
- Drug: 각 약물 개념의 연속된 기간(era)과 선택한 시간 간격을 이용해서 공변량 구축. 환자가 약물 era 테이블에서 코호트 시작일 이전의 지정된 시간 간격 동안 era가 있는 개념 ID를 가지고 있는 경우 공변량 값은 1이며, 그렇지 않으면 0이다. (역자 주: 위 condition 변수와 같은 구성인데 condition era 대신 drug era를 쓴 것)
- Drug group: 각 약물 개념 및 그 하위(자식) 개념을 포함하는 연속된 기간(era)과 선택한 시간 간격을 이용해서 공변량을 구축. 환자가 약물 era 테이블에서 코호트 시작일 이전의 지정된 시간 간격 기간 동안 개념 ID 또는 하위 개념 ID(any descendant concept ID) 를 가진 경우 공변량 값은 1이며, 그렇지 않으면 0이다.
겹치는 시간 간격 설정은 약물 또는 질병 발생대(era)가 코호트 시작 날짜 이전에 시작하고 코호트 시작 날짜 이후에 끝나야 하므로 코호트 시작 날짜와 겹친다. Era start 옵션은 선택한 시간 간격 동안 시작되는 질병 또는 약물 era를 찾는 것으로 제한된다. (역자 주: 위 drug에서 하위(자식) 개념도 포함한 것)
완료된다면, 그림 13.9과 같이 보일 것이다.
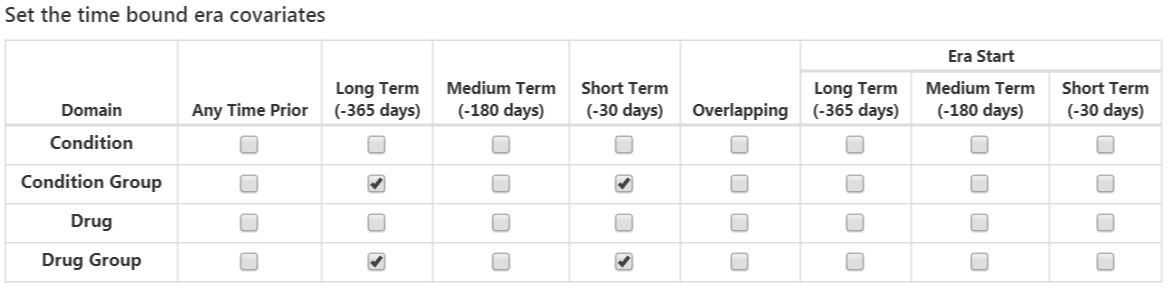
Figure 13.9: Time bound era 공변량.
다음은 도메인별 개념 concept ID에 대해 다양한 시간 간격을 옵션으로 가진 공변량을 선택한다.
- Condition: 각 질병 concept ID와 선택한 시간 간격으로 공변량을 구축. 코호트 시작일 이전을 기준으로 선택한 시간 간격 동안 질병 개념 ID가 나타나면 공변량 값은 1이며, 그렇지 않으면 0이다. (역자 주: 예를 들어 연구 대상군에 포함된 환자는 100명이고, 그들 환자 데이터에서 코호트 시작일 이전 사전 관찰 기간 동안 총 1,000종류의 고유한 진단명이 나타났다고 하면, 총 1,000개의 진단명 변수가 생성된다. 이하 아래 모든 도메인별 변수도 마찬가지. 결과적으로 입력변수가 아주 많은 초기 토폴로지가 구성되고, 이후 변수 선택과정에서 학습에 기여하지 않는 변수는 제거된다)
- Condition Primary Inpatient: 입원 시 주 진단명 별로 생성되는 이항 공변량.
- Drug: 각 약물 개념 ID 및 시간 간격으로 공변량을 계산하고 약물 노출 테이블의 코호트 시작일 이전에 지정된 시간 간격 동안 기록된 개념 ID를 가지고 있는 경우 공변량 값은 1, 그렇지 않으면 0이다.
- 시술 Procedure: 각 시술 개념 ID와 선택한 시간 간격으로 공변량을 구축. 환자의 수술 발생 테이블의 코호트 시작일 이전에 지정된 시간 간격 동안 기록된 개념 ID를 가지고 있는 경우 공변량 값은 1, 그렇지 않으면 0이다.
- 검사 Measurement: 각 검사 개념 ID와 선택한 시간 간격으로 공변량을 구축. 환자가 측정 테이블의 코호트 시작일 이전에 지정된 시간 간격 동안 기록된 개념 ID를 가지고 있는 경우 공변량 값은 1, 그렇지 않으면 0이다.
- 검사 값 Measurement Value: 각 검사 개념 ID, 값 및 선택한 시간 간격으로 공변량을 구축. 환자가 측정 테이블의 코호트 시작일 이전에 지정된 시간 간격 동안 기록된 개념 ID를 가지고 있는 경우 값은 측정값, 그렇지 않으면 0이다. 다른 변수들과 다르게 해당 값에는 혈압=120 과 같이 연속 변수 continuous value이 들어간다.
- 검사 값 범위 그룹 Measurement range group: 검사 값이 정상 범위 이하인지 이내인지 또는 그 이상인지를 나타내는 이항 공변량이다.
- 관찰 Observation: 각 관찰 개념 ID와 선택한 시간 간격으로 공변량을 구축. 환자가 관찰 테이블의 코호트 시작일 이전에 지정된 시간 간격 동안 기록된 concept ID를 가지고 있는 경우 공변량 값은 1이며, 그렇지 않으면 0이다.
- 기기 Device: 각 기기 개념 ID와 선택한 시간 간격으로 공변량을 구축. 환자가 기기 테이블의 코호트 시작일 이전에 지정된 시간 간격 동안 기록된 개념 ID를 가지고 있는 경우 공변량 값은 1이며, 그렇지 않으면 0이다.
- 방문 수 Visit count: 각 방문과 선택한 시간 간격으로 공변량을 구축. 시간 간격 동안 기록된 방문 수를 공변량 값으로 계산한다.
- 방문 개념 수 Visit Concept Count: 각 방문 개념 ID와 선택한 시간 간격으로 공변량을 구축. 방문 유형 및 시간 간격 동안 기록된 도메인 당 기록의 수를 공변량 값으로 계산한다.
중복배제 distinct count 옵션은 도메인 및 지정한 시간 간격별로 고유한 개념 IDs의 수를 계산한다.
완료된다면, 그림 13.10과 같이 보일 것이다.
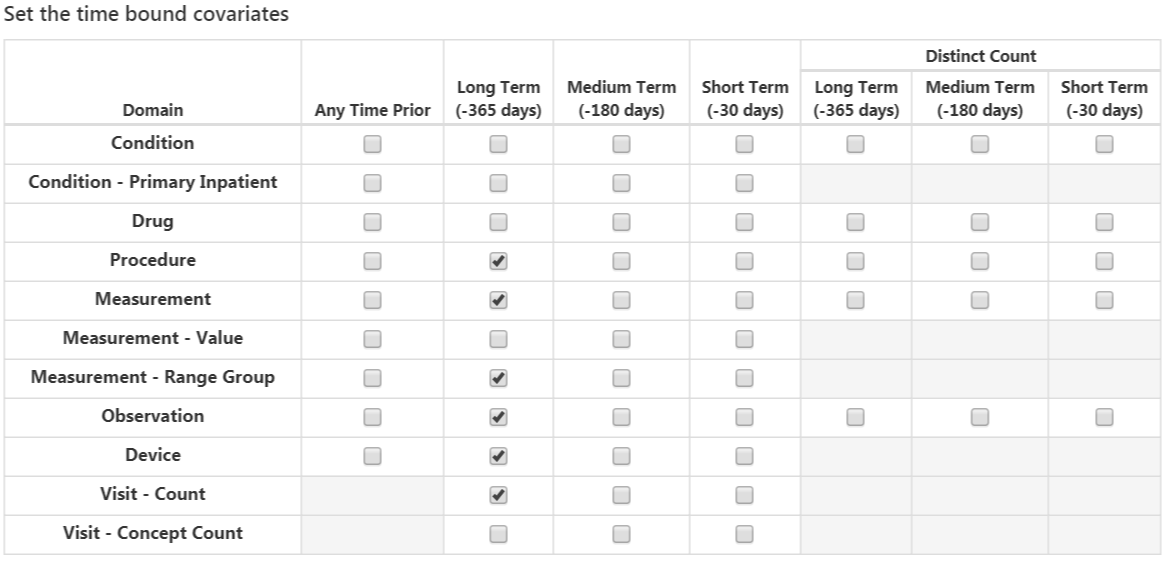
Figure 13.10: Time bound 공변량.
마지막 옵션은 흔하게 사용하는 위험 점수를 공변량으로 포함할 것인지 여부이다. 위험점수 설정을 다 마치면 그림 13.11과 같이 된다.
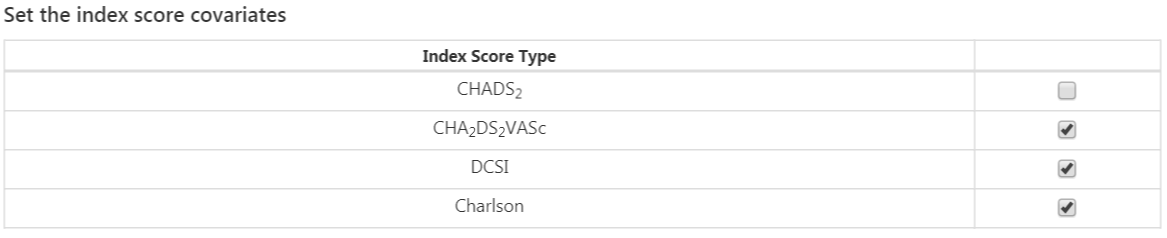
Figure 13.11: 위험 점수 공변량 설정.
모집단 설정
모집단 설정에서 추가적인 선정 기준과 위험 노출 기간 time-at-risk(TAR)을 연구 대상 군에 적용할 수 있다. 연구에서 모집단 설정을 추가하기 위해서는 “모집단 설정 추가 [Add Population Settings]” 버튼을 클릭하면 모집단 설정 창이 열린다.
옵션의 첫 번째 설정에서 위험 노출 기간을 사용자가 지정할 수 있다. 위험 노출 기간이란 관심 결과가 발생하는지 알아보는 시간 간격이다. 만약 환자의 위험 노출 기간 내에 관심 결과가 있다면 그것을 “결과가 있음 [Has outcome]”이라고 분류하고, 그렇지 않으면 “결과가 없음 [No outcome]”으로 분류할 것이다. “대상 코호트 시작 날짜를 기준으로 위험 노출 기간 시작 정의 Define the time-at-risk window start, relative to target cohort entry:” 는 대상 코호트 시작과 끝 일자에 비례하여 위험 노출 기간을 정의한다. 마찬가지로, “위험 노출 기간 종료 정의 Define the time-at-risk window end:” 는 위험 노출 기간의 끝을 결정한다.
“최소 기저(선행) 관찰 기간 Minimum lookback period applied to target cohort” 는 코호트 시작일 이전에 환자의 기저상태를 관찰하기 위해 필요한 최소의 선행 관찰 기간을 정의한다. 365일이 기본값이다. 이 숫자를 더 크게 하면 환자를 보다 완벽하게 파악할 수 있지만 (더 오래 관찰하므로), 최소 기저 관찰 기간이 짧은 환자는 연구 대상에서 제거될 것이다.
“위험 노출 기간이 없는 대상자를 제거해야 하는가? Should subjects without time at risk be removed?” 을 예로 설정하면, “최소 위험 노출 기간 Minimum time at risk” 값도 필요하다. 이를 통해 추적 기간 동안 중도 절단된 대상 (즉, 위험 노출 기간 동안 데이터베이스에서 떠난 경우) 을 제거 할 수 있다. 예를 들어, 위험 노출 기간이 코호트 시작 다음 날 부터 365일까지라면 전체 위험 노출 기간은 364 (365-1)일이다. 만약 전체 관찰 기간에 환자가 포함되길 원한다면 최소 위험 기간을 364일이라고 설정한다. 사람들이 처음 100일 동안 위험 노출 기간이 있는 것이 좋다면, 최소 위험 노출 기간을 100일로 선택한다. 위험 노출 기간의 시작이 코호트 시작으로부터 1일 후이기 때문에 코호트 시작일로부터 적어도 101일 동안 데이터베이스에 남아있는 환자가 포함될 것이다. 만약 “위험 노출 기간이 없는 대상자를 제거해야 하는가? Should subjects without time at risk be removed?”에서 아니오라고 선택하면, 모든 환자, 즉, 위험 노출 기간 동안 데이터베이스에서 이탈한 환자들까지도 연구에 포함될 것이다.
“최소 위험 기간 이내에 결과가 관찰된 사람을 포함하겠습니까? Include people with outcomes who are not observed for the whole at risk period?” 옵션은 이전 옵션과 관련된다. 예라고 설정하면 위험 노출 기간 Time-at-Risk(TAR) 중에 결과가 발생했지만, 최소 위험 기간을 채우지 못해서 탈락할 사람도 연구에 포함된다. (역자 주: 예를 들어 TAR를 365일이라고 설정했는데, 한 환자가 60일째 결과가 발생하고 그 이후 기록이 없으면 그 환자의 TAR은 60일이 되어 연구 대상에서 탈락하게 된다. 이 기능은 그것을 방지하여 설정한 TAR 이내에 결과가 발생해서 TAR이 짧은 환자도 당연히 연구에 포함될 수 있도록 하는 기능이다.)
“대상자마다 첫 번째 노출만 포함되어야 하는가? Should only the first exposure per subject be included?” 옵션은 한 환자가 코호트 시작일이 다른 여러 개의 관찰 기간을 가질 경우에 유용하다. “예”를 선택하면 환자당 가장 처음의 대상 코호트만 유지될 것이다. 그렇지 않으면 같은 환자가 한 데이터 모음에 여러 번 포함되게 된다.
“코호트 시작일 전에 해당 관심 결과가 있던 환자를 제거하시겠습니까? Remove patients who have observed the outcome prior to cohort entry?”에서 예를 선택하면 위험 노출 기간 시작일 이전에 해당 결과가 발생한 환자를 제거할 수 있다. 즉 그 관심 결과를 단 한 번도 경험하지 않은 환자를 위한 모형이 된다. 아니오를 선택한다면 환자는 코호트 시작 이전에도 해당 결과를 경험했을 수 있다. 종종, 이전에 결과를 발생한 경우에는 또다시 그런 결과가 발생할 것이라고 쉽게 예측할 수 있다.
완료된다면, 모집단 설정 화면은 그림 13.12와 같다.
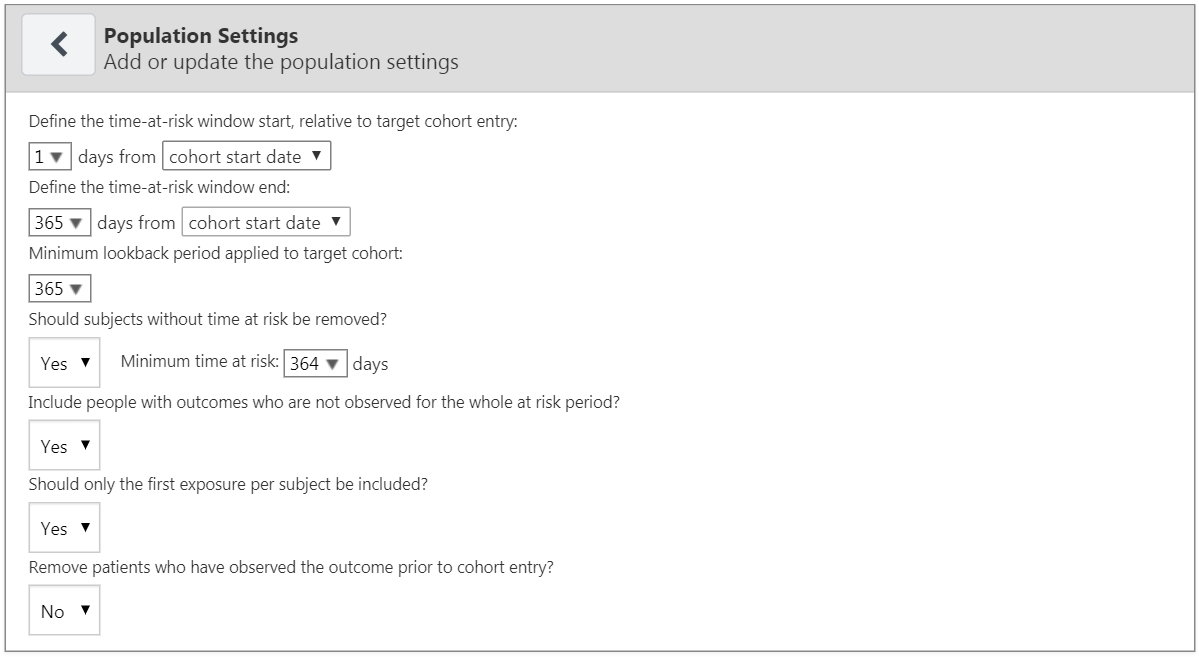
Figure 13.12: 모집단 설정.
이제 분석 설정을 끝냈으므로, 전체적인 설정 화면은 그림 13.13과 같다.
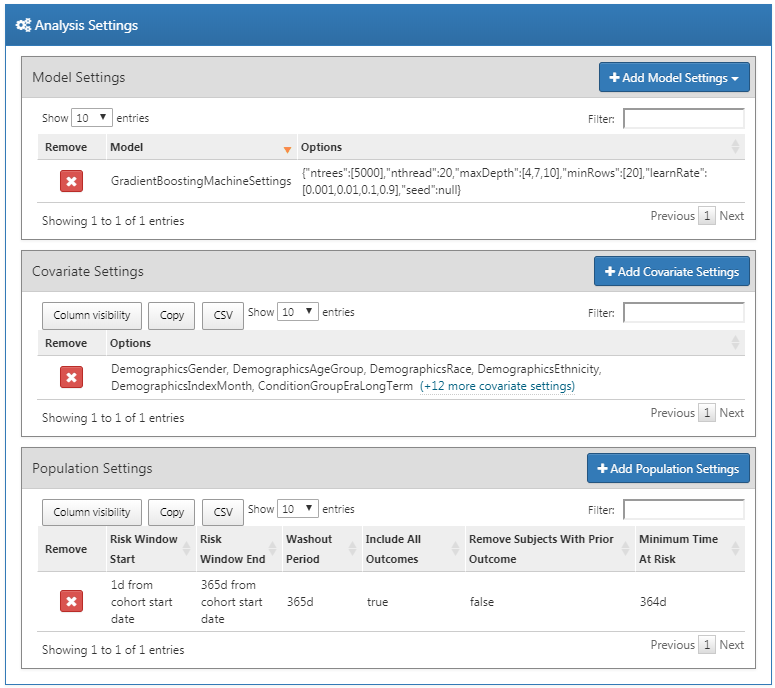
Figure 13.13: 분석 설정.
13.6.3 실행 설정
여기에는 세 가지의 옵션이 있다:
- “표본추출 실행 Perform sampling”: 표본추출을 실행할지 말지 선택할 수 있다 (기본값 = “아니오 NO”). 만약 “예 yes”라고 설정하면, 또 다른 옵션이 나타날 것이다: “몇 명의 환자를 부분집합으로 사용할 것인가?”. 여기서 표본 크기는 결정될 수 있다. 예를 들어 대상환자 수가 매우 클 경우 (예를 1,000만)을 일부만을 추출하여 모형을 만들고 구축해볼 수 있을 것이다. 하지만, 그 추출한 표본으로 만든 모형의 AUC가 0.5에 가까우면, 그 모형은 쓸모가 없으므로 버려야 할 것이다.
- “최소 공변량 발생: 만약 어떤 공변량이 지정한 값보다 적은 수의 대상에서만 발생하면 제거된다: Minimum covariate occurrence: If a covariate occurs in a fraction of the target population less than this value, it will be removed:” 여기서 최소 공변량 발생을 선택할 수 있다 (기본값 = 0.001). 전체 모집단을 대표하지 않은 드문 사건을 제거하려면 공변량 발생의 최소 임계값이 필요하다. 기본값을 사용한다면, 0.1%로 발생하는 condition, drug 등의 변수는 예측 인자에 포함되지 않을 것이다.
- “공변량 정규화 Normalize covariate”: 여기서 공변량을 표준화할 것인지 선택할 수 있다 (기본값 = yes). 대부분의 예측 인자는 이항 변수 (0 또는 1)이지만 measurement value, visit count 등은 1 이상의 값을 가질 수 있기 때문에 정규화를 해주는 것이 좋다. PLP package에서는 현재 최소-최대 정규화를 사용하고 있다. 공변량의 표준화는 LASSO 모형을 적용하기 위해서는 반드시 필요하다.
예를 들어, 그림 13.14처럼 선택한다.

Figure 13.14: 실행 설정.
13.6.4 학습 설정
여기에는 네 가지 옵션이 있다:
- “검증/학습 모음을 분할하는 방법 지정 Specify how to split the test/train set:” 학습/검증 데이터를 사람 (관심 결과에 따라 분류됨) 별로 구분할지, 시간 (모형을 학습하기 위해서는 이전 데이터를, 모형을 평가하기 위해서는 최근 데이터 사용) 별로 구분할지를 선택한다.
- “테스트 모음으로 사용될 데이터의 백분율 Percentage of the data to be used as the test set (0-100%)”: 테스트 데이터로 사용될 데이터의 백분율을 선택한다 (기본값 = 25%).
- “교차 검정에 사용된 폴드 수 The number of folds used in the cross validation”: 최적 하이퍼-파라미터 선택에 사용되는 교차 검증을 위한 폴드 수를 선택한다 (기본값 = 3).
- “사람을 기준으로 검증/학습 모음을 분할하는 경우, 사용할 시드 (선택적) The seed used to split the test/train set when using a person type testSplit” : 사용자 유형 testSplit에서 학습/검증 모음을 분할하는 데 사용되는 seed로 임의의 초기값을 선택한다.
이 예에서는 그림 13.15와 같이 선택했다.
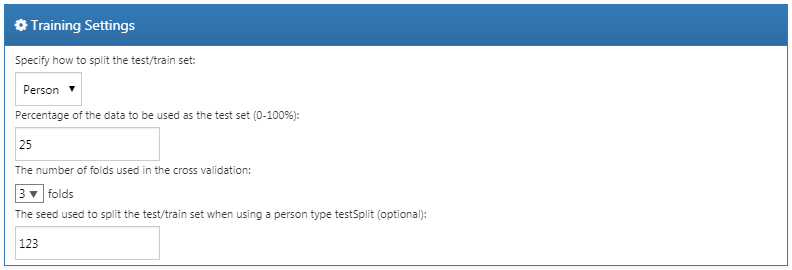
Figure 13.15: 학습 설정.
13.6.5 연구 가져오기 및 내보내기
연구를 내보내려면 “유틸리티 Utilities” 아래의 “내보내기 Export” 탭을 클릭한다. ATLAS는 연구를 실행할 때 필요한 연구 이름, 코호트 정의, 선택된 모형, 공변량, 설정과 같은 모든 데이터를 직접 복사하여 붙여넣을 수 있는 JSON 형식의 파일을 생성할 것이다. 연구를 가져오려면 “유틸리티 Utilities” 아래의 “가져오기 Import” 탭을 클릭한다. 환자 수준 예측 연구 JSON 파일의 내용을 이 창에 붙여넣은 다음 다른 탭 버튼 아래에 있는 가져오기 버튼을 클릭한다. 이 작업은 해당 연구에 대한 이전 설정을 모두 덮어쓰므로 일반적으로 비어있는 새 연구 디자인에 수행해야 한다는 점을 유의해야 한다.
13.6.6 연구 패키지 다운로드
“유틸리티 Utilities” 탭 아래의 “리뷰 & 다운로드 Review & Download” 클릭한다. “연구 패키지 다운로드 Download Study Package” 부분에서 R 패키지 이름으로 허용되지 않은 모든 문자는 ATLAS가 파일 이름에서 자동으로 제거한다는 점에 유의하여 R 패키지 이름을 작성해야 한다.  를 클릭하여 R 패키지를 로컬 폴더로 다운로드할 수 있다.
를 클릭하여 R 패키지를 로컬 폴더로 다운로드할 수 있다.
13.6.7 연구 실행
R 패키지를 실행하려면 8.4.5절에 설명된 대로 R, RStudio, Java가 설치되어 있어야 한다. 또한, 다음과 같이 R에 설치할 수 있는 PatientLevelPrediction 패키지가 필요하다:
install.packages("drat")
drat::addRepo("OHDSI")
install.packages("PatientLevelPrediction")기계학습 알고리즘의 몇몇은 추가적인 소프트웨어 설치를 요구한다. PatientLevelPrediction 패키지를 설치하는 방법에 대한 자세한 내용은 “Patient-Level Prediction Installation Guide” vignette를 참조하면 된다.
작성된 연구 R 패키지를 사용하려면 R Studio를 이용하는 것을 추천한다. 로컬에서 R Studio를 사용 중인 경우 ATLAS에서 생성한 파일의 압축을 풀고 .Rproj를 두 번 누르면 RStudio에서 열린다. RStudio 서버에서 RStudio를 실행 중인 경우 파일을 업로드하고 압축을 푼 다음  을 클릭하여 연구프로젝트를 열면 된다.
을 클릭하여 연구프로젝트를 열면 된다.
일단 R Studio에서 연구프로젝트를 열면 README 파일을 열 수 있고 그 설명을 따르면 된다. 모든 파일 경로를 시스템의 기존 경로로 변경해야 한다.
13.7 R에서의 연구 실행
ATLAS 사용하는 방법 외에도 R에서 코드를 직접 작성하여 연구를 진행할 수 있다. PatientLevelPrediction 패키지에서 제공하는 함수를 사용할 수 있다. 패키지는 OMOP CDM으로 변환된 데이터로부터 데이터 추출, 모델 구축 및 모델 평가를 가능하게 한다.
13.7.1 코호트 예시화
우선 대상 코호트와 결과 코호트를 만들어야 한다. 코호트 만드는 것은 10장에 설명되어 있다. 부록에선 대상 코호트 (부록 B.1)와 결과 코호트 (부록 B.4)에 대한 전체 정의를 제공한다. 이 예제에서 우리는 ACE 억제제 코호트를 ID 1, 혈관부종 코호트를 ID 2로 가정한다.
13.7.2 데이터 추출
우선 R에서 서버를 연결해야 한다. PatientLevelPrediction은 다양한 데이터베이스 관리 시스템(DBMS)에 필요한 구체적인 설정을 위한 createConnectionDetails. Type ?createConnectionDetails 라고 불리는 기능을 제공하는 DatabaseConnector 패키지를 사용한다. 예를 들어 이 코드를 사용하여 PostgreSQL 데이터베이스에 연결할 수 있다.
library(PatientLevelPrediction)
connDetails <- createConnectionDetails(dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
cdmDbSchema <- "my_cdm_data"
cohortsDbSchema <- "scratch"
cohortsDbTable <- "my_cohorts"
cdmVersion <- "5"마지막 4줄은 cdmDbSchema, cohortsDbSchema 및 cohortsDbTable 변수와 CDM 버전을 정의한다. 나중에 이것을 사용하여 CDM 형식의 데이터 위치, 관심 있는 코호트 위치, 사용되는 CDM 버전을 R에 입력한다. Microsoft SQL의 경우 데이터베이스 스키마는 데이터와 스키마 모두 지정해야 한다. 예를 들어 cdmDbSchema <- “my_cdm_data.dbo”이다.
먼저 코호트 항목 수를 세어 코호트 생성이 되었는지 확인하는 것이 좋다:
sql <- paste("SELECT cohort_definition_id, COUNT(*) AS count",
"FROM @cohortsDbSchema.cohortsDbTable",
"GROUP BY cohort_definition_id")
conn <- connect(connDetails)
renderTranslateQuerySql(connection = conn,
sql = sql,
cohortsDbSchema = cohortsDbSchema,
cohortsDbTable = cohortsDbTable)## cohort_definition_id count
## 1 1 527616
## 2 2 3201이제 PatientLevelPrediction을 통해 분석에 필요한 모든 데이터를 추출할 수 있다. 공변량은 FeatureExtraction 패키지를 사용하여 추출된다. FeatureExtraction 패키지에 대한 자세한 내용은 해당 vignettes에서 볼 수 있다. 예제 연구에서는 다음과 같은 설정을 사용하기로 하였다:
covariateSettings <- createCovariateSettings(
useDemographicsGender = TRUE,
useDemographicsAge = TRUE,
useConditionGroupEraLongTerm = TRUE,
useConditionGroupEraAnyTimePrior = TRUE,
useDrugGroupEraLongTerm = TRUE,
useDrugGroupEraAnyTimePrior = TRUE,
useVisitConceptCountLongTerm = TRUE,
longTermStartDays = -365,
endDays = -1)데이터를 추출의 마지막 단계는 getPlpData 함수를 실행하고 코호트가 저장되는 데이터베이스 스키마, 코호트와 결과를 위한 코호트 정의 ID, 해당 사람이 데이터에 포함되도록 관찰되어야 하는 코호트 색인 일자 이전의 최소 일자인 최소 휴약기 washout period와 같은 연결 세부사항을 입력하는 것이고 마지막으로 이전에 생성된 공변량 설정을 입력하는 것이다.
plpData <- getPlpData(connectionDetails = connDetails,
cdmDatabaseSchema = cdmDbSchema,
cohortDatabaseSchema = cohortsDbSchema,
cohortTable = cohortsDbSchema,
cohortId = 1,
covariateSettings = covariateSettings,
outcomeDatabaseSchema = cohortsDbSchema,
outcomeTable = cohortsDbSchema,
outcomeIds = 2,
sampleSize = 10000
)getPlpData 함수에는 추가적으로 많은 매개변수 parameter가 있으며, PatientLevelPrediction 매뉴얼에 모두 정리되어 있다. 결과상 나오는 plpData object는 R 에서 큰 데이터 사용시 메모리를 효율적으로 관리하기 위하여 ff패키지를 사용한다. plpData object를 생성하는 것은 꽤나 시간이 걸릴 수 있어서 (머신 러닝 모델 학습 및 교차검증, 결과 분석까지 완료해야하기 때문에), 나중을 위해 저장하는 것이 좋다. plpData 데이터는 ff 패키지 기반으로 만들어지기 때문에, 일반적인 save 함수로는 저장할 수 없다. 대신에, savePlpData 함수를 이용한다:
savePlpData(plpData, "angio_in_ace_data")loadPlpData() 를 이용하여 PLP 모델을 불러올 수 있다.
13.7.3 추가 포함 기준
최종 연구 모집단은 이전에 정의된 2개의 코호트에서 추가적인 제약조건을 적용하여 얻어진다. 예를 들어 최소 위험에 노출된 시간을 적용할 수 있으며 (requireTimeAtRisk, minTimeAtRisk), 이것이 결과를 가진 환자에게도 적용되는지 여부를 지정할 수 있다 (includeAllOutcomes). 또한 여기서 대상 코호트 시작에 관련된 위험 기간 risk window의 시작과 끝을 지정한다. 예를 들어 위험 코호트가 시작된 후 30일부터 위험 기간으로 시작하고 1년 후 종료하려면 riskWindowStart = 30, riskWindowStart = 365로 설정할 수 있다. 때에 따라 위험 기간은 코호트 종료일에 시작해야 한다. addExposureToStart = Ture로 설정하면 코호트 (노출) 시간을 시작일에 추가할 수 있다.
아래의 예에서는 연구를 위해 정의한 모든 설정을 시행한 것이다:
population <- createStudyPopulation(plpData = plpData,
outcomeId = 2,
washoutPeriod = 364,
firstExposureOnly = FALSE,
removeSubjectsWithPriorOutcome = TRUE,
priorOutcomeLookback = 9999,
riskWindowStart = 1,
riskWindowEnd = 365,
addExposureDaysToStart = FALSE,
addExposureDaysToEnd = FALSE,
minTimeAtRisk = 364,
requireTimeAtRisk = TRUE,
includeAllOutcomes = TRUE,
verbosity = "DEBUG"
)13.7.4 모델 개발
알고리즘의 설정 기능에서 사용자는 각 하이퍼-파라미터에 대한 적합한 값의 목록을 지정할 수 있다. 하이퍼-파라미터에서 가능한 모든 조합은 학습 모음에 교차 검증을 사용하는 이른바 그리드서치에 포함된다. 만일 사용자가 어떤 값도 지정하지 않으면 기본값이 사용된다.
예를 들어 점진적 부스팅 머신에 다음 설정을 사용하는 경우: ntrees = c(100,200), maxDepth = 4 그리드서치는 점진적 부스팅 머신 알고리즘을 다른 하이퍼-파라미터의 기본 설정을 더한 ntrees = 100과 maxDepth = 4 , 다른 하이퍼-파라미터의 기본 설정을 더한 ntrees = 200과 maxDepth = 4에 적용할 것이다. 최고의 교차 검증 실행을 이끄는 하이퍼-파라미터는 마지막 모델로 선택될 것이다. 이 문제를 위해 여러 하이퍼-파라미터값을 가지고 점진적 부스팅 머신을 만들기로 하였다:
gbmModel <- setGradientBoostingMachine(ntrees = 5000,
maxDepth = c(4,7,10),
learnRate = c(0.001,0.01,0.1,0.9))runPlp 함수는 모델을 훈련하고 평가하기 위해 모집단, plpData 및 모델 설정을 사용한다. 데이터를 75% ~ 25%로 분할하기 위해 testSplit(사람/시간)과 testFraction 파라미터를 사용하고 환자 수준 예측 파이프라인을 실행할 수 있다:
gbmResults <- runPlp(population = population,
plpData = plpData,
modelSettings = gbmModel,
testSplit = 'person',
testFraction = 0.25,
nfold = 2,
splitSeed = 1234)패키지 안에 R xgboost 패키지를 사용하여 데이터의 75%를 사용하는 점진적 부스팅 머신 모델을 맞추고 나머지 25%에 대해 모델을 평가한다. 결과 데이터 구조는 모델과 성능에 대한 정보가 포함되어 있다.
runPlp 함수에는 기본적으로 TRUE로 설정된 plpData, plpResults, plpplots, evaluation 등을 저장할 수 있는 몇 가지 파라미터가 있다.
다음을 사용하여 모델을 저장할 수 있다:
savePlpModel(gbmResults$model, dirPath = "model")다음과 같이 모델을 불러올 수 있다:
plpModel <- loadPlpModel("model")또한, 다음을 사용하여 전체 결과 구조를 저장할 수 있다:
savePlpResult(gbmResults, location = "gbmResults")다음과 같이 전체 결과 구조를 불러 올 수도 있다:
gbmResults <- loadPlpResult("gbmResults")13.7.5 내부 검증
연구를 실행하면 runPlp함수는 학습/테스트 모음에서 학습된 모델과 학습/테스트 모음에서 모델의 평가를 해준다. viewPLP(runPLP = gbmResults)를 실행하여 인터액티브하게 결과를 볼 수 있다. 이것은 대화식 그림을 포함하여 프레임워크에 생성한 모든 측정값을 볼 수 있는 Shiny 앱을 열 것이다 (Shiny 애플리케이션 섹션의 그림 13.16 참조).
모든 평가 그림을 폴더에 생성하고 저장하려면 다음 코드를 실행하면 된다:
plotPlp(gbmResults, "plots")도표는 13.4.2장에서 더욱 자세하게 기술된다.
13.7.6 외부 검증 External Validation
항상 외적 타당도를 수행하는 것을 권장한다. 즉 가능한 많은 새로운 데이터에 최종모델을 적용하고 성능을 평가해야 한다. 여기서 이미 두 번째 데이터베이스에서 데이터 추출이 수행되어 newData 폴더에 저장되었다고 가정한다. 이전에 장착된 모델을 model 폴더로부터 로딩한다.
# load the trained model
plpModel <- loadPlpModel("model")
#load the new plpData and create the population
plpData <- loadPlpData("newData")
population <- createStudyPopulation(plpData = plpData,
outcomeId = 2,
washoutPeriod = 364,
firstExposureOnly = FALSE,
removeSubjectsWithPriorOutcome = TRUE,
priorOutcomeLookback = 9999,
riskWindowStart = 1,
riskWindowEnd = 365,
addExposureDaysToStart = FALSE,
addExposureDaysToEnd = FALSE,
minTimeAtRisk = 364,
requireTimeAtRisk = TRUE,
includeAllOutcomes = TRUE
)
# apply the trained model on the new data
validationResults <- applyModel(population, plpData, plpModel)또한 필요한 데이터를 추출하는 외부 검증을 더욱 쉽게 하기 위해 externalValidatePLP 함수를 제공한다. result <- runPlp(...)을 실행했다고 가정했을 때 모델에 필요한 데이터를 추출하여 새 데이터에 대해 평가할 수 있다. 검증 코호트가 ID 1과2가 있는 테이블 mainschema.dob.cohort과 CDM 데이터가 스키마 cdmschema.dob 라고 가정한다:
valResult <- externalValidatePlp(
plpResult = result,
connectionDetails = connectionDetails,
validationSchemaTarget = 'mainschema.dob',
validationSchemaOutcome = 'mainschema.dob',
validationSchemaCdm = 'cdmschema.dbo',
databaseNames = 'new database',
validationTableTarget = 'cohort',
validationTableOutcome = 'cohort',
validationIdTarget = 1,
validationIdOutcome = 2
)모델을 검증할 데이터베이스가 여러 개 있는 경우 다음을 실행 할 수 있다:
valResults <- externalValidatePlp(
plpResult = result,
connectionDetails = connectionDetails,
validationSchemaTarget = list('mainschema.dob',
'difschema.dob',
'anotherschema.dob'),
validationSchemaOutcome = list('mainschema.dob',
'difschema.dob',
'anotherschema.dob'),
validationSchemaCdm = list('cdms1chema.dbo',
'cdm2schema.dbo',
'cdm3schema.dbo'),
databaseNames = list('new database 1',
'new database 2',
'new database 3'),
validationTableTarget = list('cohort1',
'cohort2',
'cohort3'),
validationTableOutcome = list('cohort1',
'cohort2',
'cohort3'),
validationIdTarget = list(1,3,5),
validationIdOutcome = list(2,4,6)
)13.8 결과 보급
13.8.1 모델 성능
viewPlp 함수를 사용하면 예측 모델의 성능을 탐색하기에 가장 쉽다. 이 함수는 결과 객체를 입력값으로 한다. R에서 모델을 개발하는 경우에는 runPlp의 결과를 입력값으로 사용할 수 있다. ATLAS로 만든 연구 패키지를 이용한다면 모델 중 하나를 로딩해야 한다 (이 예제에서는 Analysis_1을 로딩할 것이다):
plpResult <- loadPlpResult(file.path(outputFolder,
'Analysis_1',
'plpResult'))여기서 “Analysis_1”은 앞에서 설명한 분석에 해당한다.
이후에 다음을 실행하여 Shiny 앱을 시작할 수 있다:
viewPlp(plpResult)Shiny 앱은 그림 13.16에서 볼 수 있듯이 훈련 모음과 평가 모음에 있는 성능 지표의 요약본을 보여준다. 결과를 보면 훈련 모음에서 AUC가 0.78이고 평가 모음에서는 0.74까지 떨어지는 것을 보여준다. 평가 모음 AUC가 좀 더 정확한 측정값이다. 전반적으로 이 모델은 ACE 억제제를 처음 사용하는 사용자에서 결과를 예측할 수 있을 것처럼 보이지만 훈련 모음이 평가 모음보다 성능이 더 좋기 때문에 다소 과적합 되었다. ROC 도표는 그림 13.17에 제시되어 있다.
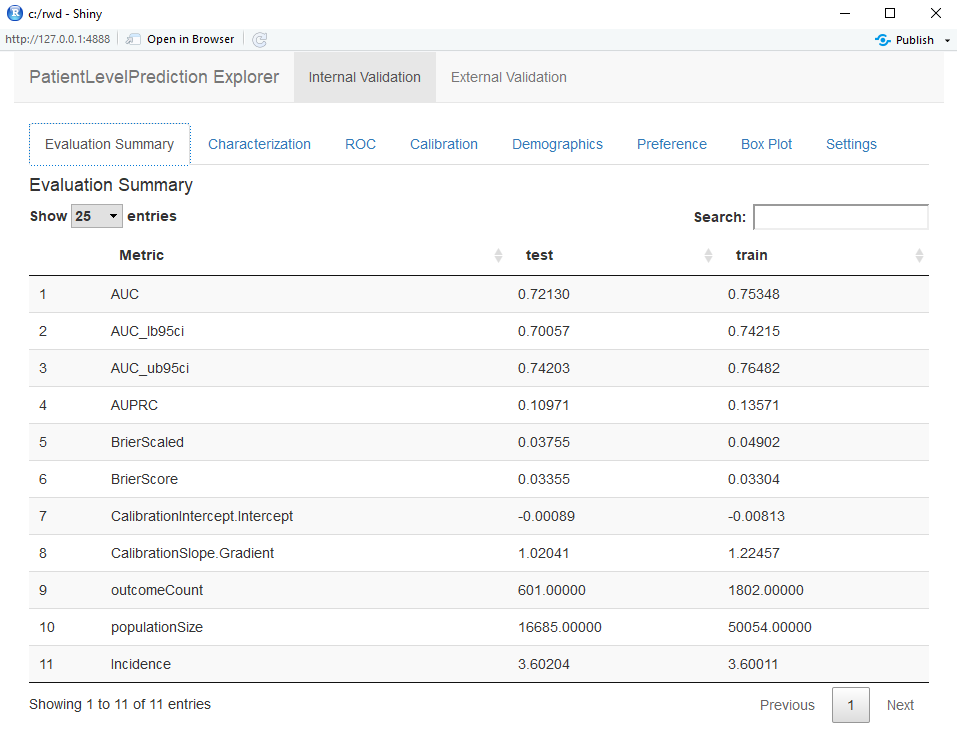
Figure 13.16: Shiny 앱에서의 요약 평가 통계.
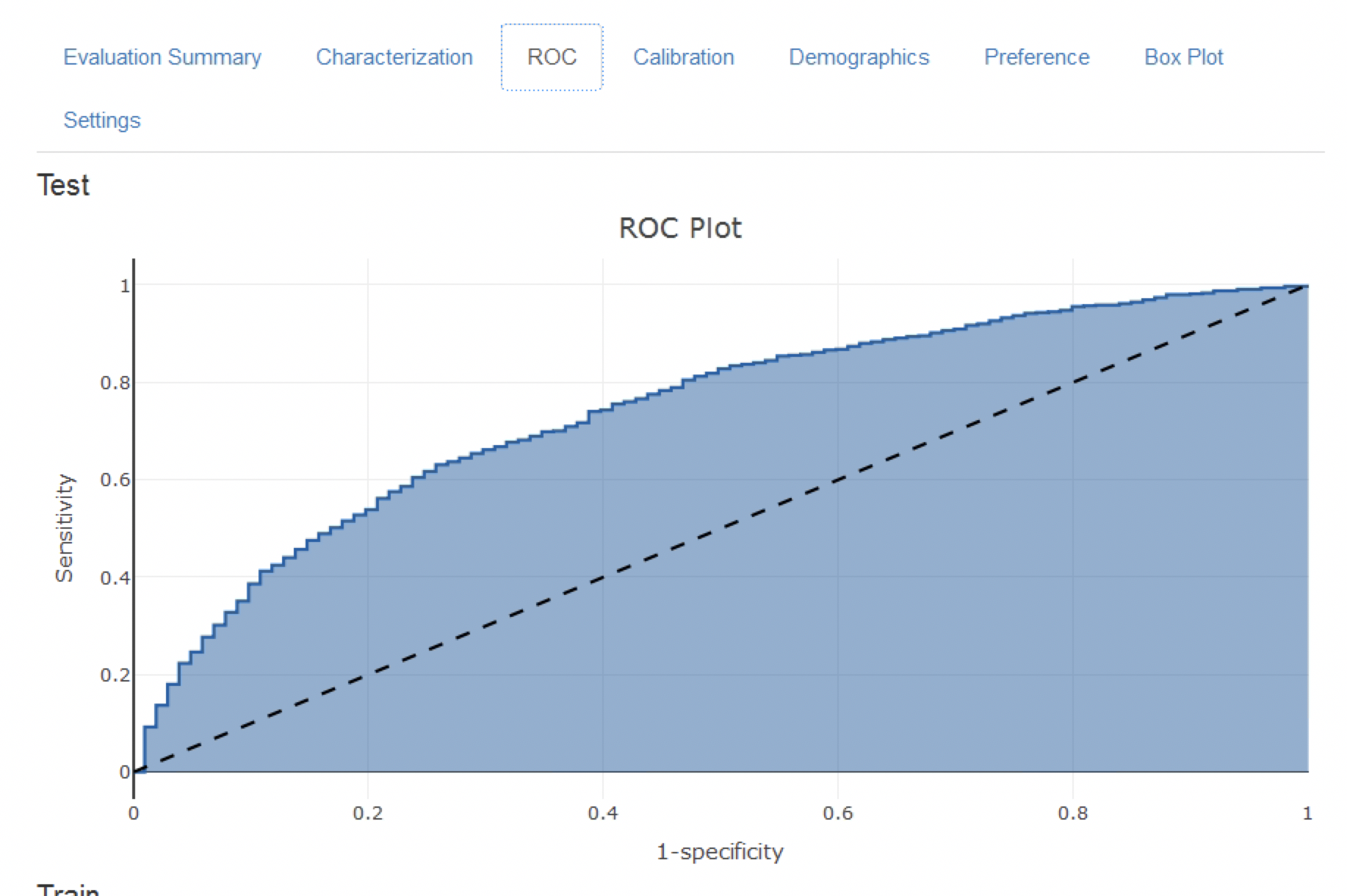
Figure 13.17: ROC 도표.
그림 13.18에 있는 모델 적합 도표는 점들이 대각선 주위에 있을 때 일반적으로 관찰된 위험이 예측된 위험과 일치함을 보여준다. 그러나 그림 13.19에 있는 인구통계학적 그래프는 하늘색 선 (예측위험)이 40세 미만의 적색선 (관측된 위험)과 다르기 때문에 모델이 젊은 환자에 대해 잘 보정되지 않았음을 보여준다. 이것은 대상 모집단 target population에서 40대 미만을 제거해야 할 필요가 있다는 것을 보여 준다 (젊은 환자에서는 관찰된 위험이 거의 0이므로).
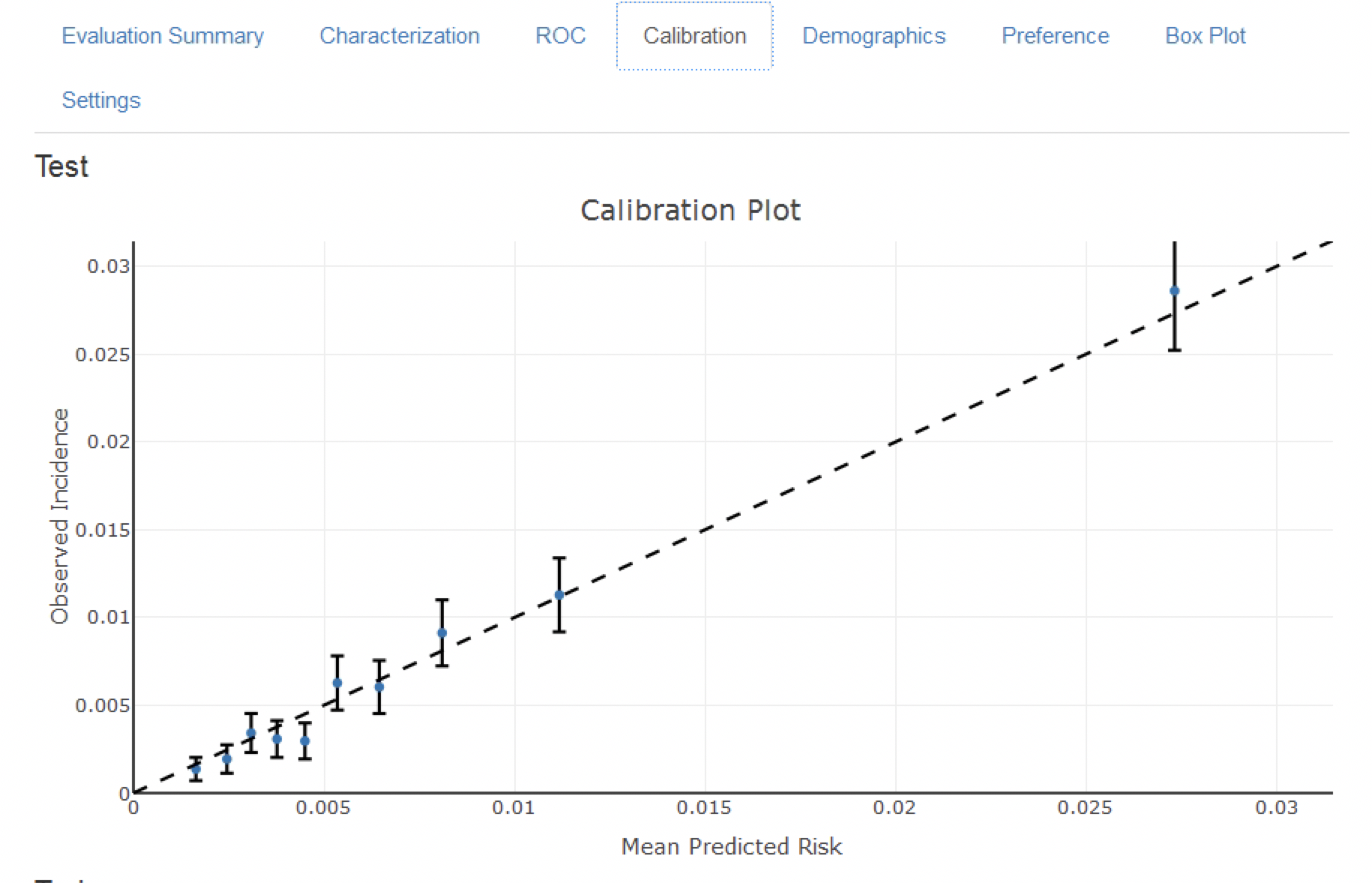
Figure 13.18: 모델 보정
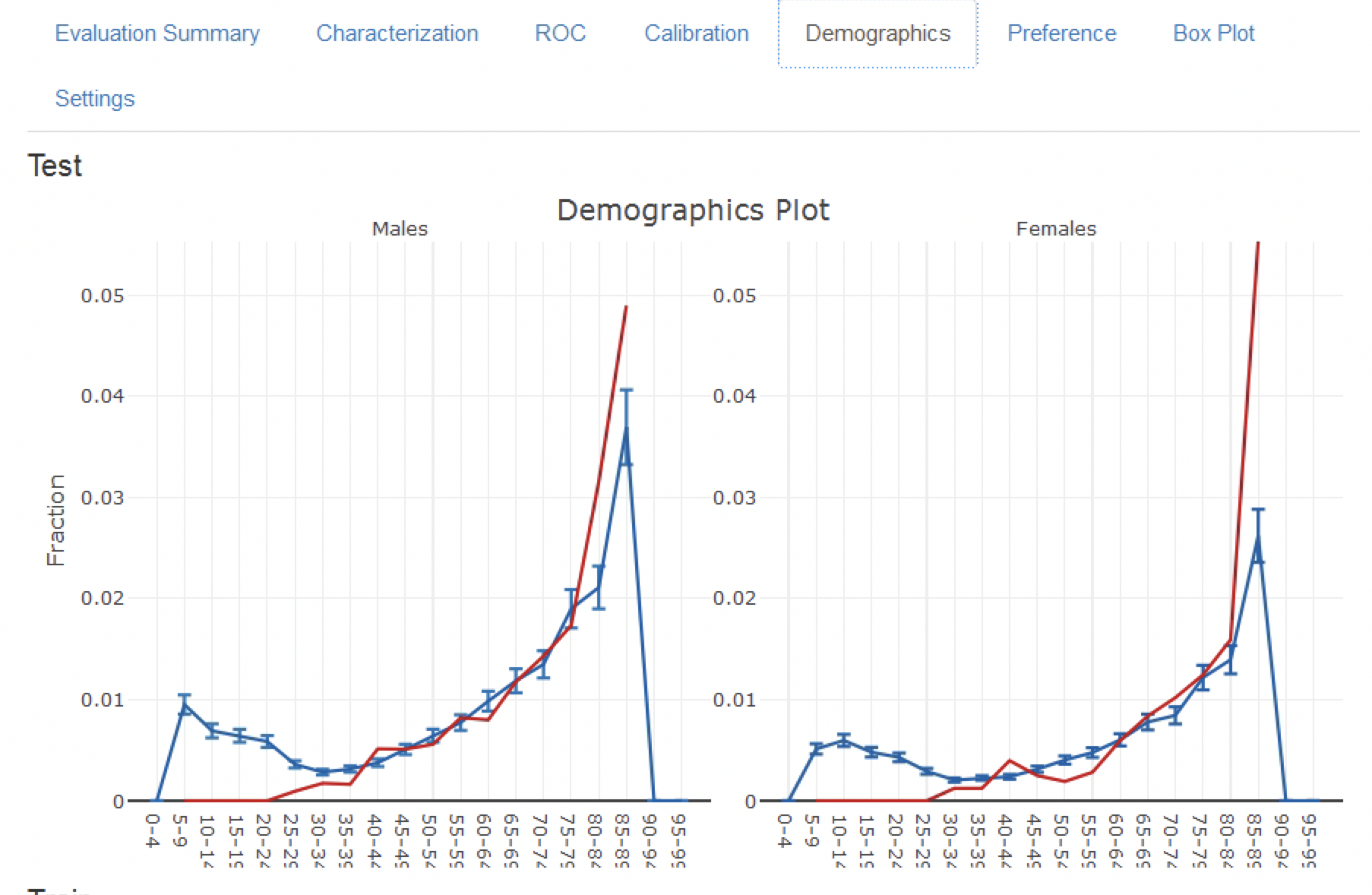
Figure 13.19: 모델의 인구 통계학적 보정
마지막으로, 손실 도표 attrition plot는 선정/제외 기준에 기초해서 라벨링 된 데이터에서 손실된 환자 수를 나타낸다 (그림 13.20 참조). 아래 표는 대상 모집단에서 많은 연구대상자가 위험 노출 기간 조건 (1년)을 만족하지 못해서 손실됐다는 것을 보여준다. 흥미롭게도, 결과가 발생한 환자는 상대적으로 적게 손실되었다.
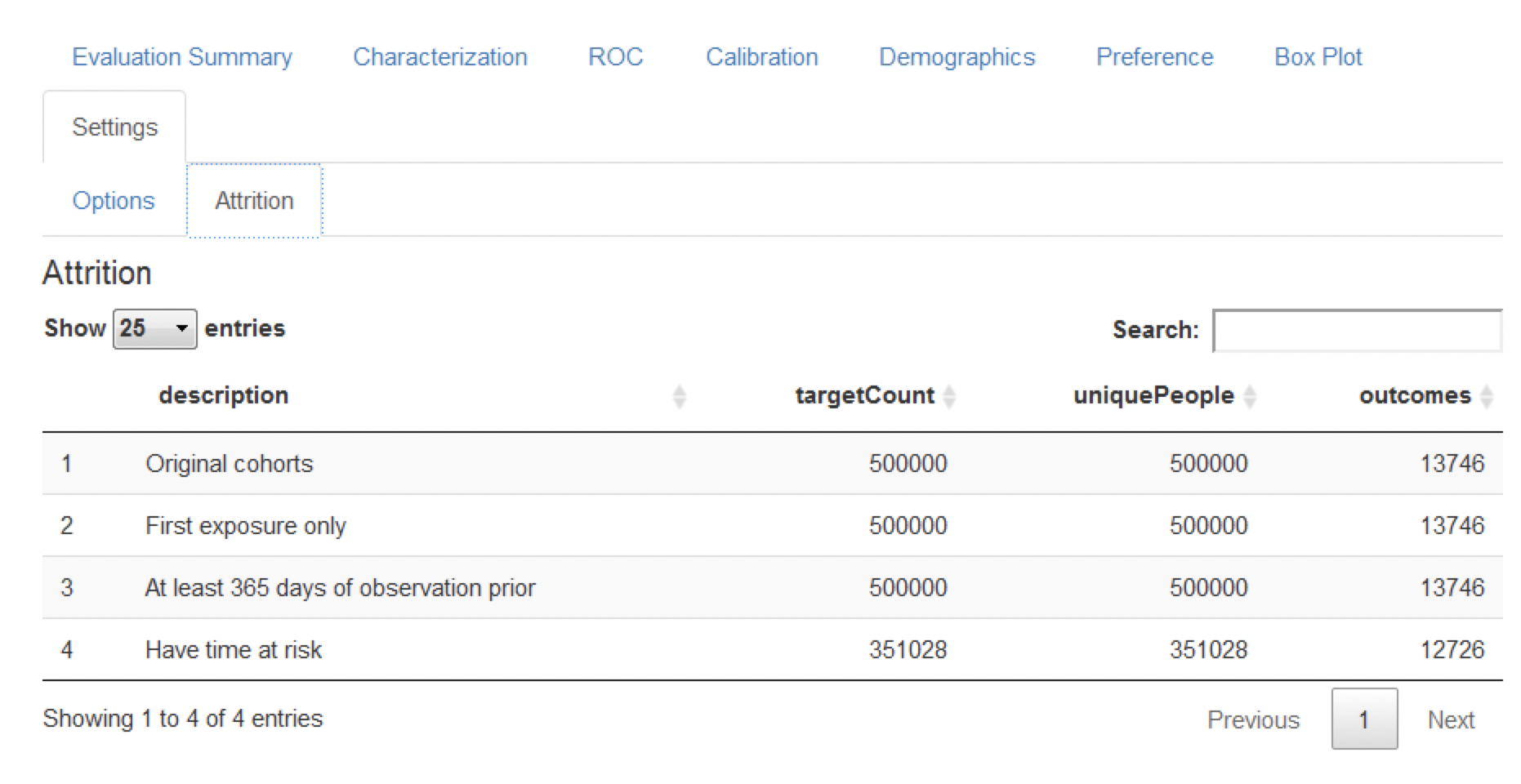
Figure 13.20: 분석한 예측 문제에서의 손실 도표
13.8.2 모델간 비교
ATLAS가 생성한 연구 패키지는 다른 예측 문제에 대해 다른 예측 모델을 생성하고 평가하는데 사용할 수 있다. 이를 위해서 특별히, 연구 패키지가 생성한 결과에 대해 여러 모델을 볼 수 있도록 Shiny 앱을 개발하였다. 앱을 시작하기 위해서는 viewMultiplePlp(outputFolder)라고 실행하는데 outputFolder는 execute 명령을 실행할 때 지정된 분석 결과를 저장하는 경로이다 (우리 예제에서는 “Analysis_1”이라는 하위 폴더가 포함되어야 한다).
모델 요약 보기 및 설정
대화형 Shiny 앱은 그림 13.21과 같이 요약 페이지를 보여준다.
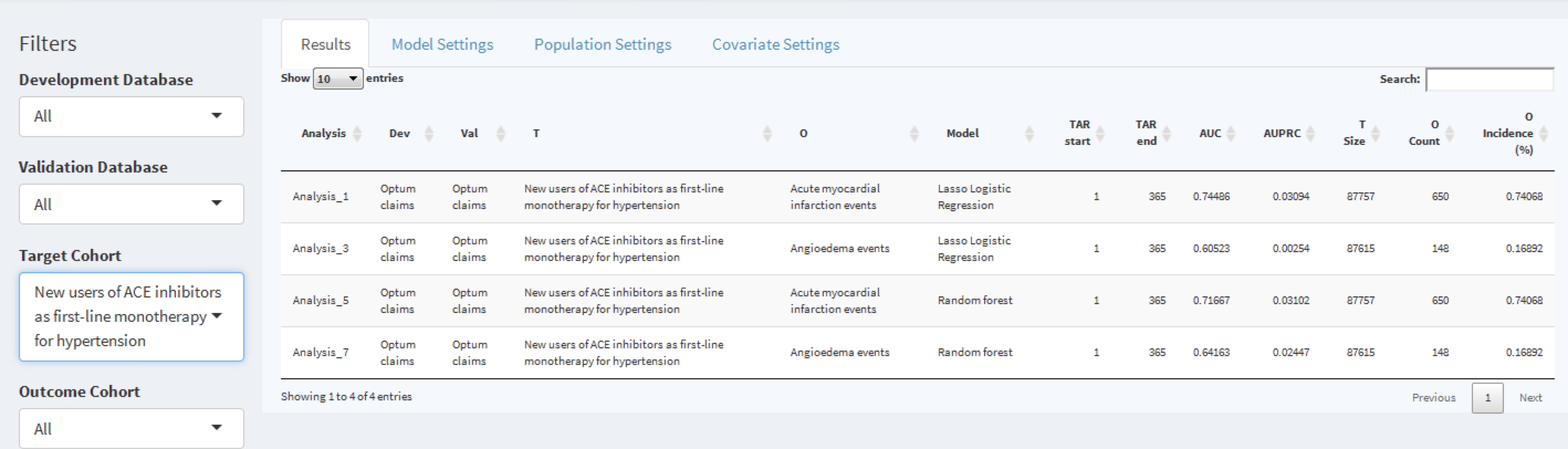
Figure 13.21: 학습된 각 모델에 대해서 핵심 성능 지표를 보여주는 shiny 요약 페이지
요약 페이지 테이블에는 아래 내용이 있다:
- 모델에 관한 기본 정보 (예를 들어, 데이터베이스 정보, 분류 유형, 위험 노출 기간 설정, 대상 모집단, 결과 명)
- 대상 모집단 수와 결과 발생률
- 판별 지표 discrimination metrics: AUC, AUPRC
표 왼쪽에는 필터 옵션이 있는데 여기서는 해당 코호트의 개발/검증 데이터베이스, 모델 유형, 관심 있는 위험 노출 기간 설정이 있다. 예를 들어, 대상 모집단 “고혈압의 일차 단일 요법으로 ACE 억제제의 신규 사용자 [New users of ACE inhibitors as first line mono-therapy for hypertension”에 해당하는 모델을 선택하려면 대상 코호트[Target Cohort] 옵션을 선택하면 된다.
해당 행을 클릭하여 모델을 탐색하면, 선택된 행이 강조 표시된다. 행을 선택하면 모델 설정 Model Settings 탭을 클릭하여 모델을 개발할 때 사용되는 모델 설정을 탐색할 수 있다:
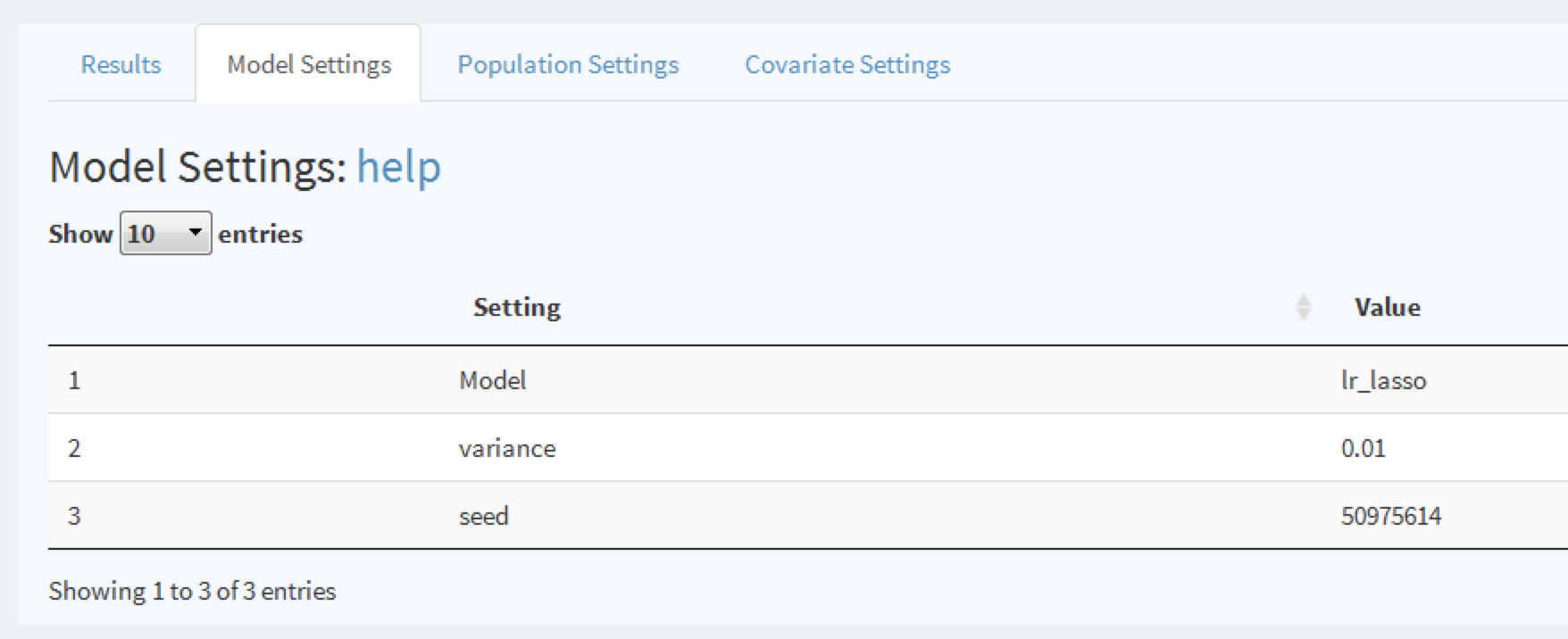
Figure 13.22: 모델을 개발에 사용된 설정 보기.
비슷하게, 다른 탭에서 모델을 생성하는데 사용된 모집단과 공변량 설정을 탐색할 수 있다.
모델 성능 보기
일단 모델 행을 선택하면 모델 성능도 볼 수 있다. 임계값 성능 요약을 보기 위하여  를 클릭하면 그림 13.23처럼 나타난다.
를 클릭하면 그림 13.23처럼 나타난다.
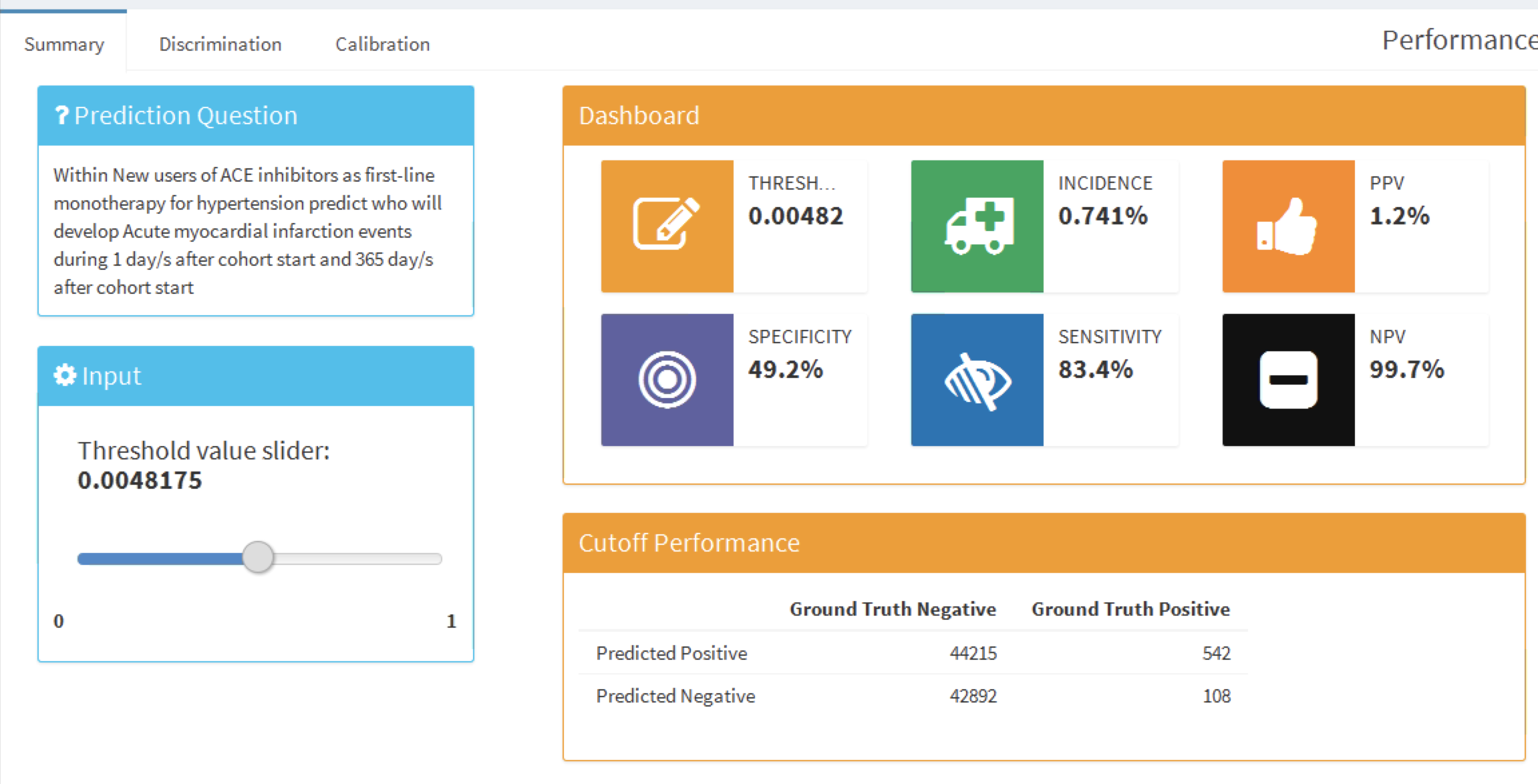
Figure 13.23: 설정된 임계값에 따른 성능 측정값 요약표
이 요약 보기에는 표준화한 형식의 예측 질문, 임계값 선택기 threshold selector 및 대시보드에 양성예측도 PPV, 음성예측도 NPV, 민감도 및 특이도 (13.4.2절 참조)와 같은 주요 임계값 기반 지표를 포함하고 있다. 그림 13.23에서 임계값 0.00482에서 민감도는 83.4% (다음 1년간 결과가 발생한 83.4% 환자는 0.00482 이상의 위험을 가지고 있다) 이고 PPV는 1.2% (0.00482보다 크거나 같은 위험을 가진 환자의 1.2%는 다음 1년간 그 결과가 발생한다) 이다. 연간 결과 발생률이 0.741%이므로 위험이 0.00482 이상인 환자를 식별하는 것은 모집단의 평균 위험의 거의 두 배 (1.2%)가 되는 환자그룹을 찾을 수 있다는 것이다. 슬라이더를 사용하여 임계값을 조정할 수 있다.
모델의 전체적인 예측력을 보려면 “Discrimination” 탭을 클릭하면 ROC 도표, 정밀도-검출률 precision-recall 도표, 분포 도표를 볼 수 있다. 그림의 수직선은 선택한 임계값 포인트에 해당한다. 그림 13.24는 ROC와 정밀도-검출률 도표를 보여준다. ROC 도표는 모델이 1년 이내에 결과가 생길 사람과 그렇지 않은 사람을 구별할 수 있음을 보여준다. 그러나 결과 발생률이 낮다는 것은 거짓 양성률이 높다는 것을 의미하기 때문에 정밀도-재현율 도표를 보면 성능이 그다지 인상적이지는 않아 보인다.
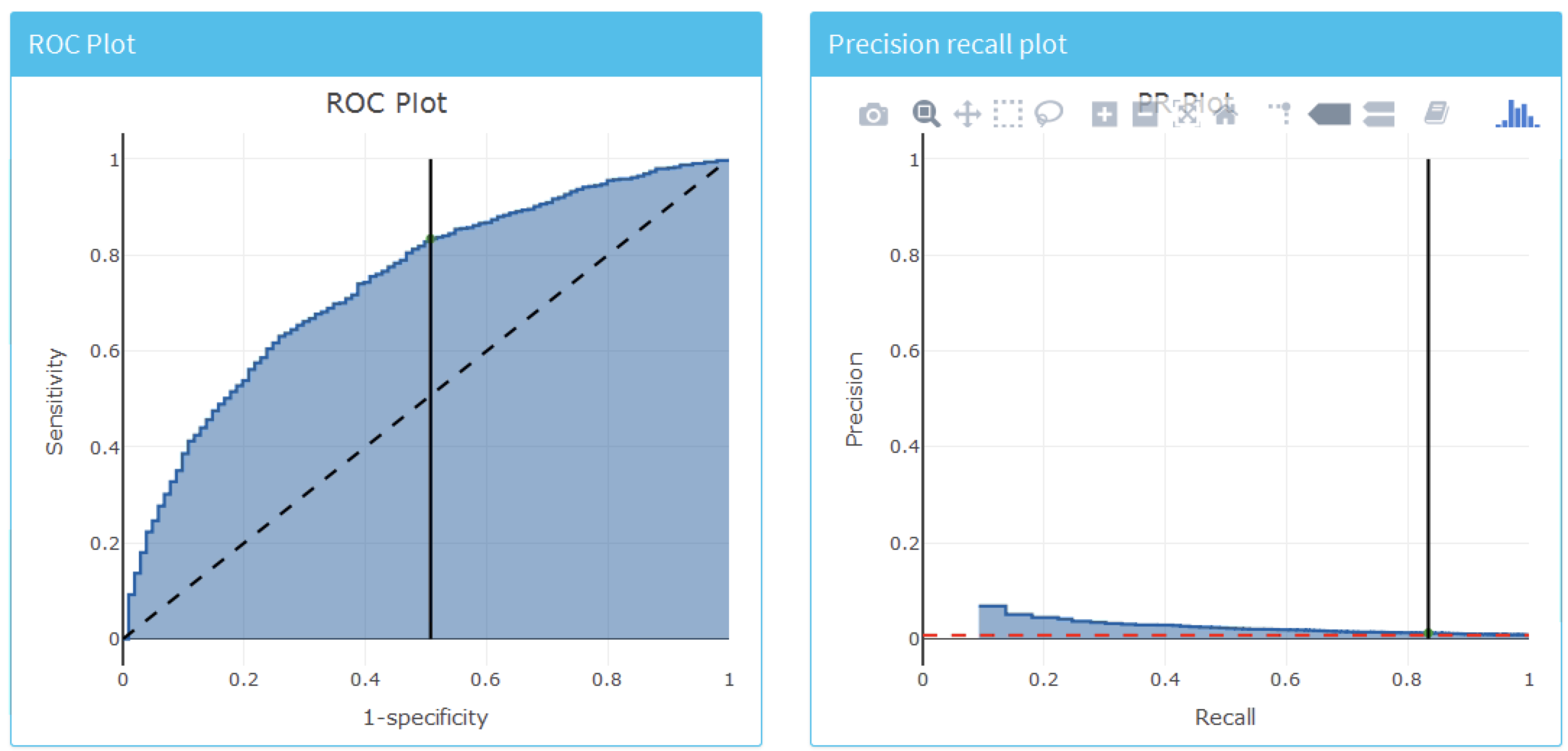
Figure 13.24: 모델의 전체적인 판별도를 평가하기 위한 ROC와 정밀도-검출률 도표.
그림 13.25는 예측과 선호 점수 분포를 보여준다.
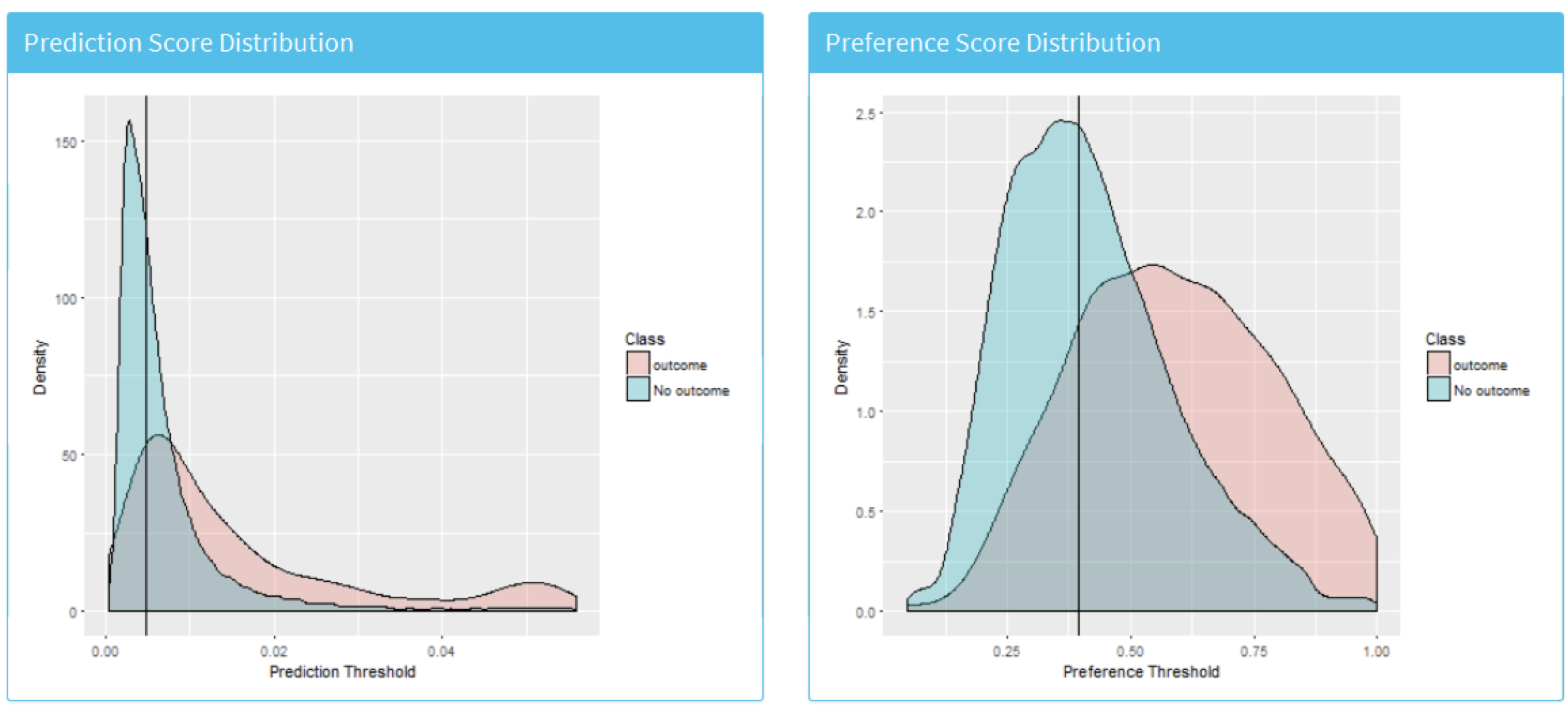
Figure 13.25: 결과가 생긴 군과 생기지 않은 군에서 예측 위험 분포. 이 도표간에 중복이 심할 수록 판별력은 나빠진다.
마지막으로 “Calibration” 탭을 클릭하여 모델의 보정 calibration을 조사할 수 있다. 그림 13.26은 보정 도표 calibration plot와 인구통계학적 보정을 보여준다.
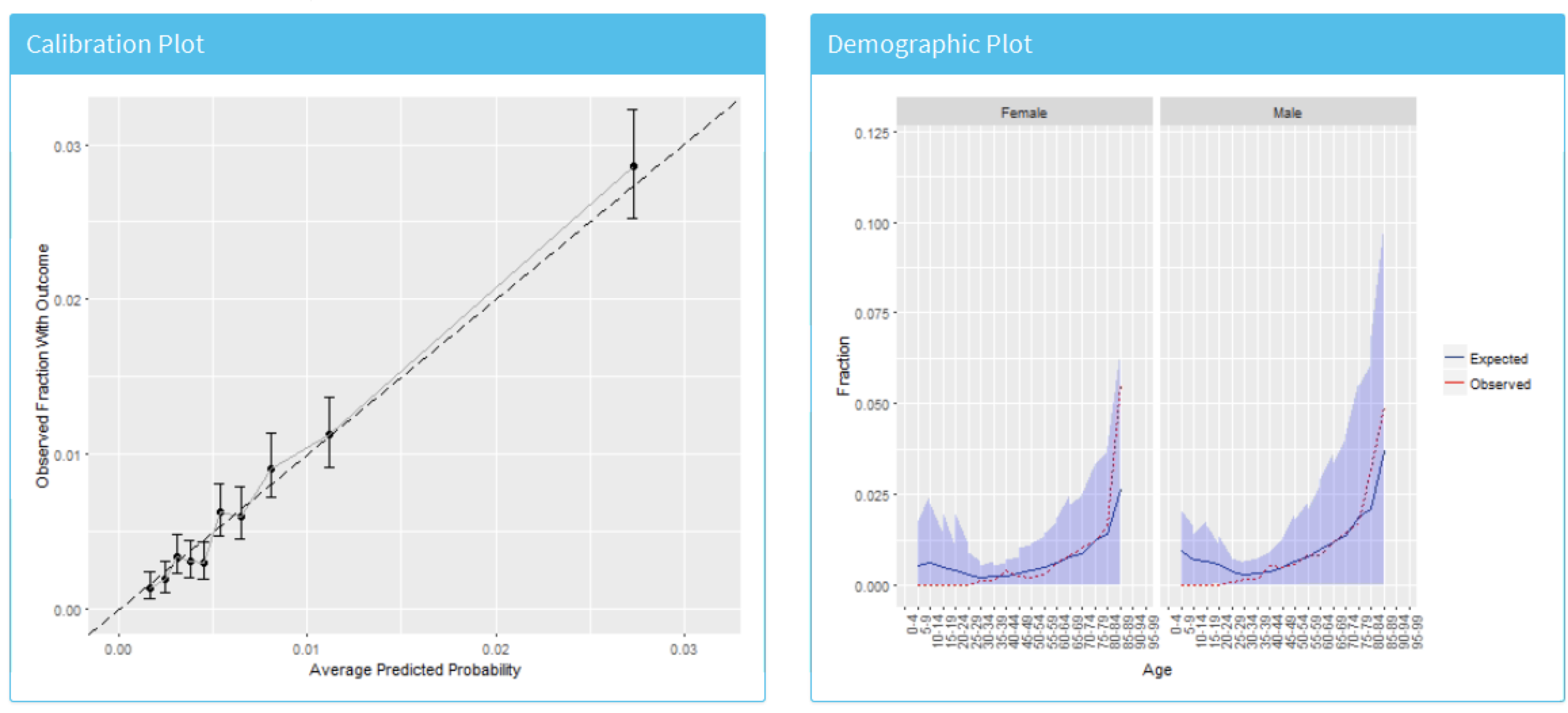
Figure 13.26: 위험 층화 보정 및 인구통계학적 보정
평균 예측 위험은 1년 이내 결과를 경험한 관측된 비율과 일치하는 것으로 나타나므로 모델은 잘 보정되어 있다. 흥미롭게도 인구통계학적 보정을 보면 젊은 환자에게서 기대 선이 관찰 선보다 더 높게 나왔다는 것을 보여주고 있어서, 모델이 젊은 연령 집단에 있어서 위험을 실제보다 더 높게 예측하는 것을 알 수 있다. 이것은 젊은 환자나 고령 환자를 위한 모델을 분리하여 별도로 개발해야 할 수도 있다는 것을 의미한다.
모델 보기
최종 모델을 확인하려면 왼쪽 메뉴에서  옵션을 선택하면 된다. 옵션을 선택하면 그림 13.27과 13.28과 같이 모델의 각 변수에 대한 그래프와 공변량에 대한 요약표를 볼 수 있다. 변수 그래프는 범주형 변수와 연속형 변수로 구분된다. x축은 결과가 없는 환자의 유병률/평균이고 y축은 결과가 있는 환자의 유병률/평균이다. 그래프를 보면 결과가 있는 환자는 대각선 아래보다 대각선 위에 더 많이 분포하고 있다.
옵션을 선택하면 된다. 옵션을 선택하면 그림 13.27과 13.28과 같이 모델의 각 변수에 대한 그래프와 공변량에 대한 요약표를 볼 수 있다. 변수 그래프는 범주형 변수와 연속형 변수로 구분된다. x축은 결과가 없는 환자의 유병률/평균이고 y축은 결과가 있는 환자의 유병률/평균이다. 그래프를 보면 결과가 있는 환자는 대각선 아래보다 대각선 위에 더 많이 분포하고 있다.
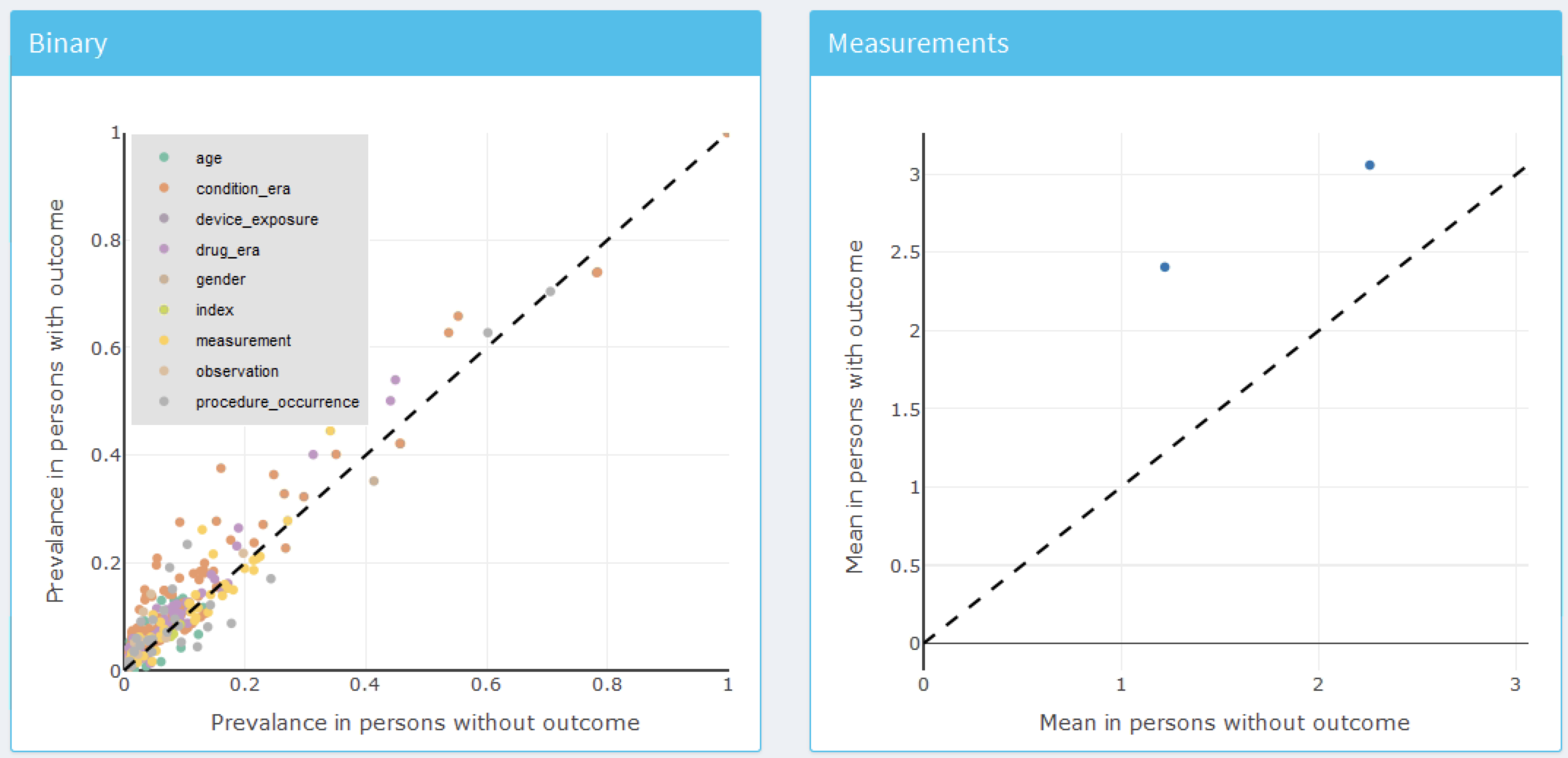
Figure 13.27: 모델 요약 도표. 각 점은 모델에 포함된 변수에 해당한다.
그림 13.28의 표에는 공변량과 공변량으로 사용될 수 있는 모든 변수의 값 (일반 선형 모델을 사용할 경우 계수, 그렇지 않을 경우 변수 중요도), 그리고 결과 평균 (결과가 있는 사람의 평균), 비-결과 평균 (결과가 없는 사람의 평균)이 나타나 있다.
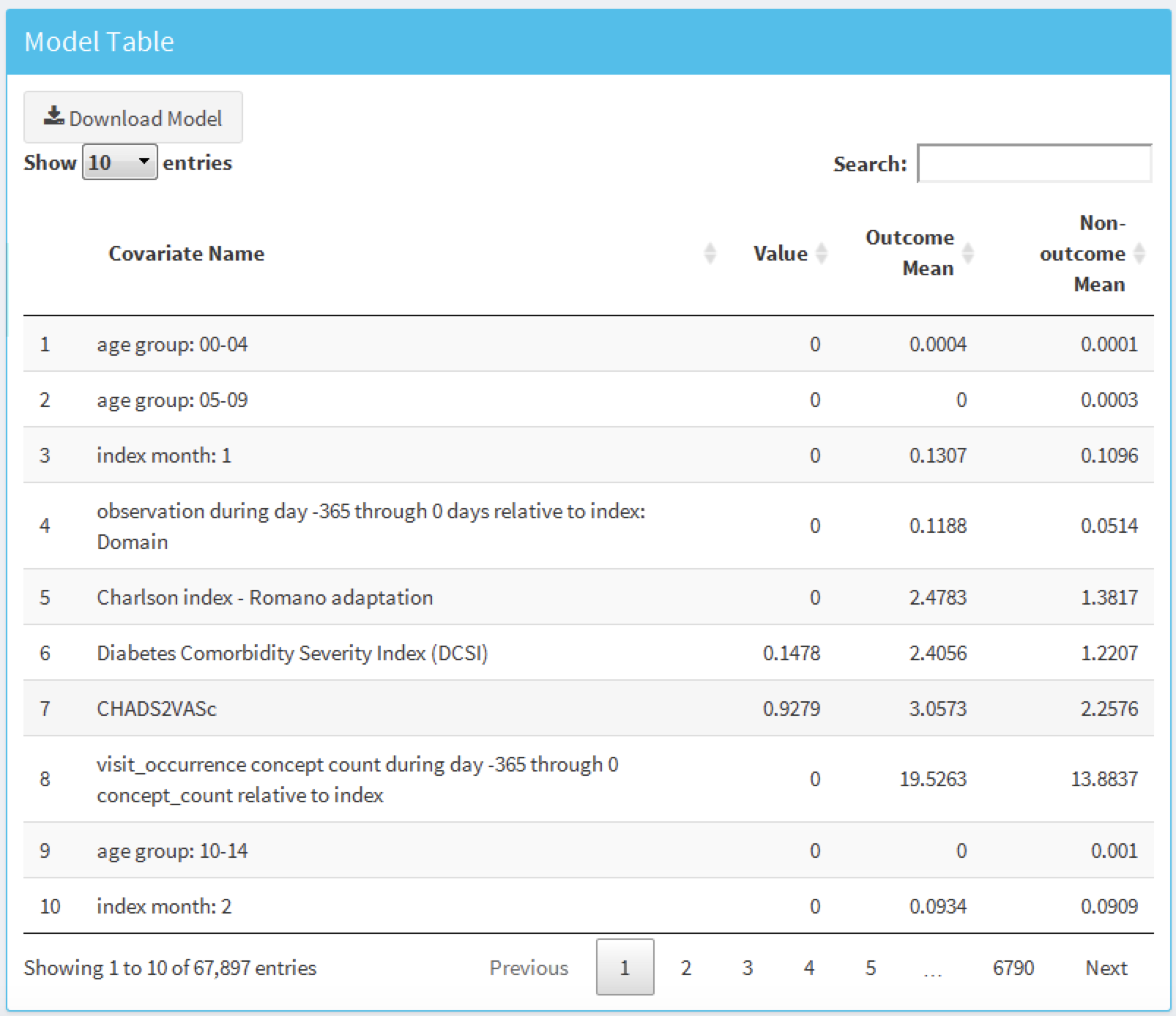
Figure 13.28: 모델 세부사항 표.
13.9 추가적 환자-수준 예측 변수
13.9.1 논문 제출용 문서 작성
논문지에 논문을 실을 수 있도록 워드 문서를 자동으로 생성하는 기능이 추가되었다. 그 문서에는 도출된 연구의 많은 세부사항과 결과가 포함되어 있다. 외적 타당도를 수행한 경우 그 결과도 추가 할 수 있다. 선택적으로, 대상 집단의 공변량이 포함된 표를 추가할 수 있다. 다음 기능을 사용하여 논문의 초안을 작성할 수 있다:
createPlpJournalDocument(plpResult = <your plp results>,
plpValidation = <your validation results>,
plpData = <your plp data>,
targetName = "<target population>",
outcomeName = "<outcome>",
table1 = F,
connectionDetails = NULL,
includeTrain = FALSE,
includeTest = TRUE,
includePredictionPicture = TRUE,
includeAttritionPlot = TRUE,
outputLocation = "<your location>")더욱 자세한 내용에 대해서는 기능의 도움 페이지를 참조하라.
13.10 요약
환자-수준 예측은 과거의 데이터를 사용하여 미래의 사건을 예측하는 모델을 개발하는 것을 목표로 한다.
모델 개발을 위한 최고의 기계학습 알고리즘 선택은 경험적인 문제이다. 즉 당면한 문제와 데이터에 의해 결정된다.
환자 수준 예측 PatientLevelPrediction 패키지는 OMOP-CDM의 데이터를 사용하여 예측 모델을 개발하고 검증하기 위한 모범 프로세스를 제공한다.
모델 및 그 성능 지표 보급은 대화식 interactive 대시보드로 수행된다.
- OHDSI의 예측 프레임워크는 인공지능의 임상 허가 전제 조건인 예측 모델의 대규모 외적 타당도 검증을 가능하게 한다.
13.11 예제
전제조건
이 내용을 연습하기 위하여 8.4.5절에 설명한 대로 R, R-Studio, Java가 설치돼야 한다. 또한 SqlRender, DatabaseConnector, Eunomia, 그리고 PatientLevelPrediction 패키지도 필요하며 다음을 설치하면 된다: SqlRender, DatabaseConnector, Eunomia and PatientLevelPrediction
install.packages(c("SqlRender", "DatabaseConnector", "devtools"))
devtools::install_github("ohdsi/Eunomia", ref = "v1.0.0")
devtools::install_github("ohdsi/PatientLevelPrediction")Eunomia 패키지는 자신의 PC에서 R을 실행할 수 있도록 모의 CDM 데이터를 제공한다. 세부 내용은 다음을 통해 확인 할 수 있다:
connectionDetails <- Eunomia::getEunomiaConnectionDetails()CDM 데이터베이스 스키마는 “main”이다. 이 예제는 여러 코호트를 사용한다. Eunomia 패키지의 createCohorts 함수는 코호트 테이블에서 다음과 같이 작성된다:
Eunomia::createCohorts(connectionDetails)문제 정의
NSAID를 처음 사용하기 시작한 환자에서 누가 내년에 위장관 출혈을 일으킬지 예측.
NSAID 신규 사용자 코호트는 COHORT_DEFINITION_ID = 4를 갖고, 위장관 출혈 코호트는 COHORT_DEFINITION_ID = 3을 갖는다.
Exercise 13.1 PatientLevelPrediction R 패키지를 사용하여 예측에 사용할 공변량을 정의하고, CDM에서 PLP 데이터를 추출하고, PLP 데이터를 요약하라.
Exercise 13.2 최종 대상 모집단을 정의하기 위하여 연구 선택사항을 다시 살펴보고 createStudyPopulation 함수를 사용하여 이를 지정하라. 선택한 것이 최종 모집단의 크기에 어떤 영향을 미칠 것인가?
Exercise 13.3 LASSO를 사용하여 예측 모델을 만들고 Shiny 앱을 사용하여 성능을 평가하라. 모델 성능은 어느 정도인가?
답안은 부록 E.9에서 찾을 수 있다.
References
Byrd, J. B., A. Adam, and N. J. Brown. 2006. “Angiotensin-converting enzyme inhibitor-associated angioedema.” Immunol Allergy Clin North Am 26 (4): 725–37.
Cicardi, M., L. C. Zingale, L. Bergamaschini, and A. Agostoni. 2004. “Angioedema associated with angiotensin-converting enzyme inhibitor use: outcome after switching to a different treatment.” Arch. Intern. Med. 164 (8): 910–13.
Norman, J. L., W. L. Holmes, W. A. Bell, and S. W. Finks. 2013. “Life-threatening ACE inhibitor-induced angioedema after eleven years on lisinopril.” J Pharm Pract 26 (4): 382–88.
O’Mara, N. B., and E. M. O’Mara. 1996. “Delayed onset of angioedema with angiotensin-converting enzyme inhibitors: case report and review of the literature.” Pharmacotherapy 16 (4): 675–79.
Reps, J. M., M. J. Schuemie, M. A. Suchard, P. B. Ryan, and P. R. Rijnbeek. 2018. “Design and implementation of a standardized framework to generate and evaluate patient-level prediction models using observational healthcare data.” Journal of the American Medical Informatics Association 25 (8): 969–75. doi:10.1093/jamia/ocy032.
Thompson, T., and M. A. Frable. 1993. “Drug-induced, life-threatening angioedema revisited.” Laryngoscope 103 (1 Pt 1): 10–12.